|
Molecola del Mese di settembre
2010
L'enzima isocitrato deidrogenasi realizza la terza tappa del ciclo di
Krebs, l'ossidazione e la decarbossilazione dell'acido isocitrico
 Introduzione Introduzione
Lo zucchero ha un buon sapore ed è ovvio
perché il glucosio è la principale fonte di energia per
tutti gli organismi aerobi. Lo zucchero viene demolito nella via metabolica
principale chiamata respirazione cellulare che comprende glicolisi
(mdm 2/2004) , decarbossilazione
ossidativa (mdm 9/2012) e ciclo
di Krebs (mdm10-2012) e alla fine porta alla sintesi di ATP.
Gli enzimi di questo percorso biochimico spezzano la catena di carboni
del glucosio producendo CO2
e catturano l'energia liberata in ogni passaggio. L'enzima isocitrato
deidrogenasi realizza la terza reazione del ciclo di Krebs nella
quale rilascia una molecola di CO2
in seguito all'ossidazione e alla decarbossilazione dell'acido
isocitrico. Nella reazione vengono rimossi due idrogeni, il primo è
uno ione idruro H-
che, durante l'ossidazione, viene trasferito al NAD+
e lo trasforma in NADH che servirà poi per azionare, attraverso
la catena
respiratoria (mdm 12/2011), la produzione di ATP da parte dell'enzima
ATP
sintasi (mdm 12/2005). L'altro idrogeno viene rimosso come H+
durante la decarbossilazione.
Approcci diversi per lo stesso compito
Nelle nostre cellule, così come in quelle
di tutti gli eucarioti, queste reazioni sono realizzate da un
enzima complesso, composto di otto catene proteiche.
Qui a lato in alto è mostrato l'enzima di lievito (file PDB 3blw).
L'enzima è composto di quattro catene catalitiche (azzurre) che
realizzano le reazioni, e quattro catene regolatorie (blu) che accendono
o spengono l'enzima a seconda dei livelli di ADP e ATP nella cellula.
I batteri (procarioti) usano una diversa strategia. Utilizzano
un enzima più semplice composto di due sole catene proteiche
identiche che contengono due siti attivi uguali, come è illustrato
qui sopra in basso (file PDB 9icd).
Anche le nostre cellule possiedono una versione piccola dell'enzima
isocitrato deidrogenasi, ma lo usano nel citoplasma per interconvertire
isocitrato e alfa-chetoglutarato quando servono per altri scopi sintetici
(il ciclo di Krebs, invece, è localizzato nei mitocondri).
 Controllo
con la fosforilazione Controllo
con la fosforilazione
L'enzima batterico isocitrato deidrogenasi
non è controllato dai livelli di ADP e ATP come il nostro enzima
che lavora nei mitocondri. Dato però che anche i batteri devono
poter spegnere l'enzima quando c'è abbastanza ATP, regolano l'enzima
isocitrato deidrogenasi aggiungendo un fosfato alla catena proteica
per inattivarlo. Per questa fosforilazione usano l'enzima isocitrato
deidrogenasi chinasi/fosfatasi che è mostrato qui a lato
in arancione (file PDB 3lcb) mentre è
legato all'isocitrato deidrogenasi (azzurro). Questo enzima è
in grado di realizzare entrambe le reazioni: aggiunge fosfato
per spegnere l'enzima e lo rimuove per attivarlo. Questo enzima
regolatore decide quale reazione è meglio effettuare a seconda
dei livelli di AMP nella cellula: quando i livelli sono alti, AMP si
lega ad un sito di regolazione attivando la reazione di rimozione del
fosfato, questo porta all'attivazione del ciclo di Krebs e quindi alla
sintesi di ATP. Bassi livelli di AMP, invece, lo fanno diventare una
chinasi cioè un enzima che aggiunge fosfato.
Esplorando la struttura
I cristallografi hanno esplorato molti degli stadi della reazione condotta
dall'enzima isocitrato deidrogenasi. Le prime strutture ottenute mostravano
i complessi che l'enzima realizza con i suoi vari substrati e con i
prodotti di reazione. Negli archivi PDB ci sono quindi strutture dell'enzima
con isocitrato e magnesio (file PDB 8icd),
NADP (file PDB 9icd), alfa-chetoglutarato
(file PDB 1ika), e inoltre l'apoenzima
(enzima nudo, file PDB 3icd), e l'enzima
fosforilato inattivo (file PDB 4icd).
Per studiare i dettagli della reazione, però, è stato
necessario ricorrere a speciali tecniche sperimentali.
Sincronizzando con precisione l'addizione di substrato a forme mutate
dell'enzima e poi utilizzando la tecnica di diffrazione Laue per ottenere
dati cristallografici in millisecondi, i ricercatori sono stati in grado
di osservare gli intermedi instabili della reazione.
Qui sotto è mostrato l'enzima mutante Y160F (file PDB 1ide)
che rallenta molto il primo stadio della reazione, rivelando
così la struttura del complesso appena formato tra l'enzima,
l'isocitrato, il NADP e il magnesio, prima che la reazione di ossidazione
abbia inizio.
Nell'isocitrato (piccole sfere azzurre) il carbonio che regge l'OH (con
asterisco) è circondato in basso dallo ione magnesio (verde)
e in alto dalla porzione ossidante del NADP+
(con asterisco). Questo è il momento
in cui l'socitrato cede lo ione H-
al NADP+. La reazione
è rallentata perchè una tirosina critica nel sito attivo
(Tyr 160) è stata sostituita con una fenilalanina (rosa).
. . . . .
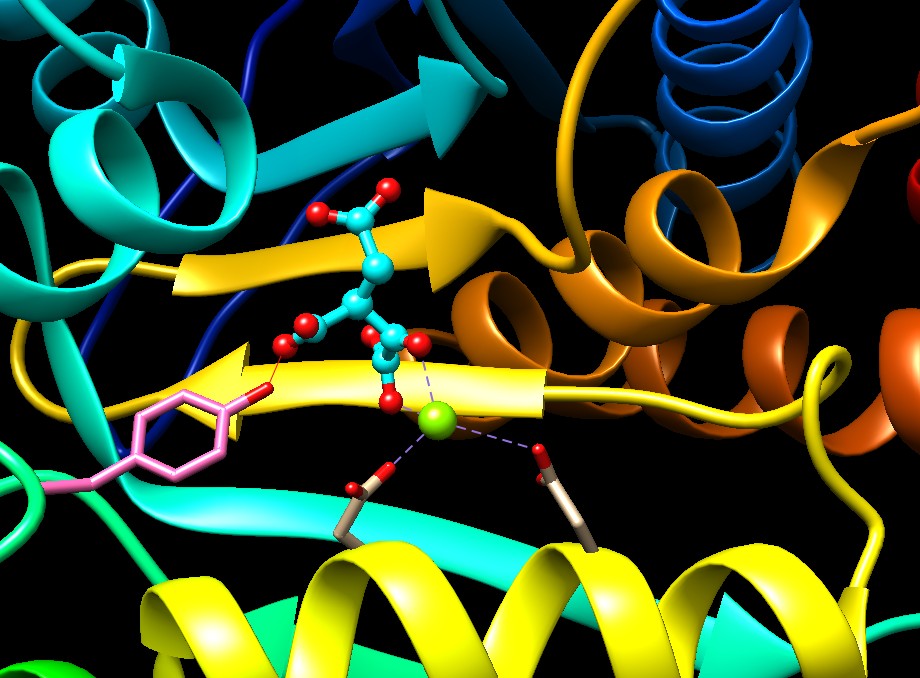
Qui sotto è mostrato l'enzima mutante K230M (file PDB 1idc)
che, invece, rallenta il secondo stadio della reazione, rivelando
così la struttura dell'enzima legato all'intermedio ossalacetato
prima che avvenga la decarbossilazione e cioè la perdita
di CO2. Si osservi
che l'isocitrato ossidato è diventato acido ossalosuccinico
(piccole sfere azzurre) . E' circondato oltre che dal magnesio
(verde) che si lega all'ossigeno del carbonile (rosso al centro), anche
da una tirosina (Tyr 160, rosa) che con il suo ossigeno (rosso)
sta per strappare l'H+
al carbossile centrale dell'acido ossalosuccinico . Questo, poi, si
staccherà come CO2
formando alfa-chetoglutarato, il prodotto finale.
. . . . .
Spunti per ulteriori esplorazioni
Negli archivi PDB sono disponibili le strutture
di molti degli otto enzimi del ciclo di Krebs. Provate a trovarle.
L'enzima isocitrato deidrogenasi sa distinguere tra i due stereoisomeri
dell'isocitrato. Riesce a farlo perchè circonda l'isocitrato
e forma specifiche interazioni con ognuno dei suoi gruppi funzionali.
Provate ad identificare gli amminoacidi nella catena proteica dell'enzima
che sono importanti per queste interazioni. Cercate anche di capire
il ruolo dello ione magnesio.
 Bibliografia Bibliografia
J. Zheng, Z. Jia (2010) Structure of the
bifunctional isocitrate dehydrogenase kinase/phosphatase. Nature
465, 961-965.
A. B. Taylor, G. Hu, P. J. Hart and L. McAlister-Henn (2008)
Allosteric motions in structures of yeast NAD+-specific
isocitrate dehydrogenase. Journal of Biological Chemistry 283,
10872-10880.
J. M. Bolduc, D. H. Dyer, W. G. Scott, P. Singer, R. M. Sweet,
D. E. Koshland Jr. and B. L. Stoddard (1995) Mutagenesis and Laue structure
of enzyme intermediates: isocitrate dehydrogenase. Science 268,
1312-1318.
J. H. Hurley, A. M. Dean, D. E. Koshland Jr. and R. M. Stroud
(1991) Catalytic mechanism of NADP+-dependent
isocitrate dehydrogenase: implications from the structures of magnesium-isocitrate
and NADP+ complexes.
Biochemistry 30, 8671-8678.
|
|
|

 Introduzione
Introduzione Controllo
con la fosforilazione
Controllo
con la fosforilazione
