|
|
Fattori di allungamento |
|||||
|
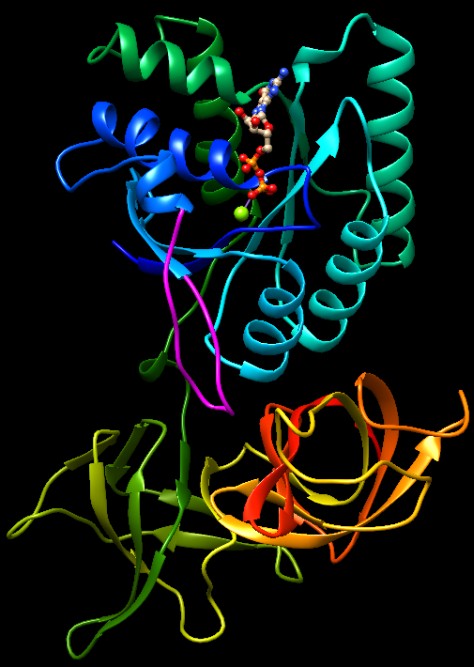
Molecola del Mese di Settembre 2006 La sintesi di proteine richiede l'assistenza di molti fattori di allungamento per guidare ogni passaggio 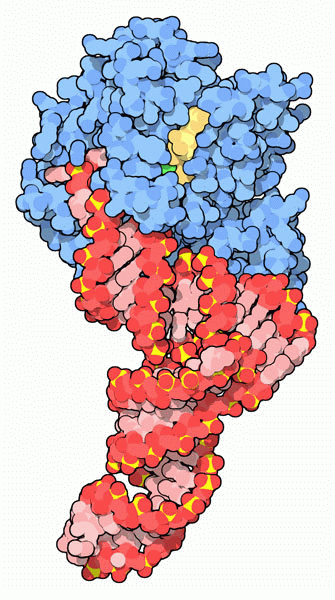 Introduzione IntroduzionePiù di metà degli strumenti molecolari in una tipica cellula batterica è dedicata alla sintesi proteica. Questi strumenti comprendono il DNA (mdm 11-2001) e l'RNA messaggero (mdm 1-2012) che forniscono l'informazione, l'RNA transfer (mdm 3-2001) che traduce l'informazione di sequenze di nucleotidi nei corrispondenti amminoacidi e i ribosomi (mdm 1-2010) che svolgono il vero lavoro di assemblaggio di amminoacidi per formare la proteina. La sintesi delle proteine richiede anche una serie di fattori proteici per regolare i vari passaggi. Questi includono fattori di inizio (mdm 2-2019) che fanno cominciare il processo, fattori di rilascio che interrompono la sintesi quando si raggiunge la tripletta di stop e fanno sganciare la proteina finita e fattori di allungamento che assistono i molti passaggi tra l'inizio e la fine della sintesi. I fantastici tre Tutti gli organismi viventi producono tre tipi di fattori di allungamento che hanno più o meno la stessa forma. Questi sono chiamati EF-Tu, EF-Ts e EF-G (EF = Elongation Factor). Come accade spesso, comunque, esistono anche altri nomi per ognuno di questi, assegnati nel corso degli anni dai ricercatori. Così, per gli eucarioti, questi tre fattori possono essere anche chiamati EF-1A, EF-1B e EF-2, talvolta preceduti da una "e" (eucaryotic) come in eEF-1A. Quindi non stupitevi se troverete nomi diversi in letteratura. Trasporto speciale Il fattore EF-Tu, mostrato qui a destra dal file PDB 1ttt, svolge l'importante funzione di guidare ogni RNA transfer fino al ribosoma, sfruttando l'energia di una molecola di GTP. Il fattore EF-Tu è la proteina più abbondante nelle cellule batteriche, infatti ci deve essere almeno una proteina EF-Tu per ogni tRNA. Il fattore EF-Tu (azzurro) si lega al tRNA (rosso) dopo che questi si è legato al proprio amminoacido. Successivamente il complesso EF-Tu/tRNA si inserisce nel sito attivo del ribosoma. Quando l'anticodone del tRNA si accoppia correttamente con il codone dell'mRNA, un segnale del ribosoma fa cambiare forma ad EF-Tu e viene scissa la molecola di GTP (gialla) che contiene. Questo fa staccare EF-Tu dal tRNA e consente al tRNA di eseguire la reazione di aggancio del proprio amminoacido alla catena proteica in crescita. Riattivazione Dopo che EF-Tu ha completato il suo lavoro, deve essere riattivato allontanando il GDP dal sito attivo e introducendo al suo posto un nuovo GTP. Questo compito viene svolto dal fattore EF-Ts che si lega a EF-Tu che contiene GDP e ne provoca una deformazione che destabilizza il legame con il GDP. La struttura qui sotto (file PDB 4pc2) mostra il complesso EF-Tu/EF-Ts del batterio Escherichia coli. Notate come il fattore EF-Ts (magenta) si stringe intorno ad EF-Tu (arcobaleno) proprio nel punto dove questo lega GDP (sfere colorate). . . . . . . . 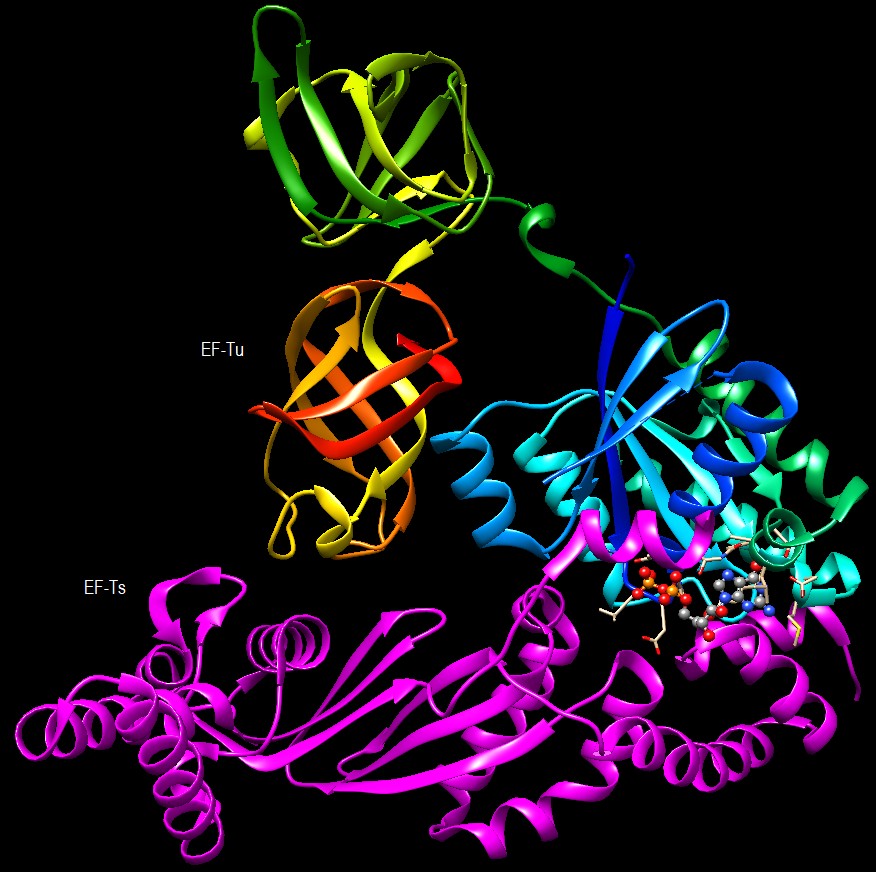 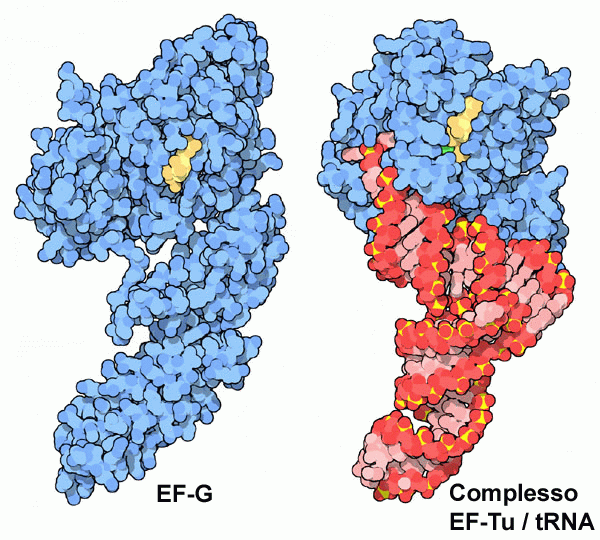 Avanzare AvanzareDopo che il ribosoma ha legato il nuovo amminoacido alla catena proteica in crescita, deve spostarsi lungo l'mRNA ed espellere la molecola di tRNA non più legata alla catena proteica, creando spazio perchè il successivo tRNA possa arrivare a legarsi al nuovo codone. Questo avviene con l'assistenza dal fattore EF-G. Come si può vedere nella figura qui a lato, il fattore EF-G (file PDB 1dar) ha una forma molto simile a quella del complesso EF-Tu/tRNA (file PDB 1ttt). Sfruttando questa somiglianza, il fattore EF-G si lega al ribosoma e lo sposta lungo l'mRNA in avanti di un codone, sfruttando l'energia dell'idrolisi di una molecola di GTP (gialla). Questo passaggio è ben descritto nella molecola del mese ribosoma 70S (1-2010). Come si può vedere nel prossimo paragrafo, questa idrolisi è accompagnata da un grande cambiamento di forma. del fattore EF-G. Esplorando la struttura Come accade quasi sempre con le proteine che utilizzano GTP o ATP, anche i fattori di allungamento vanno incontro a grandi cambiamenti di forma nel corso della loro azione. Confrontando le due molecole mostrate qui sotto, si può vedere la variazione di struttura provocata dall'idrolisi del GTP nel fattore EF-Tu. Sulla sinistra il fattore EF-Tu lega il GTP (sferette colorate) o meglio un suo analogo meno idrolizzabile, il GDPNP che ha un atomo di azoto (blu) che lega l'ultimo fosfato in basso (file PDB 1eft). Sulla destra il fattore EF-Tu lega GDP che ha solo due gruppi fosfato (arancioni e rossi) (file PDB 1tui). Nelle molecole è presente anche uno ione magnesio (verde). Notate la grande variazione di struttura del tratto di catena magenta che a sinistra lega l'ultimo fosfato, mentre a destra, dato che questo non c'è più, si è allungata in basso generando un ulteriore riarrangiamento soprattutto nel tratto verde e nell'elica ciano. 
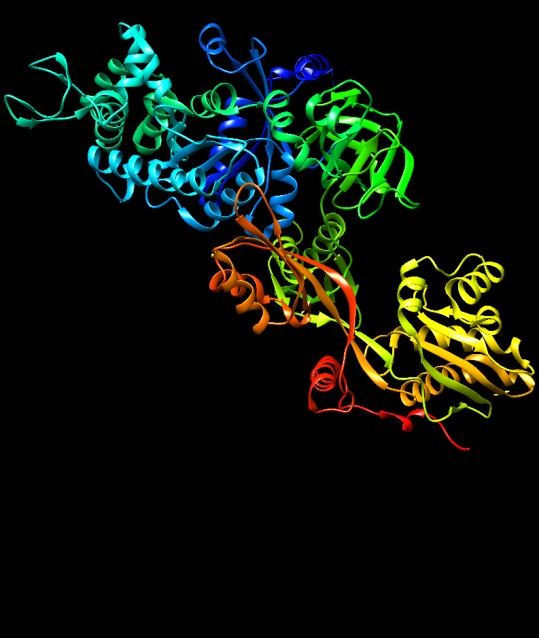
 Qui sotto sono mostrati due fattori EF-2, una variante di EF-G presente nel lievito: la variazione di forma tra le due strutture è molto evidente . Nella molecola sulla sinistra (file PDB 1n0u) è legata la sordarina (file PDB sordarina), un farmaco antifungino. Nella molecola sulla destra (file PDB 1n0v), invece, non vi è nessuna molecola legata. Notate quanto grande è la piegatura che subisce il dominio in basso passando da una struttura all'altra. Variazioni così grandi di struttura si sono osservate anche nei ribosomi. Queste immagini sono state ottenute con Chimera (vedi chimica al computer). 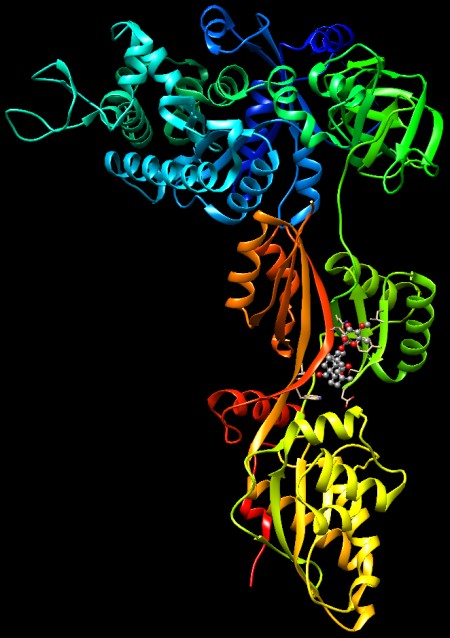 .
.
G. R. Andersen, P. Nissen and J. Nyborg (2003) Elongation factors in protein biosynthesis. Trends in Biochemical Sciences 28, 434-441. I. M. Krab, A. Parmeggiani (2002) Mechanisms of eEF-Tu, a pioneer GTPase. Progress in Nucleic Acids Research 71, 513-551.
|
||||||
|
|
||||||
