|
Molecola del Mese di gennaio 2010
I ribosomi sono complesse macchine molecolare che
realizzano la sintesi delle proteine nella cellula
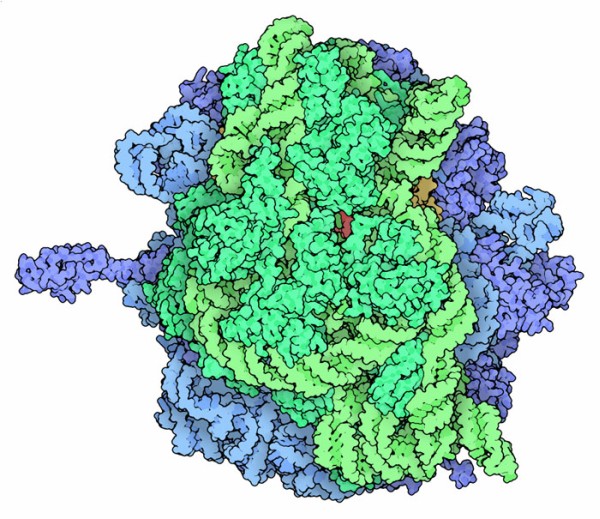 Introduzione Introduzione
I ribosomi sono una delle meraviglie del mondo cellulare
che potete esplorare visitando gli archivi PDB. Nell'anno 2000, i biologi
strutturali V. Ramakrishnan, T. Steitz e A. Yonath hanno reso disponinili
nel PDB le prime strutture di subunità ribosomiali. Nel 2009 hanno
vinto il premio Nobel per quel lavoro. Oggi sono disponibili le strutture
di molte altre molecole coinvolte nella sintesi proteica, compresi gli
RNA-transfer
(mdm 3-2001) e i fattori
di allungamento (mdm 9-2006). A partire da queste strutture sono
state ricostruite centinaia di strutture di ribosomi interi che rivelano
i dettagli atomici di molti dei passaggi della sintesi proteica.
Ribosomi in azione
Dopo avere risolto le strutture delle due subunità,
piccola e grande, dei ribosomi, il passo successivo nella ricerca è
stato quello di determinare la struttura del ribosoma intero. Questo ha
rappresentato il completamento di una ricerca durata decine di anni e
cominciata con le prime immagini sfocate di ribosoma ottenute al microscopio
elettronico, continuata con ricostruzioni più dettagliate di
micrografia crioelettronica, e ora arrivata con la diffrazione
ai raggi X a strutture che mostrano la posizione dei singoli atomi.
Utilizzando piccoli frammenti di mRNA, varie forme di tRNA accorciato
o chimicamente modificato, fattori proteici purificati e ribosomi modificati,
i ricercatori hanno determinato la struttura dei ribosomi nell'atto di
sintetizzare le proteine. Queste strutture sono così grandi che
non possono essere contenute in un singolo file PDB. Per esempio, la struttura
mostrata qui sopra è stata divisa in due file PDB 2wdk
e 2wdl.
Ribosomi 70S
Osservando le forme di vita sulla Terra, troviamo
che tutti gli organismi viventi hanno ribosomi e che questi sono sostanzialmente
di due misure. I batteri e gli archeobatteri hanno ribosomi più
piccoli chiamati ribosomi 70S, che sono composti di una subunità
minore 30S e di una maggiore 50S. La S sta per Svedbergs o fattore di
Sedimentazione che misura quanto velocemente la molecola si muove in
una centrifuga. Notate che questi valori non sono additivi, cioè
30 più 50 non fa 70, infatti la velocità di sedimentazione
è proporzionale alla massa e alla forma della molecola, ma non
in modo lineare.
I ribosomi delle nostre cellule, come quelli degli altri animali, delle
piante e dei funghi, sono più grandi e vengono detti ribosomi
80S, sono composti di una subunità minore 40S e di una subunità
maggiore 60S. Stranamente i nostri mitocondri hanno ribosomi piccoli
70S che vengono sintetizzati in loco e sono diversi da quelli più
grandi presenti nel citoplasma. Questa osservazione ha suggerito l'ipotesi
che i mitocondri (e così pure i cloroplasti nelle cellule vegetali)
siano in realtà dei batteri che sono stati catturati all'interno
di cellule più evolute all'inizio dell'evoluzione delle cellule
eucariote. Oggi vivono e si riproducono felicemente all'interno delle
nostre cellule dove si occupano della produzione di energia e si affidano
alla cellula ospite per la maggior parte delle loro necessità.
Ribozimi
Le prime strutture di ribosoma ottenute molti anni
addietro, anche se poco dettagliate, hanno rivelato le caratteristiche
fondamentali dei ribosomi. Hanno dimostrato che sono ribozimi,
cioè enzimi fatti di RNA, infatti usano RNA e non proteine per
catalizzare la reazione di sintesi delle proteine. Questo ha avvalorato
l'idea che l'RNA è stato fondamentale nella prima parte dell'evoluzione
della vita perchè può svolgere contemporaneamente sia
il ruolo di enzima (oggi svolto dalle proteine) sia quello di
trasmettere il patrimonio genetico (oggi svolto dal DNA). Le
prime strutture hanno anche mostrato l'importanza delle proteine ribosomiali
per stabilizzare e tenere in forma la struttura dell'RNA nel ribosoma.
Le strutture ottenute recentemente, però, sono molto più
dettagliate e ci hanno permesso di investigare a livello atomico il
meccanismo della lettura dell'informazione genetica e della sintesi
delle proteine.
La biosintesi delle proteine si svolge in tre tappe fondamentali: inizio,
allungamento, terminazione. Negli archivi PDB sono disponibili
strutture che illustrano ognuna di queste tre fasi.
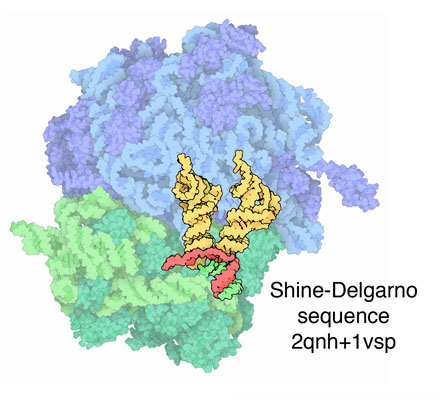 Inizio Inizio
Il ribosoma comincia ad agire in un processo chiamato
inizio. Alcune proteine, chiamate fattori di inizio, trasportano
l'mRNA alla subunità minore, allineano la prima
parte dell'mRNA e promuovono l'associazione con la subunità
maggiore. La struttura illustrata qui a fianco (file PDB
2qnh e 1vsp) mostra una particolare
sequenza nell'mRNA, chiamata sequenza Shine-Delgarno dai suoi scopritori,
che è associata con l'ultima parte della catena di RNA della
subunità minore. Questo fa allineare l'mRNA nel punto giusto
e lo predispone a legarsi con uno speciale tRNA iniziatore. In questa
figura si vede un piccolo tratto di mRNA (in rosso) e due tRNA (in giallo).
Una nota su questa figura e le successive:
l'mRNA, il tRNA e i fattori proteici si legano tutti all'interno del
ribosoma, tra le due subunità, quindi non è facile creare
un'immagine che mostri quello che accade. Queste immagini mostrano l'mRNA,
il tRNA e altre molecole inserite in una struttura semplificata
(e sfumata) del ribosoma per mostrare in modo più chiaro dove
sono inserite nel complesso intero.
Allungamento
Dopo che il ribosoma è stato assemblato,
comincia a scorrere verso sinistra lungo la catena di mRNA e a costruire
la proteina, un amminoacido alla volta. Nella prima struttura mostrata
qui sotto a sinistra (file PDB 2wrn e
2wro), un nuovo tRNA viene trasportato
da un fattore di allungamento, la proteina EF-Tu (magenta).
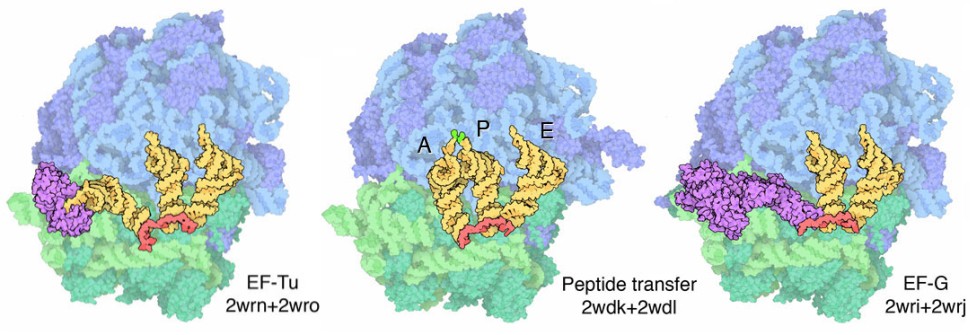
Nella seconsa struttura al centro (file PDB 2wdk
e 2wdl), tre molecole di tRNA sono legate
all'interno del ribosoma. Il tRNA nel sito A (amminoacido) trasporta l'amminoacido
che deve essere aggiunto (verde), il tRNA nel sito P (proteina) lega la
catena proteica in crescita (verde), mentre il tRNA nel sito E (exit)
ha finito il suo compito ed è pronto per essere espulso. La catena
proteica viene trasferita dal tRNA del sito P all'amminoacido (verde)
legato al tRNA del sito A.
Nella terza struttura sulla destra, la proteina
EF-G ha aiutato a spingere verso sinistra il ribosoma rispetto
all'mRNA liberando così il sito A (file PDB 2wri
e 2wrj), in questo modo il processo può
ricominciare come nella prima figura con l'arrivo nel sito A di un nuovo
tRNA trasportato dal fattore di allungamento EF-Tu.
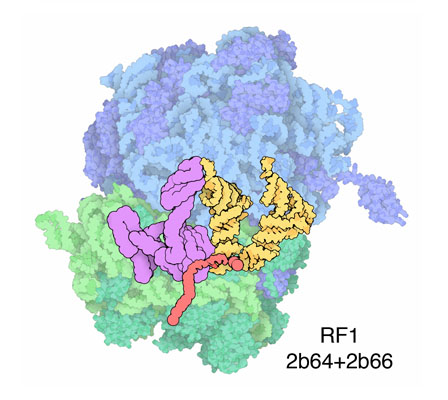 Terminazione Terminazione
Alla fine del gene, cioè alla fine del tratto
di mRNA che codifica per la proteina, il ribosoma incontra un codone
di stop (per es la tripletta di basi UGA) che gli dice di smettere
di costruire la proteina. Speciali proteine chiamate fattori di rilascio
riconoscono il codone di stop e costringono il tRNA nel sito P a rilasciare
la proteina finita. Questa struttura (file PDB 2b64
e 2b66) mostra uno dei fattori di rilascio
(magenta) legato al codone stop dell'mRNA nel sito A del ribosoma. Questa
struttura è stata ottenuta a bassa risoluzione, quindi mostra in
modo un po' approssimato il legame con la proteina fattore di rilascio
e così pure non contiene le parti terminali dell'mRNA che sono
state aggiunte con un programma di modellistica molecolare.
Antibiotici
Dato che i ribosomi sono essenziali per la vita, sono
un bersaglio ideale per i farmaci antibiotici. Naturalmente è indispensabile
che l'antibiotico attacchi solo i ribosomi dei batteri senza colpire i
nostri, altrimenti combatteremmo anche noi stessi insieme con l'infezione.
Per fortuna, i ribosomi dei batteri hanno molte piccole differenze rispetto
ai nostri e quindi ci sono vari antibiotici che attaccano in modo specifico
i ribosomi 70S.
Qui sotto ne sono mostrati due esempi, ma ce ne sono molti altri negli
archivi PDB. Sulla sinistra è illustrata una tetraciclina
(rossa) legata alla subunità minore del ribosoma (file PDB 1hnw),
dove impedisce il legame con l'mRNA. Sulla destra è mostrato il
cloramfenicolo (rosso, appena visibile) legato in un punto del
ribosoma (file PDB 1nji) che impedisce la
reazione che aggiunge nuovi amminoacidi alla catena proteica in crescita.

Esplorando la struttura
Le nuove strutture di ribosomi interi 70S rivelano
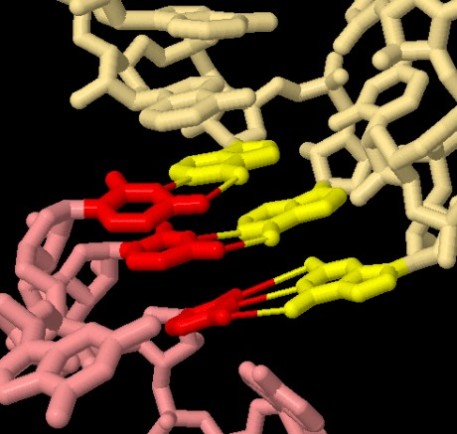
il segreto della vita. Le figure qui sotto mostrano in primo piano il
centro di decodifica del ribosoma (file PDB 2wdg).
Questo è il luogo dove l'anticodone del tRNA in arrivo
(giallo) si accoppia con il codone dell'mRNA (rosso).
Come si può immaginare, è essenziale che questo accoppiamento
sia perfetto per cui solo il giusto tRNA si può legare al suo
codone e così solo il corretto amminoacido può essere
aggiunto alla catena della proteina in crescita. Il ribosoma utilizza
molte interazioni per controllare che l'accoppiamento tra le basi azotate
di codone e anticodone sia corretto.
 . . .
. . . 
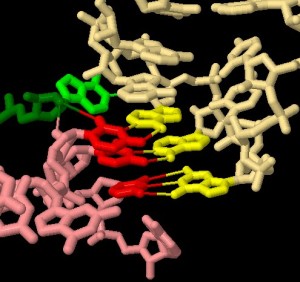
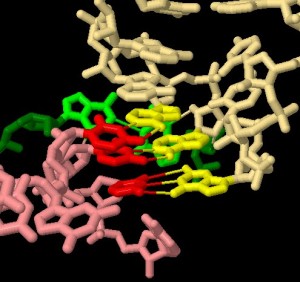
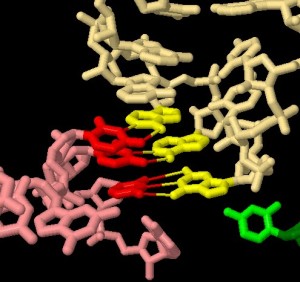
Nelle tre immagini qui sotto, si può vedere in dettaglio
l'accoppiamento codone-anticodone rispetto alle tre paia di basi coinvolte.
Alcune porzioni del ribosoma (verde) interagiscono col tRNA (giallo)
e con l'mRNA (rosso) in corrispondenza delle tre paia di basi di codone
e anticodone. Nelle tre immagini sono mostrate solo le parti del ribosoma
che interagiscono rispettivamente con la prima, la seconda e la terza
coppia di basi.
Nelle prime due immagini si vede che le interazioni che controllano
l'accoppiamento delle prime due paia di basi sono numerose e
assicurano così che solo le basi che si accoppiano alla perfezione
possano legarsi.
Nell'ultima immagine, invece, si vede che la terza coppia di basi
è controllata solo da una debole interazione che consente molta
libertà, infatti spesso in questa terza posizione si possono
trovare basi diverse non perfettamente accoppiate. E' per questo che
talvolta uno stesso amminoacido può essere codificato da più
triplette che sono uguali nelle prime due posizioni, ma che
differiscono nella terza. Per esempio la serina viene codificata da
quattro diversi codoni: UCU, UCC, UCA, UCG.
 . .  .
. 
Spunti per ulteriori esplorazioni
1) Il ribosoma è formato da due subunità
che si assemblano attorno all'mRNA per formare un complesso funzionale.
Quali sono i vantaggi di questa strategia? Trovate altri esempi di molecole
che sono in grado di legarsi attorno alla catena di RNA o DNA.
2) I ribosomi sono molecole molto difficili da studiare. Esplorando
le strutture di ribosomi negli archivi PDB, confrontate i divesi tipi
di dati che sono stati utilizzati per determinare le strutture, incluse
le strutture cristallografiche a risoluzione atomica o quasi atomica
e le ricostruzioni fatte da immagini al microscopio elettronico a più
bassa risoluzione.
 Bibliografia Bibliografia
A. Korostelev, H. F. Noler (2007) The ribosome
in focus: new structures bring new insights. Trends in Biochemical
Sciences 32, 434-441.
T. A. Steitz (2008) A structural understanding of the dynamic
ribosome machine. Nature Reviews Molecular Cell Biology 9,
242-253.
T. M. Schmeing, V. Ramakrishnan (2009) What recent ribosome structures
have revealed about the mechanism of translation. Nature 461,
1234-1242.
E. Zimmerman, A. Yonath (2009) Biological implications of the
ribosome's stunning stereochemistry. ChemBioChem 10, 63-72.
|
|
|
 Introduzione
Introduzione
 Terminazione
Terminazione

 Inizio
Inizio

 .
.  .
. 