|
Molecola del Mese di Marzo 2016
Una singola mutazione in una proteina RAF può
trasformare una cellula normale in cancerosa
 Introduzione Introduzione
Talvolta la modifica di un singolo amminoacido in
una proteina può avere conseguenze drammatiche. Queste modifiche
o mutazioni possono essere causate anche dalla variazione di un solo
nucleotide di un gene che viene chiamata variante a singolo nucleotide
o SNV.
Le SNV cambiano il significato di una tripletta del gene e quindi cambiano
l'amminoacido codificato in quella posizione.
Per esempio il cambiamento nel gene di una Guanina con una Timina
trasforma la tripletta GGC in GTC.
La prima codifica per Glicina, mentre la seconda codifica per
Valina.
In questo modo vengono sintetizzate proteine mutanti.
Un esempio perfetto di questo tipo si trova nel gene che codifica per
la proteina di segnalazione BRAF: un cambiamento di un singolo nucleotide
può trasformare una cellula normale in una cellula di melanoma.
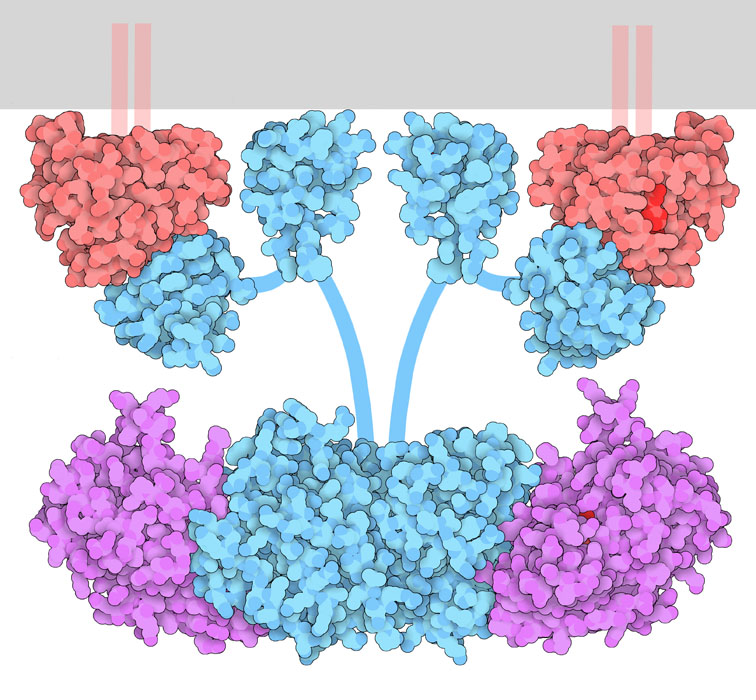
Mandare il segnale
La proteina RAF (in azzurro nella figura qui sopra)
ha un ruolo importante nel controllare la crescita cellulare. Il processo
di segnalazione comincia quando viene attivato un recettore, come quello
dell'ormone
della crescita (mdm 4-2004, non mostrato qui sopra). Il segnale
da questo è trasmesso ad una serie di proteine che operano in
cascata all'interno della cellula: da RAS
(mdm 4-2012, rossa), a RAF (azzurra), a MEK (magenta)
e infine a ERK che si muove nella cellula per innescare i meccanismi
cellulari di crescita. Molte di queste proteine sono chinasi che aggiungono
gruppi fosfato alla superficie della successiva proteina della catena,
per attivarla.
Da RAS a RAF
La proteina RAF (azzurra qui sopra), è una
molecola complessa con molti domini che svolgono diverse funzioni. Finora
i biochimici strutturali hanno studiato molti di questi domini. Il dominio
di RAF che lega la proteina RAS (rossa, file PDB 3kud)
si lega a questa solo se è attivata cioè se è legata
a GTP. Dato che molte copie di RAS sono legate alla superficie interna
della membrana cellulare, costringono le proteine RAF legate ad avvicinarsi
tra loro, favorendone la dimerizzazione, questo è il segnale
di attivazione per RAF.
Un altro dominio di RAF, chiamato dominio ricco di cisteine
(azzurro in alto al centro nella figura in alto, file PDB 1far),
aiuta la dimerizzazione interagendo con la superficie interna della
membrana cellulare.
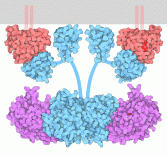
Da RAF a MEK
Il dimero di RAF è attivato, quindi a sua
volta attiva MEK (magenta) fosforilandola. Questo processo è
stato catturato dalla struttura PDB 4mne,
ed è mostrato nella figura qui sopra in basso dove si vede il
dimero di RAF al centro con due copie di MEK legate ai lati.
Alcuni filamenti flessibili (schematizzati con segmenti azzurri) legano
tra loro le varie porzioni di RAF.
Nelle nostre cellule si trovano molte forme di RAF chiamate ARAF, BRAF,
CRAF che lavorano insieme formando spesso dei dimeri misti nei
quali si legano insieme due forme diverse, in questo modo possiamo controllare
i molteplici aspetti della crescita cellulare.
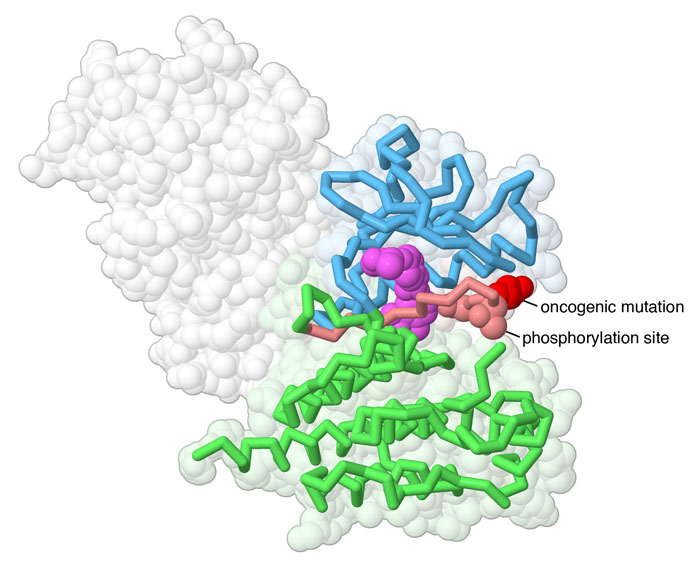 Mutazione
oncogenica Mutazione
oncogenica
Un piccolo segmento di BRAF, mostrato in rosa nella
figura qui a fianco (file PDB 1uwh), è
chiamato segmento di attivazione perchè è il punto
chiave per la segnalazione. Questo tratto è normalmente fosforilato
in più punti quando la proteina è attivata e la costringe
a mantenere l'assetto tridimensionale attivo. Un particolare amminoacido
in questo segmento (nella posizione 600) è il sito della mutazione
oncogenica. Quando in questo punto al posto di una valina
si trova acido glutammico (chiamata mutazione V600E: V
= valina, E = acido glutammico), la parte restante del segmento si sposta
e rende l'enzima sempre attivo. Questo cambiamento strutturale consente
alle cellule cancerose di crescere eludendo i normali controlli. Questa
mutazione è la conseguenza di una SNV (mutazione a singolo nucleotide)
nel gene che codifica RAF. Questa mutazione si trova solo nelle cellule
maligne, mentre è assente nei tessuti normali dello stesso individuo.
Negli Stati Uniti la FDA (Food and Drug Administration) ha approvato
il farmaco Vermurafenib che era stato progettato in modo specifico
per combattere la forma mutante V600E della proteina RAF che si trova
nella metà dei melanomi maligni, una forma mortale di cancro
della pelle. L'uso del Vermurafenib è limitato ai soli pazienti
che hanno un melanoma con la mutazione V600E che può essere
individuata da un test diagnostico sviluppato insieme al farmaco.
Esplorando la struttura
La conoscenza della struttura mutata di RAF ha permesso
ai ricercatori di progettare un farmaco in grado di bloccarne l'azione
frenando così la crescita innaturale delle cellule cancerose.
Questi farmaci sono efficaci su alcuni tipi di tumore, come il melanoma,
ma possono risultare controproducenti in altri tipi di tumore accelerandone
la crescita. Quando i ricercatori hanno studiato più a fondo
questo aspetto, hanno scoperto che la crescita inattesa poteva essere
causata da una interazione insolita: talvolta BRAF e CRAF formano
eterodimeri, ma il farmaco si lega solo a BRAF. Questo problema
è stato superato usando dosi più alte del farmaco per
bloccare tutte e due le forme, ma ora stanno lavorando su un farmaco
di seconda generazione capace di impedire a RAF di formare dimeri
e quindi di attivarsi.
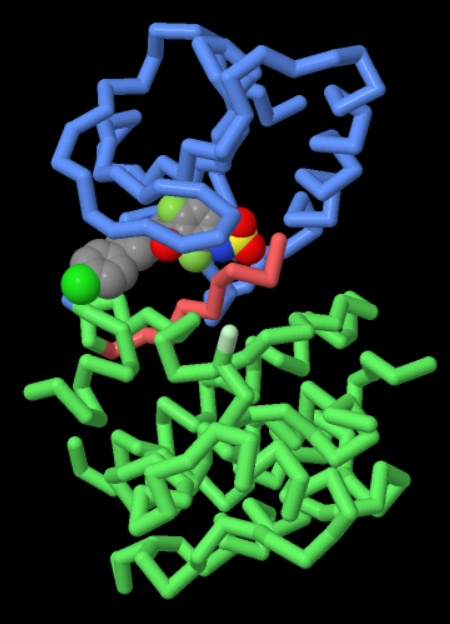
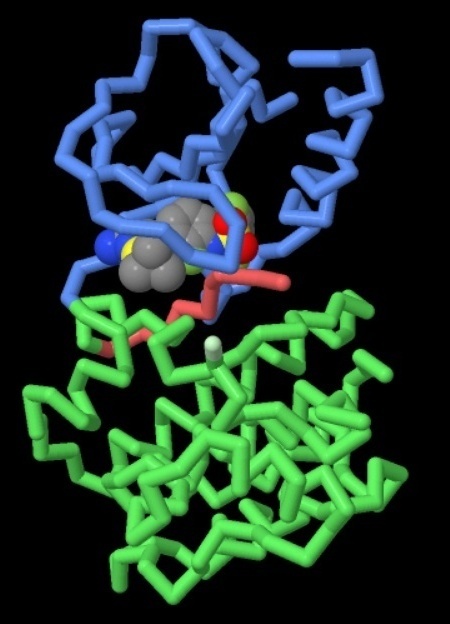
Nelle due figure qui sotto sono mostrate proteine RAF legate a tre diversi
farmaci:
nella figura di sinistra potete vedere alternativamente Vemurafenib
(file PDB 3og7) e Dabrafenib (file
PDB 4xv2)
nella figura a destra potete vedere alternativamente Dabrafenib
(file PDB 4xv2) e il farmaco sperimentale
PLX7904 (file PDB 4xv1).
Passando con la freccia del mouse sulle figure,
vedrete alternativamente i due farmaci indicati.
 . . .
. . . 
Spunti per ulteriori esplorazioni
Potete usare lo strumento "Protein Feature
View" per BRAF per scoprire se le strutture dei domini di RAF corrispondono
alla struttura di altre proteine
Molti dei farmaci che attaccano la proteina RAF sono stati progettati
per imitare la struttura di ATP, dato che ATP è essenziale nella
reazione di fosforilazione. Cercate di identificare le porzioni dei
farmaci che imitano la base azotata di ATP o il gruppo fosfato
 Bibliografia Bibliografia
4xv1, 4xv2: C. Zhang, W. Spevak, Y. Zhang,
E. A. Burton, Y. Ma, G. Habets, J. Zhang, J. Lin, T. Ewing, B. Matusow,
G. Tsang, A. Marimuthu, H. Cho, G. Wu, W. Wang, D. Fong, H. Nguyen,
S. Shi, P. Womack, M. Nespi, R. Shellooe, H. Carias, B. Powell, E. Light,
L. Sanftner, J. Walters, J. Tsai, B. L. West, G. Visor, H. Rezaei, P.
S. Lin, K. Nolop, P. N. Ibrahim, P. Hirth & G. Bollag (2015) RAF
inhibitors that evade paradoxical MAPK pathway activation. Nature
526, 583-586.
4mne: J. R. Haling, J. Sudhamsu, I. Yen, S. Sideris, W. Sandoval,
W. Phung, B. J. Bravo, A. M. Giannetti, A. Peck, A. Masselot, T. Morales,
D. Smith, B. J. Brandhuber, S. G. Hymowitz & S. Malek (2014) Structure
of the BRAF-MEK complex reveals a kinase activity independent role for
BRAF in MAPK signaling. Cancer Cell 26, 402-413.
3og7: G. Bollag, P. Hirth, J. Tsai, J. Zhang, P. N. Ibrahim,
H. Cho, W. Spevak, C. Zhang, Y. Zhang, G. Habets, E. A. Burton, B. Wong,
G. Tsang, B. L. West, B. Powell, R. Shellooe, A. Marimuthu, H. Nguyen,
K. Y. Zhang, D. R. Artis, J. Schlessinger, F. Su, B. Higgins, R. Iyer,
K. D’Andrea, A. Koehler, M. Stumm, P. S. Lin, J. Grippo, I. Puzanov,
K. B. Kim, A. Ribas, G. A. McArthur, J. A. Sosman, P. B. Chapman, K.
T. Flaherty, X. Xu, K. L. Nathanson & K. Nolop (2010) Clinical efficacy
of a RAF inhibitor needs broad target blockade in BRAF-mutant melanoma.
Nature 467, 596-599.
3kud: D. Filchtinski, O. Sharabi, A. Ruppel, I. R. Vetter, C.
Herrmann & J. M. Shifman (2010) What make Ras an efficient molecular
switch: a computational, biophysical, and structural study of Ras-GDP
interactions with mutants of Raf. Journal of Molecular Biology
399, 422-435.
1uwh: P. T. C. Wan, M. J. Garnett, S. M. Roe, S. Lee, D. Niculescu-Duvaz,
V. M. Good, C. M. Jones, C. J. Marshall, D. Barford & R. Marais
(2004) Mechanism of activation of the Raf-Erk signaling pathway by oncogenic
mutations of B-Raf. Cell 116, 855-867.
C. Wellbrock, M. Karasarides & R. Marais (2004) The Raf proteins
take centre stage. Nature Reviews Molecular Cell Biology 5,
875-885.
1far: H. R. Mott, J. W. Carpenter, S. Zhong, S. Ghosh, R. M.
Bell & S. L. Campbell (1996) The solution structure of the Raf-1
cysteine-rich domain: a novel ras and phospholipid binding site. Proceedings
of the National Academy of Science USA 93, 8312-8317.
|
|
|