|
|
Proteina Ras |
|||||
|
Molecola del Mese di Aprile 2012 La proteina RAS controlla la crescita e la moltiplicazione cellulare, mutazioni in questa proteina possono portare al cancro 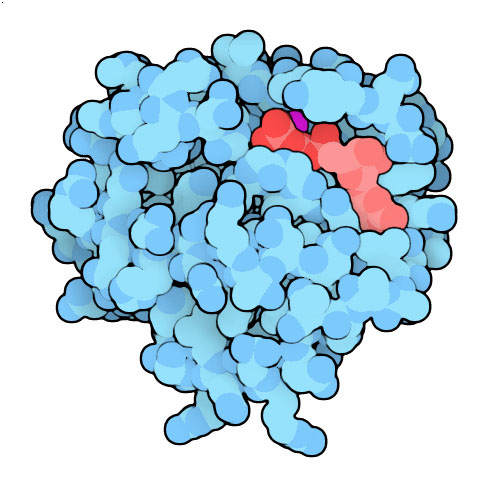 Introduzione IntroduzioneLe cellule mandano continuamente messaggi per comunicare quanti nutrienti vogliono, per sapere quando possono crescere e moltiplicarsi e per gestire le loro necessità interne. Questi messaggi devono essere forti e chiari perchè devono essere sentiti distintamente sovrastando il rumore di fondo delle normali attività del citoplasma. Un modo per rinforzare il segnale è di legarlo ad un processo irreversibile come la rottura di ATP o GTP. La proteina Ras, mostrata qui a destra (file PDB 5p21), usa questa strategia. Normalmente si trova nello stato inattivo e lega GDP. Si può trasformare nella forma attiva sostituendo GDP con GTP. Poi, dopo che il segnale è stato mandato, il GTP viene tagliato in GDP e fosfato disattivando la proteina. E' molto importante che la proteina Ras abbia uno stato inattivo ben riconoscibile, perchè è direttamente coinvolta nel controllo della crescita cellulare e nella mitosi. Oncogene Ras Alcuni geni del DNA, gli oncogeni, se mutati, possono provocare il cancro. Uno dei primi oncogeni scoperti è stato quello che codifica per la proteina Ras. La mutazione di un oncogene cambia la sequenza di amminoacidi e quindi la funzione della proteina codificata, che così può assumere proprietà pericolose che possono essere sfruttate dal cancro per crescere e diffondersi. Altri geni, gli oncosoppressori, se mutati, possono non riuscire più a contrastare il cancro, dato che codificano per proteine coinvolte nell'apoptosi, la morte cellulare programmata, come il soppressore di tumore p53, (mdm 7-2002). Le mutazioni che provocano il cancro, sugli oncogeni o sugli oncosoppressori, consentono alle cellule di evitare i controlli rigorosi che normalmente impediscono la crescita cellulare incontrollata. La proteina Ras è coinvolta nei segnali tra le cellule che controllano la crescita e la moltiplicazione cellulare. La mutazione della proteina Ras che può produrre il cancro, la obbliga a rimanere sempre nella forma attiva. Questo è un disastro perchè la proteina Ras mutata stimola continuamente la crescita delle cellule del cancro vincendo i normali freni che controllano la crescita cellulare. 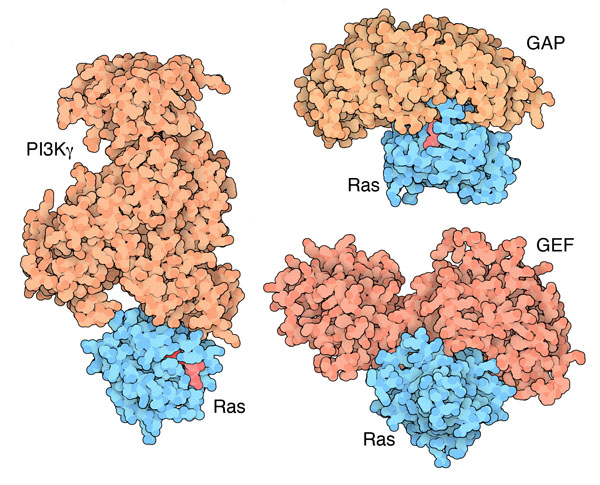 I
partner della proteina Ras I
partner della proteina RasRas si trova nel mezzo di una complessa rete di segnalazione che trasmette messaggi di crescita ed è affiancata da molte altre proteine. Le proteine GEF (Guanina nucleotide Exchange Factor), come Sos-1 mostrata qui a fianco (file PDB 1bkd), hanno il compito di attivare Ras. Forzano l'apertura del sito di legame della proteina Ras e così consentono a GDP di uscire. Nel sito di legame vuoto entra quindi GTP che rende la proteina Ras attiva e la fa interagire con effettori come PI3Kgamma (file PDB 1he8), una proteina chinasi che fosforila altre proteine della rete di segnalazione. Le proteine GAP (GTPase Activating Protein) come p120 GAP mostrata qui a fianco (file PDB 1wq1), hanno invece il compito di disattivare Ras. Inseriscono un amminoacido di arginina nel sito di legame di Ras, questa, insieme con la glutammina 61 di Ras, favorisce il trasferimento di un fosfato dal GTP che si trasforma in GDP. Ras, comunque sa inattivarsi anche da sola rompendo il legame fosfato del GTP con la sola glutammina 61, però lo fa molto più lentamente. Esplorando la struttura Sono state determinate molte strutture della proteina Ras che hanno permesso di individuare le diverse conformazioni che la proteina assume nelle varie fasi del suo ciclo di segnalazione. Le due strutture illustrate qui sotto mostrano Ras con GTP (fig. a) e con GDP (fig. b) (guanosina e ribosio bianchi, i tre gruppi fosfato rosa, arancione e rosso), quindi nei due stati attivo e inattivo (file PDB 5p21 e 4q21). Il confronto tra queste due strutture mostra che due tratti della proteina (verdi) che circondano il nucleotide cambiano conformazione. Questo è il segnale che viene riconosciuto dagli effettori e che innesca la cascata di fosforilazioni. Queste due strutture hanno rivelato l'importanza della glutammina 61 (sfere verdi) che tiene nella giusta posizione una molecola d'acqua (non mostrata) che realizza la reazione di idrolisi dell'estere fosforico. 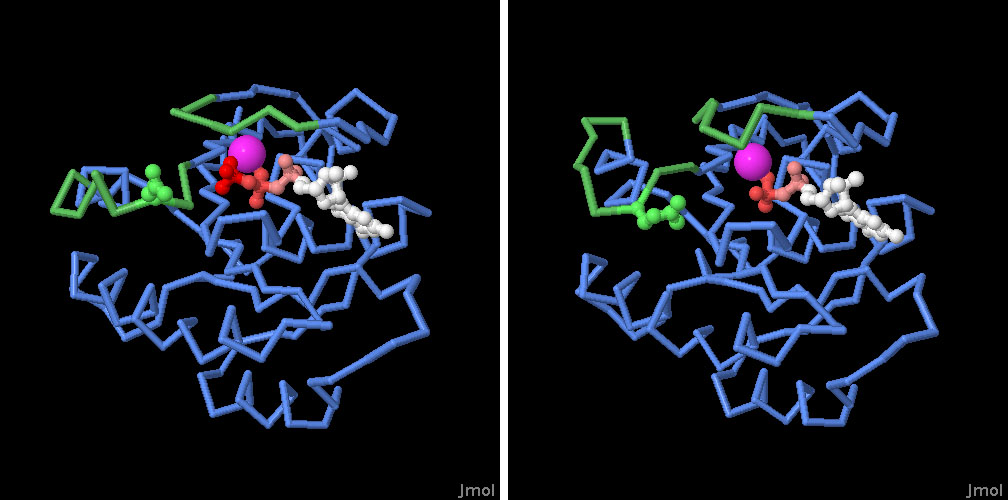 fig. a . . . . . . . . . . . . . . . . . . . . .
. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
. . . . fig. b
fig. a . . . . . . . . . . . . . . . . . . . . .
. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
. . . . fig. bLa struttura illustrata qui sotto a sinistra (file PDB 1wq1) mostra Ras legata alla proteina GAP (catena rosso scuro). GAP introduce un'arginina (non evidenziata) alle spalle del fosfato da rimuovere (rosso) e inoltre fa avvicinare al fosfato la glutammina 61 (sfere verdi) e una molecola di acqua (gialla). In questo modo la rimozione del fosfato risulta molto facile. La glutammina 61 è spesso mutata nelle cellule tumorali e quindi il GTP non può essere idrolizzato (nemmeno con l'aiuto di GAP) e così la proteina Ras resta sempre nella forma attiva inducendo le cellule cancerose a crescere senza controllo. 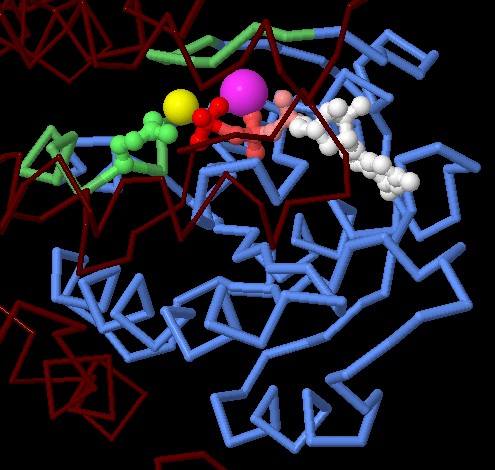 .
. 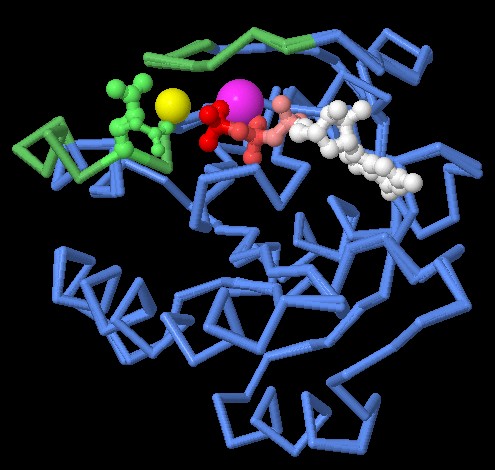 fig. c . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . fig. d La struttura illustrata qui sopra a destra (file PDB 3tgp) è stata ottenuta con una nuova tecnica cristallografica che rispetta maggiormente la vera struttura delle proteine a temperatura ambiente (mentre tradizionalmente i cristalli di proteine si ottenevano raffreddando le soluzioni proteiche a -180 °C e questo poteva alterare significativamente la struttura 3d della proteina). La proteina Ras mostrata qui sopra a destra è lievemente diversa da quella in fig.a. La glutammina 61 può assumere due conformazioni evidenziate con sfere verdi grosse o piccole. La glutammina 61 con sfere verdi piccole e la molecola di acqua (gialla) si trovano quasi nella stessa posizione della figura precedente (fig. c) che era aiutata dalla proteina GAP. Questo giustifica la capacità della proteina Ras di idrolizzare da sola GTP seppure più lentamente di quanto avviene con l'aiuto di GAP. Spunti per ulteriori esplorazioni Negli archivi PDB potete trovare molte forme mutate di Ras, comprese quelle con mutazioni a livello della glutammina 61. Usate lo strumento "Structure Comparison" nel sito PDB per confrontare queste strutture con quelle normali di Ras. Le strutture di Ras che dovrebbero contenere GTP in realtà contengono forme modificate di GTP, con azoto o carbonio legati ai gruppi fosfato. Perchè è necessario inserire GTP mutati nelle strutture che vengono risolte con la diffrazione ai raggi X? Bibliografia Y. Pylayeva-Gupta, E. Grabocka and D. Bar-Sagi (2011) Ras oncogenes: weaving a tumorigenic web. Nature Reviews Cancer 11, 761-774. I. R. Vetter, A. Wittinghofer (2001) The guanine nucleotide-binding switch in three dimensions. Science 294, 1299-1304.
|
||||||
|
|
||||||
