|
Molecola del Mese di Febbraio 2012
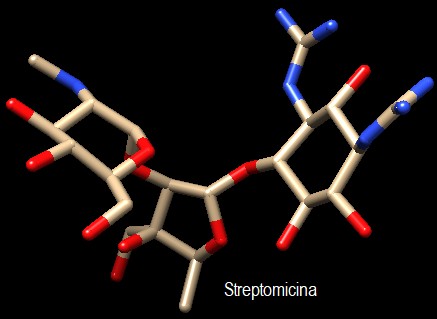
I batteri resistenti agli antibiotici amminoglicosidici, come la streptomicina,
hanno enzimi che modificano l'antibiotico o il suo bersaglio
Introduzione
La scoperta della streptomicina nel 1944 ha
fornito la prima vera cura della tubercolosi. Da quel momento è
cominciata una battaglia sempre più intensa con i batteri usando
streptomicina ed altri antibiotici amminoglicosidici. I chimici hanno
scoperto molti aminoglicosidi naturali sintetizzati dai batteri e hanno
sintetizzato nuovi antibiotici basati su queste efficaci difese
naturali. A loro volta, i batteri hanno sviluppato varie strategie
per proteggersi dall'attacco degli antibiotici. Hanno sviluppato metodi
per impedire l'ingresso del farmaco nelle cellule, pompe che espellono
il farmaco dalle cellule (mdm 11-2007 trasportatori
multifarmaco), enzimi che modificano la molecola bersaglio
dell'antibiotico o che modificano lo stesso antibiotico (mdm
7-2015 New
Dehli - metallo beta lattamasi)
 Modificare
il bersaglio Modificare
il bersaglio
Il metodo principale con cui i batteri della tubercolosi
sono diventati resistenti alla streptomicina è stato quello di
modificare il bersaglio del farmaco.
Gli amminoglicosidi attaccano i ribosomi come è mostrato più
sotto in dettaglio. Il farmaco si lega in una stretta tasca della subunità
minore del ribosoma, e così compromette il corretto appaiamento
di codoni e anticodoni durante la sintesi proteica.
I batteri resistenti hanno sviluppato enzimi che aggiungono gruppi
metilici a specifiche basi dei loro ribosomi, rendendo questa
tasca un pò troppo piccola per legare l'antibiotico.
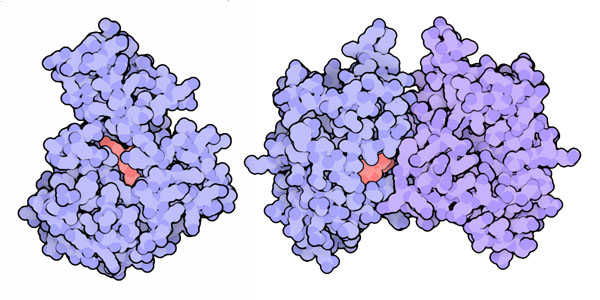
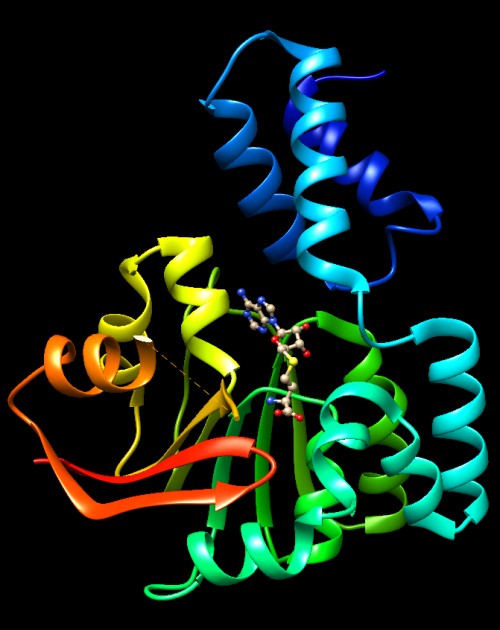
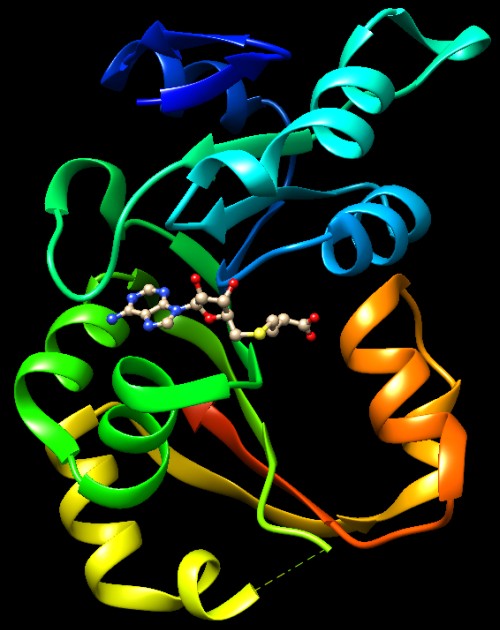
A fianco e qui sotto sono mostrati due di questi enzimi: l'enzima sulla
sinistra aggiunge un gruppo metilico alla guanina1405 del ribosoma (file
PDB 3frh),
l'enzima sula destra modifica l'adenina 1408 (file PDB 3pb3).
Nel sito attivo dei due enzimi si può vedere il nucleotide che
deve essere metilato.
 Attaccare
gli antibiotici Attaccare
gli antibiotici
Mentre i chimici, oltre alla streptomicina
mostrata qui sotto, hanno sviluppato tutta una serie di nuovi amminoglicosidi,
compresi alcuni in grado di legarsi anche ai ribosomi metilati, i batteri
hanno sviluppato metodi per attaccare direttamente il farmaco. Sono state
scoperte decine di enzimi di questo tipo in diverse classi di batteri,
e inoltre i batteri possono scambiarsi questi enzimi scambiandosi plasmidi,
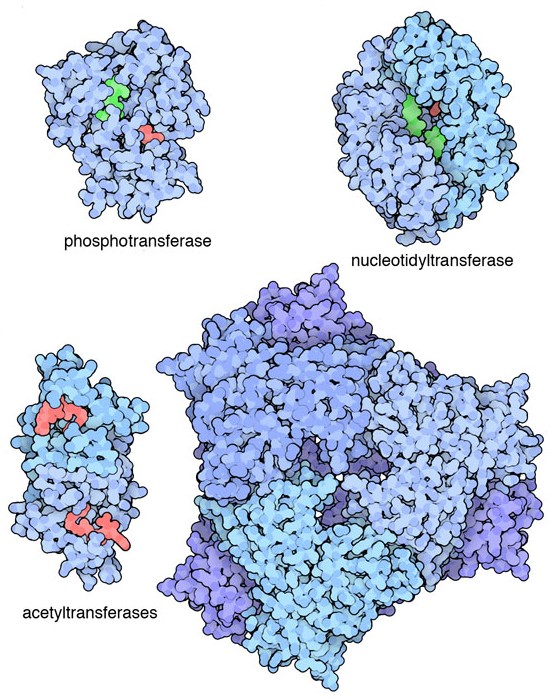
piccoli tratti di DNA circolare. Questi enzimi sono di tre categorie,
alcuni legano un fosfato al farmaco, altri aggiungono un acetile,
altri, infine aggiungono AMP. Le strutture di questi enzimi sono
diverse tra loro come sono diverse le loro reazioni. Qui ne sono mostrati
quattro:
una fosfotrasferasi (file PDB 1l8t),
una nucleotidil-trasferasi (file PDB 1kny),
una acetil-trasferasi (file PDB 1bo4),
e infine una insolita e voluminosa acetil-trasferasi che può modificare
il farmaco in quattro punti diversi (file PDB 3r1k).
Esplorando la struttura
Un'interessante serie di quattro strutture (file
PDB 1j5e, 1ibm,
1ibk, 1ibl)
ha rivelato i dettagli molecolari del modo in cui agiscono gli amminoglicosidi.
Il trattamento con amminoglicosidi costringe il batterio a compiere
molti errori nella sintesi delle sue proteine e queste proteine difettose
alla fine lo uccidono. Confrontando la struttura di ribosomi normali
con quella di ribosomi legati all'antibiotico, i ricercatori hanno scoperto
che l'antibiotico compromette la capacità di correzione degli
errori di lettura del ribosoma, consentendo a tRNA errati di legarsi
ai codoni dell'mRNA.
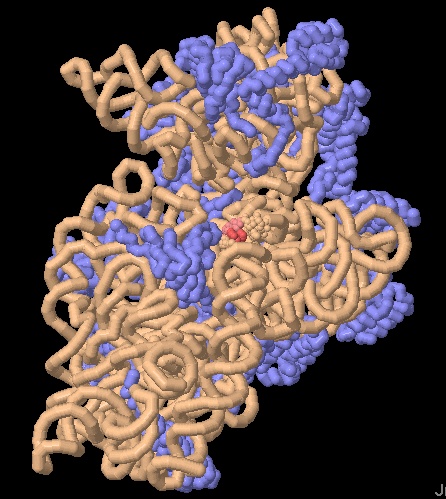
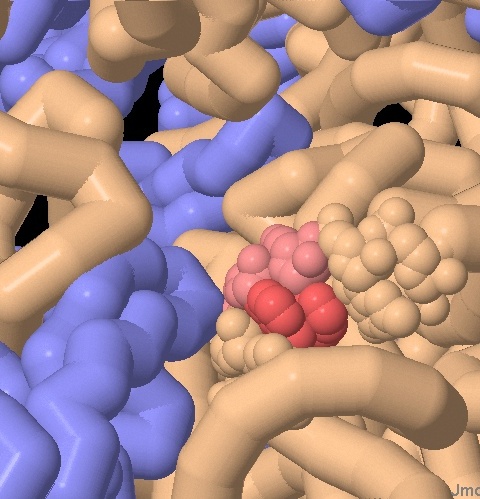
Le due immagini qui sotto sono della prima struttura che mostra la subunità
minore 30S del ribosoma con l'RNA beige e le proteine blu. Un particolare
avvolgimento dell'RNA è rosa e due importanti nucleotidi (adenina
1492 e 1493) sono rossi. Questi si vedono più in dettaglio nella
immagine ingrandita qui sotto a destra, notate la posizione di riposo
assunta dalle due adenine rosse.
 . . . .
. . . . 
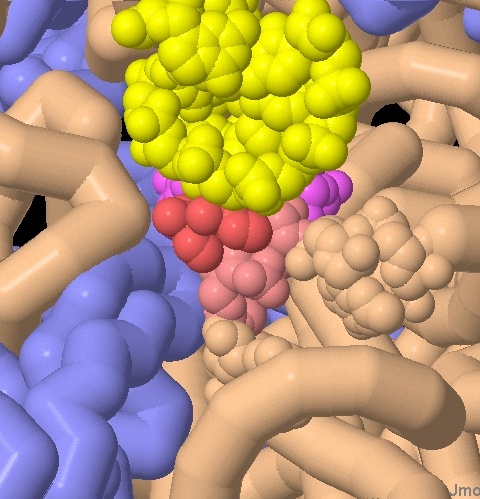
Nella seconda struttura, qui sotto a sinistra, si vede un piccolo pezzo
di tRNA (giallo) e di mRNA (magenta, un po' nascosto) che stanno interagendo
all'interno del ribosoma. Notate che le due adenine rosse si sono spostate
in fuori per interagire col complesso tRNA-mRNA, e questa torsione
richiede una certa energia che solo un forte accoppiamento codone-anticodone
può garantire. Se l'accoppiamento tra le triplette di tRNA
e mRNA non è quello ideale, il tRNA non avrà la forza
di restare legato e contemporaneamente mantenere in torsione le due
adenine.
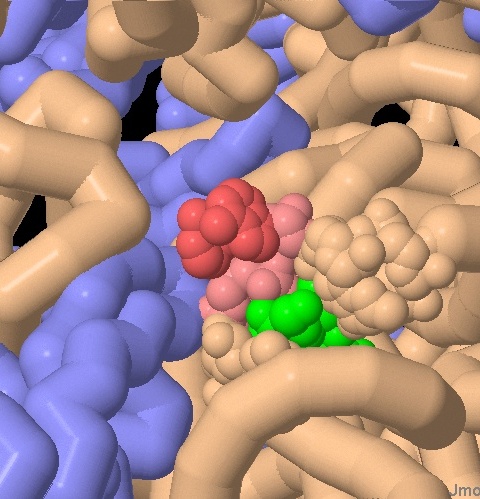
Nella terza struttura, qui sotto a destra, si vede il ribosoma legato
alla paromomicina (verde), un antibiotico amminoglicosidico.
Notate che si è legato nella piccola tasca normalmente occupata
dalle due adenine nel ribosoma libero. Questo costringe le adenine a
rimanere sempre nella posizione ruotata verso l'alto.
 . . .
. . . 
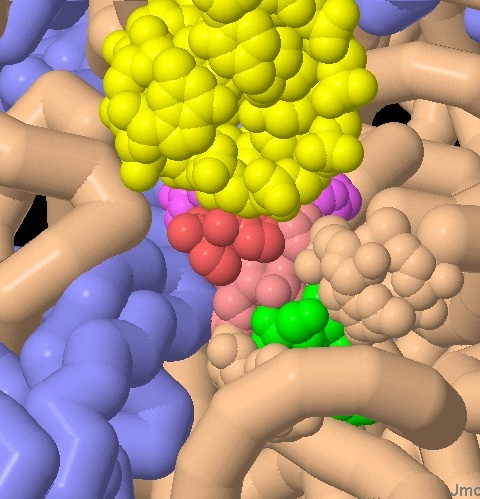
Nella quarta struttura, qui sotto, si vede il complesso tRNA-mRNA legato
nel ribosoma in presenza dell'antibiotico (verde). Dato che le due adenine
sono costrette sempre in posizione ruotata, il tRNA si può legare
al mRNA anche con legami deboli, con accoppiamenti codone-anticodone
non esatti, questo consente al ribosoma di utilizzare anche tRNA
errati nella sintesi delle proteine e di produrre quindi proteine
difettose
Spunti per ulteriori esplorazioni
Nel sito PDB ci sono molte altre strutture di enzimi che modificano
gli antibiotici amminoglicosidici . Potete trovare sia le strutture
degli enzimi da soli che quelle che contengono anche antibiotici e coenzimi.
I ricercatori stanno cercando di combattere la resistenza agli antibiotici
sviluppando inibitori che blocchino questi enzimi. Per trovarli, cercate
negli archivi PDB `aminoglycoside inhibitor`.
 Bibliografia Bibliografia
M. S. Ramirez, M. E. Tolmasky (2010) Aminoglycoside
modifying enzymes. Drug Resistance Update 13, 151-171.
S. B. Vakulenko, S. Mobashery (2003) Versatility of aminoglycosides
and prospects for their future. Clinical Microbiology Reviews
16, 430-450.
J. M. Ogle, D. E. Brodersen, W. M. Clemons Jr., M. J. Tarry,
A. P. Carter and V. Ramakrishnan (2001) Recognition of cognate transfer
RNA by the 30S ribosomal subunit. Science 292, 897-902.
G. D. Wright (1999) Aminoglycoside-modifying enzymes. Current
Opinion in Microbiology 2, 499-503.
|
|
|
 Modificare
il bersaglio
Modificare
il bersaglio

 Attaccare
gli antibiotici
Attaccare
gli antibiotici

 . . . .
. . . . 
 . . .
. . . 
