|
Molecola del mese
di aprile 2024
Il batteriofago phiX174 produce una piccola
proteina capace di uccidere i batteri.
 Introduzione Introduzione
I virus batteriofagi sono i predatori naturali dei batteri. Un batteriofago
si lega alla superficie del batterio e inietta il suo genoma che spesso
è costituito da una singola catena di DNA. Questo genoma, poi,
dirige la sintesi di centinaia di nuove copie del batteriofago che escono
dalla cellula del batterio uccidendolo.
I virus batteriofagi si trovano ovunque nell'ambiente e la maggior parte
dei batteri hanno diversi tipi di batteriofagi che li attaccano.
Per esempio, due dei batteriofagi più studiati, T4 e phiX174,
attaccano il batterio Escherichia coli.
 Rompere
la parete cellulare Rompere
la parete cellulare
Dopo che i virus batteriofagi si sono moltiplicati all'interno della
cellula batterica devono affrontare una nuova sfida: uscire all'esterno.
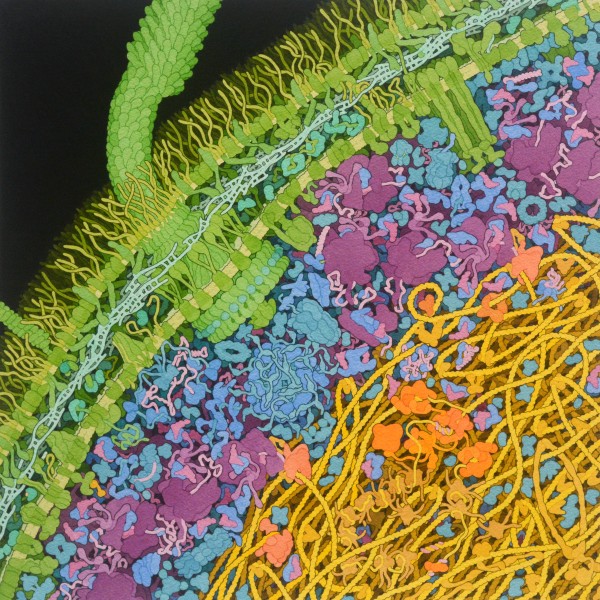
La cellula dei batteri, spesso, è circondata da una strato di
peptidoglicano, una struttura molto robusta a forma di rete composta
di proteine e zuccheri mostrata qui a lato in ciano tra le due membrane,
verdi, di Escherichia coli.
I batteriofagi hanno bisogno di proteine specializzate per rompere questa
parete protettiva.
Il batteriofago T4 usa l'enzima lisozima
(mdm 9-2000) (che abbiamo anche noi nella saliva) per tagliare le catene
di peptidoglicano. L'attacco di T4 ai batteri è mostrato nella
prossima immagine qui sotto.
Il batteriofago phiX174, invece, produce una piccola proteina, chiamata
proteina E, che blocca gli enzimi che sintetizzano la parete
di peptidoglicano
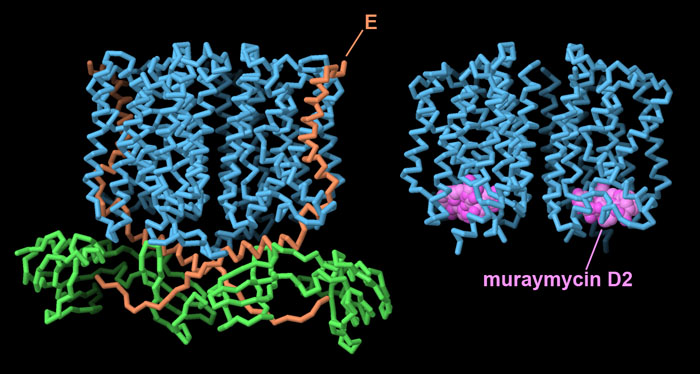
Complesso YES con la proteina E
La struttura PDB 8g02, illustrata nella
prima immagine in alto, mostra la proteina E in azione legata all'enzima
batterico transmembrana MraY, che ha un ruolo centrale nella sintesi
del peptidoglicano.
Il compito di MraY è quello di legare un lipide a dei pentapeptidi
precursori del peptidoglicano. Il lipide permette ad un'altra proteina
di spostare fuori dalla cellula i precursori e di legarli al peptidoglicano
in crescita.
La proteina E blocca questo processo legandosi a MraY nel suo sito attivo.
La proteina E richiama poi una seconda proteina, la proteina chaperone
SlyD, che circonda e protegge la proteina E e blocca del tutto il sito
attivo di MraY.
Questo complesso, chiamato YES, è mostrato qui in alto e include
l'enzima MraY, la proteina E e SlyD.
 Fago-terapia Fago-terapia
Dato che i virus batteriofagi uccidono i batteri possono diventare utili
per trattare le infezioni. La fago-terapia è conosciuta da oltre
un secolo, nel 1919 è stata usata per la prima volta per trattare
la dissenteria. In seguito è caduta in disuso nella medicina
occidentale dopo la scoperta della penicillina
(mdm 5-2002) e di altri farmaci antibiotici. Gli antibiotici avevano
alcuni vantaggi importanti, erano più facili da somministrare
e potevano attaccare un più vasto spettro di batteri. Recentemente,
però, lo sviluppo di batteri resistenti agli antibiotici ha riacceso
l'interesse per la fago-terapia che può essere un'alternativa
importante quando non ci sono antibiotici efficaci.
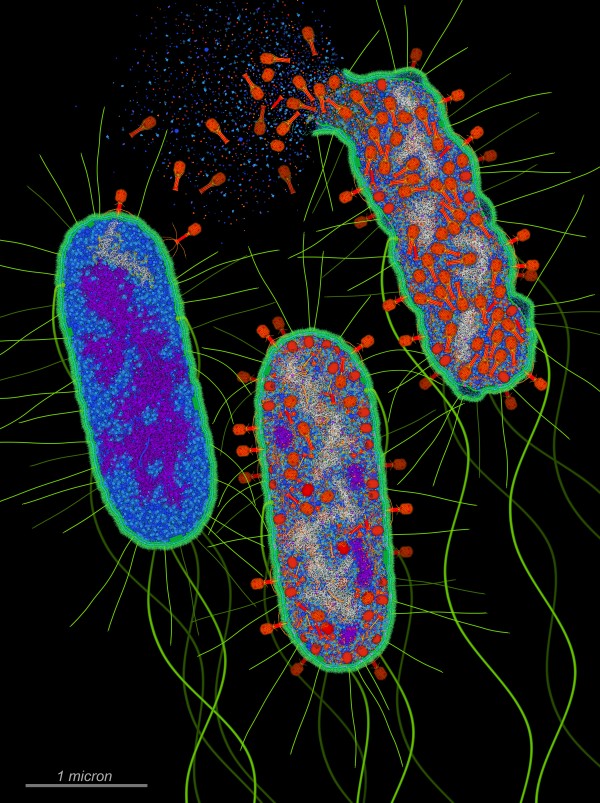
Qui a fianco è mostrato come i virus batteriofagi T4 attaccano
e uccidono i batteri di Escherichia coli.
La prima cellula batterica è appena stata attaccata da T4 che
sta iniettando il suo DNA genomico (bianco).
Nella seconda cellula batterica si vede che T4 ha preso il controllo
delle operazioni cellulari, ha distrutto quasi tutto il DNA batterico
(porpora) e sta costringendo la cellula a produrre molte copie di se
stesso.
Nella terza cellula batterica si vede che il virus T4 ha prodotto una
proteina che forma dei canali attraverso la membrana interna batterica
che fanno passare l'enzima virale lisozima che taglia la parete cellulare
di peptidoglicano (ciano). La cellula batterica, quindi, esplode liberando
centinaia di nuovi virus batteriofagi.
  Iniezione
di DNA Iniezione
di DNA
Le pareti cellulari dei batteri sono un problema anche nelle prime fasi
dell'infezione del batteriofago.
Molti batteriofagi, come T4, hanno delle code complesse che si legano
alla superficie del batterio e iniettano il loro DNA attraverso la parete
cellulare.
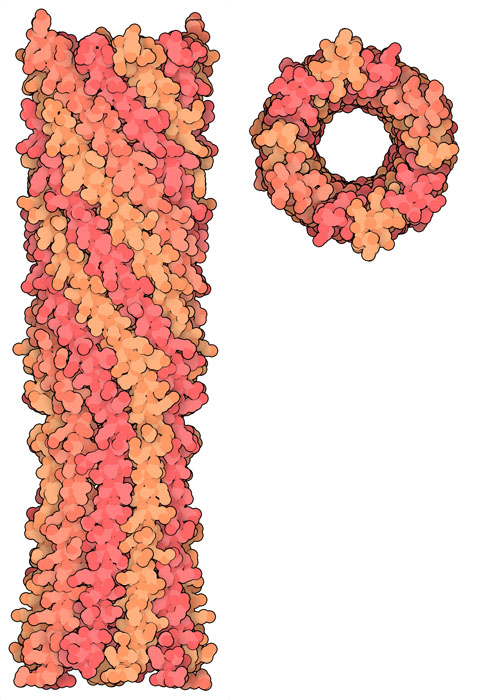
PhiX174 usa un approccio più semplice. Produce una proteina che
si avvolge formando un tubo tra le maglie del peptidoglicano e così
forma un passaggio abbastanza grande per il DNA virale.
Nel file PDB 4jpp si vede la porzione
della proteina che forma il tubo. E' costituita da 10 catene proteiche
ad alfa elica che si avvolgono tra loro per formare il canale. I due
domini teminali della struttura a tubo sono apolari e quindi possono
inserirsi nella membrana e sono adatti a perforare le due membrane,
interna ed esterna, di Escherichia coli.
 Esplorando
la struttura Esplorando
la struttura
Molti antibiotici attaccano gli enzimi batterici che costruiscono la
parete di peptidoglicano.
La penicillina, per esempio, blocca gli enzimi che uniscono tra loro
i precursori del peptidoglicano e così impedisce che si formi
il polimero a rete.
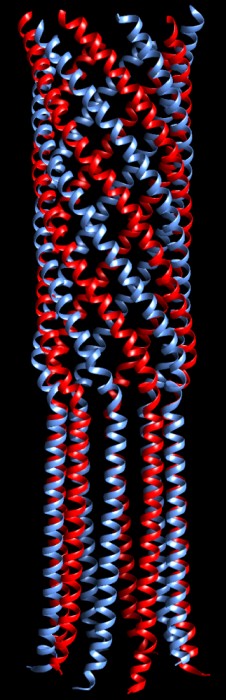
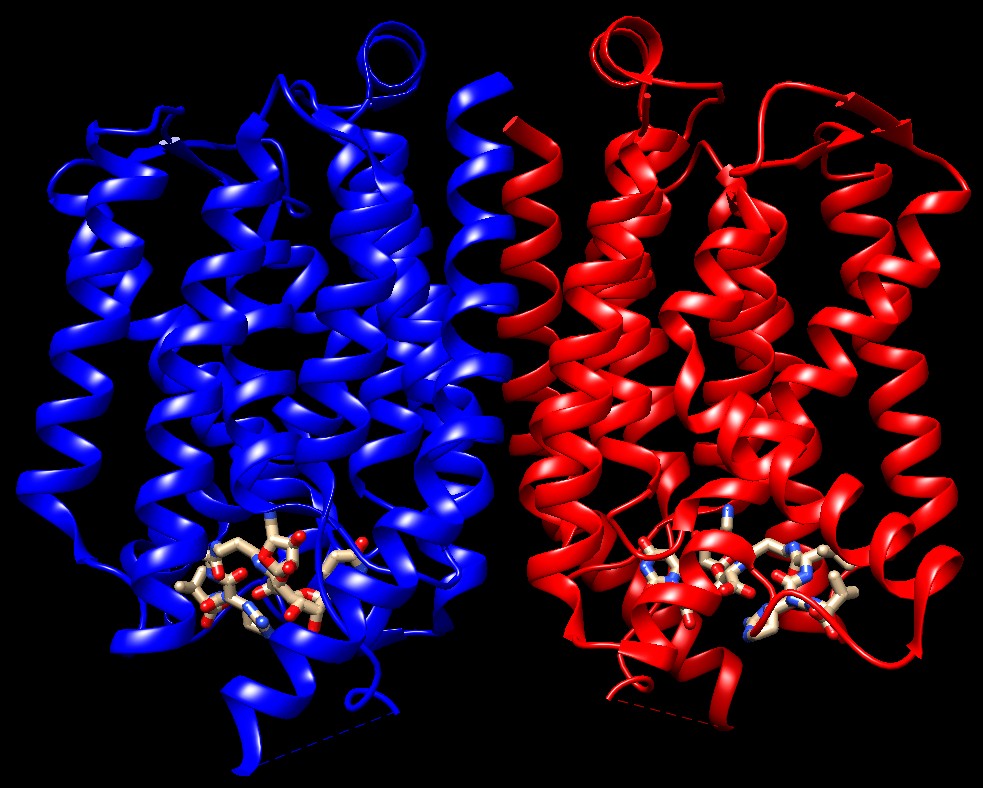
L'antibiotico naturale muraimicina D2
si lega all'enzima MraY (file PDB 5ckr)
e ne blocca il sito attivo in modo simile alla proteina E di phiX174.
Qui sotto è mostrato più in dettaglio il complesso tra
muraimicina D2 e MraY.
Spunti per ulteriori esplorazioni
Per saperne di più sui batteriofagi e su altri virus, andate
in Esplorando
la biologia strutturale dei virus
Per esplorare gli aspetti biologico-strutturali della resistenza agli
antibiotici andate su Risorse
al PDB 101.
 Bibliografia Bibliografia
8g02: Orta, A.K., Riera, N., Li, Y.E., Tanaka, S., Yun, H.G.,
Klaic, L., Clemons Jr., W.M. (2023) The mechanism of the phage-encoded
protein antibiotic from Phi X174. Science 381: eadg9091-eadg9091
Strathdee, S.A., Hatfull, G.F., Mutalik, V.K., Schooley, R.T.
(2023) Phage therapy: From biological mechanisms to future directions.
Cell 186, 17-31
5ckr: Chung, B.C., Mashalidis, E.H., Tanino, T., Kim, M., Matsuda,
A., Hong, J., Ichikawa, S., Lee, S.Y. (2016) Structural insights into
inhibition of lipid I production in bacterial cell wall synthesis. Nature
533: 557-560
4jpp: Sun, L., Young, L.N., Zhang, X., Boudko, S.P., Fokine,
A., Zbornik, E., Roznowski, A.P., Molineux, I.J., Rossmann, M.G., Fane,
B.A. (2014) Icosahedral bacteriophage Phi X174 forms a tail for DNA
transport during infection. Nature 505: 431-435
|
|
|