|
|
Cellulasi e bioenergia |
|||||
|
Molecola del mese di maggio 2023 Le cellulasi sono enzimi fungini che idrolizzano la cellulosa e sono usati nella produzione industriale di etanolo da materiali di scarto vegetali 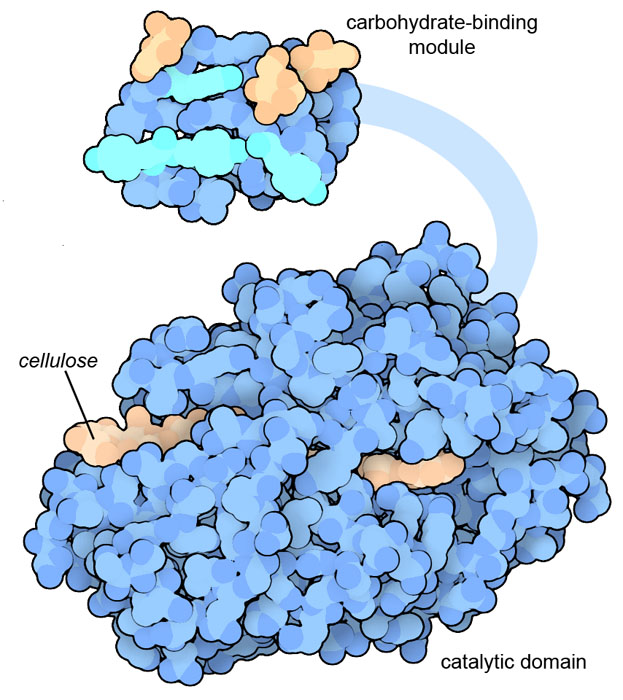 Introduzione IntroduzioneI combustibili derivati dal petrolio si stanno progressivamente esaurendo e inoltre sono i maggiori responsabili del cambiamento climatico, così i ricercatori bioenergetici stanno cercando in natura nuove sostanze per produrre combustibili più puliti e rinnovabili per soddisfare la domanda mondiale di energia. L'etanolo si sta rivelando una delle sostanze più promettenti sia come combustibile, sia come intermedio per produrre un carburante sostenibile per l'aviazione. Le bevande alcoliche sono state prodotte sin dall'antichità fermentando gli zuccheri ed ora questa stessa tecnologia viene usata per produrre etanolo da usare come combustibile. Oggi il mais e la canna da zucchero forniscono la maggior parte di tali zuccheri, ma questa pratica non è sostenibile perchè, da un lato, sottrae terreno alla produzione di cibo e, dall'altro, perchè queste coltivazioni consumano molta energia. I ricercatori, quindi, stanno cercando di convertire in etanolo la cellulosa più dura proveniente da materiali vegetali non commestibili come gli sfalci di erbacce, il legno di pioppo, o gli steli delle piante di mais. Smontare la cellulosa Sia l'amido che la cellulosa sono costituiti da catene di molecole di glucosio, ma mentre nell'amido le molecole di glucosio sono legate con legami di tipo alfa che sono facilmente digeribili, nella cellulosa le molecole di glucosio sono legate con legami di tipo beta e formano catene molto robuste e compatte che inoltre si avvolgono tra loro per formare delle fibrille ancora più forti che i nostri enzimi digestivi non sono in grado di rompere. Per questo motivo i processi industriali che trasformano la cellulosa in glucosio per produrre etanolo con la fermentazione devono prima separare queste catene e tagliarle in singole molecole di glucosio. In genere, il materiale vegetale subisce un pretrattamento con processi aggressivi con sostanze chimiche e vapore surriscaldato per rompere le pareti cellulari ed esporre le fibre di cellulosa. Poi un insieme di enzimi chiamati genericamente cellulasi rompe le catene di cellulosa per liberare le singole molecole di glucosio. Dato che noi non possediamo enzimi cellulasi per digerire la cellulosa, li ricaviamo dai funghi come il Trichoderma reesei (o Hypocrea jecorina). Questo fungo è stato scoperto durante la seconda guerra mondiale nelle isole Solomon dove cresceva sulle stoffe di cotone. Oggi è largamente usato nella produzione di bioetanolo e anche per altri scopi come per ammorbidire il denim dei jeans durante il lavaggio con pietra pomice. 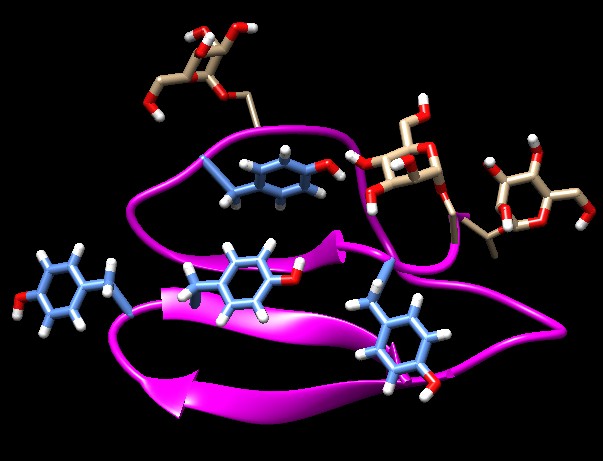 Scienza
della cellulasi Scienza
della cellulasiL'enzima Cel7A di Trichoderma reesei mostrato qui sopra (file PDB 7cel e 2mwk), contiene due domini che lo aiutano a degradare le robuste fibre della cellulosa. Il primo dominio, più grande, ha una lunga fenditura nella quale lega la catena di cellulosa. La sua forma è tale da mettere in tensione un legame glucosio-glucosio di fronte al sito catalitico che lo deve tagliare. Il secondo dominio, più piccolo (in alto e anche qui a destra), contiene alcuni amminoacidi di tirosina (ciano) e molecole di zucchero (mannosio, beige) legate a gruppi alcolici della proteina, che gli consentono di aderire alla catena di cellulosa e così permette all'enzima di trovarsi sempre nella giusta posizione per realizzare molte reazioni di taglio in rapida successione. 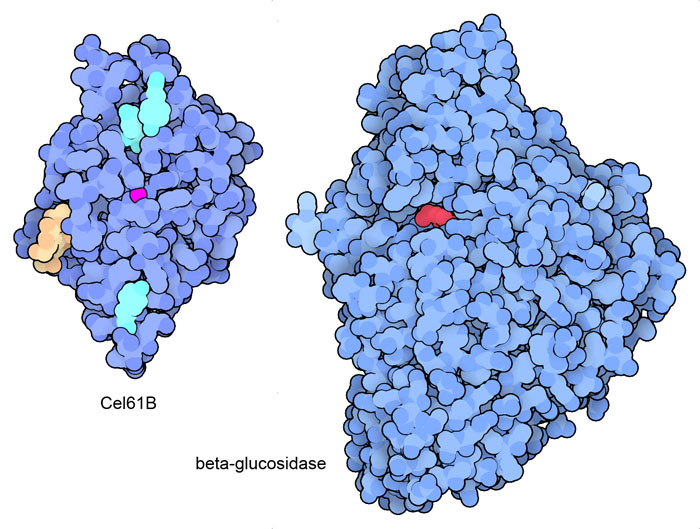 Cocktail
di cellulasi Cocktail
di cellulasiI funghi come Trichoderma reesei secernono un cocklail complesso di enzimi che operano insieme per degradare le fibre di cellulosa. Qui sono mostrati due esempi, ma negli archivi PDB ce ne sono molti altri. Il primo enzima mostrato qui a lato è Cel61B (file PDB 2vtc), una glicoside idrolasi, ma è anche chiamato endoglucanasi 7 perchè taglia la cellulosa in frammenti più corti. Opera in modo aggressivo usando un atomo di nichel (magenta) per generare la forma attivata dell'ossigeno che poi attacca la cellulosa. Anche Cel61B ha molte tirosine (ciano) e molecole di zucchero (mannosio, beige) legate alla proteina che aiutano l'enzima ad afferrare la catena di cellulosa. Il secondo enzima è una beta-glucosidasi (file PDB 3zxy) che entra in azione in un secondo momento per tagliare i corti frammenti di cellulosa, prodotti dagli altri enzimi, e liberare le singole molecole di glucosio. 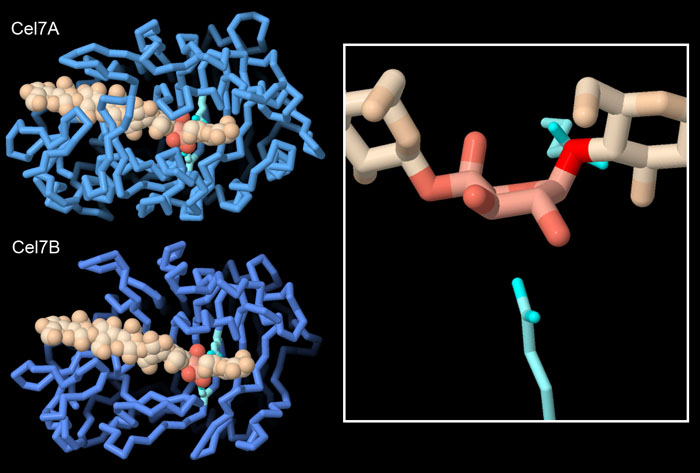 Esplorando
la struttura Esplorando
la strutturaGli enzimi cellulasi usano varie strategie molecolari per rendere più efficace la loro azione. Come visto prima, hanno spesso un secondo dominio che si lega alla superficie delle fibre di cellulosa. Inoltre, i vari enzimi legano la cellulosa con siti di struttura diversa per realizzare al meglio la loro specifica funzione. Nell'enzima Cel7A (file PDB 4c4c), alcuni segmenti della proteina abbracciano la catena di cellulosa formando un tunnel che ha il sito attivo sul fondo. In questo modo, Cel7A può staccare dei dimeri dalla fine del filamento di cellulosa cioè dei piccoli frammenti formati da due glucosi. L'enzima Cel7B (endogluconasi EG-1, file PDB 1eg1), invece, ha una fenditura aperta che si lega alla cellulosa in un punto casuale e gli consente di tagliare la catena nel mezzo formando segmenti più lunghi. L'immagine qui a fianco sulla destra mostra in dettaglio un'altra strategia molecolare: questi enzimi spesso distorgono l'anello di un glucosio (mostrato qui in rosa) e lo costringono a passare dalla forma più stabile a sedia ad una forma instabile chiamata a mezza sedia (mostrata in figura). Questa deformazione destabilizza il legame dell'ossigeno che unisce due anelli (rosso) e facilita la reazione di taglio realizzata da due amminoacidi di acido glutammico (ciano). Spunti per ulteriori esplorazioni Per trovare altre strutture di cellulasi, fate una ricerca usando i numeri EC nell' Enzyme Classification Browser che individuano con precisione il tipo di enzima. Usate 3.2.1.4 per gli enzimi cellulasi, e 3.2.1.91 per gli enzimi cellulosa 1,4-betacellobisidasi. Per vedere il segmento flessibile che unisce i due domini di Cel7A, cercate AF_AFP62694F1 e vedrete la struttura prevista dal software AlphaFold2. Notate che ci sono diverse possibilità nel tratto finale e questo aiuta il filamento ad essere flessibile, e ci sono molte treonine e serine (amminoacidi alcolici) che si prestano ad essere glicosilate. Infine notate che spesso troverete nomi diversi per lo stesso enzima. Per esempio, il nome breve Cel7A è basato su una classificazione delle famiglie di cellulasi più generale, ma lo stesso enzima può essere nominato descrivendone in dettaglio l'azione chimica che è quella di tagliare unità di due glucosi dalla fine della catena di cellulosa, per cui può essere chiamato 1,4-beta-D-glucano cellobioidrolasi I o cellulosa 1,4-beta cellobiosidasi o esoglucanasi I. Robak, K., Balcerek, M. (2020) Current state-of-the-art in ethanol production from lignocellulosic feedstocks. Microbiol Res 240: 126534 2mwk: Happs, R.M., Guan, X., Resch, M.G., Davis, M.F., Beckham, G.T., Tan, Z., Crowley, M.F. (2015) O-glycosylation effects on family 1 carbohydrate-binding module solution structures. FEBS J 282: 4341-4356 Payne, C.M., Knott, B.C., Mayes, H.B., Hansson, H., Himmel, M.E., Sandgren, M., Stahlberg, J., Beckham, G.T. (2015) Fungal cellulases. Chem Rev 115: 1308-1448 3zyz: Karkehabadi, S., Helmich, K.E., Kaper, T., Hansson, H., Mikkelsen, N.E., Gudmundsson, M., Piens, K., Fujdala, M., Banerjee, G., Scott-Craig, J.S., Walton, J.D., Phillips, G.N.J., Sandgren, M. (2014) Biochemical Characterization and Crystal Structures of a Fungal Family 3 Beta-Glucosidase, Cel3A from Hypocrea Jecorina. J Biol Chem 289: 31624-31637 4c4c: Knott, B.C., Haddad Momeni, M., Crowley, M.F., Mackenzie, L.F., Gotz, A.W., Sandgren, M., Withers, S.G., Stahlberg, J., Beckham, G.T. (2014) The Mechanism of Cellulose Hydrolysis by a Two-Step, Retaining Cellobiohydrolase Elucidated by Structural and Transition Path Sampling Studies. J Am Chem Soc 136: 321-329 2vtc: Karkehabadi, S., Hansson, H., Kim, S., Piens, K., Mitchinson, C., Sandgren, M. (2008) The First Structure of a Glycoside Hydrolase Family 61 Member, Cel61B from the Hypocrea Jecorina, at 1.6 A Resolution. J Mol Biol 383: 144-154 7cel: Divne, C., Stahlberg, J., Teeri, T.T., Jones, T.A. (1998) High-resolution crystal structures reveal how a cellulose chain is bound in the 50 A long tunnel of cellobiohydrolase I from Trichoderma reesei. J Mol Biol 275: 309-325 1eg1: Kleywegt, G.J., Zou, J.Y., Divne, C., Davies, G.J., Sinning, I., Stahlberg, J., Reinikainen, T., Srisodsuk, M., Teeri, T.T., Jones, T.A. (1997) The crystal structure of the catalytic core domain of endoglucanase I from Trichoderma reesei at 3.6 A resolution, and a comparison with related enzymes. J Mol Biol 272: 383-397 |
||||||
|
|
||||||
