|
|
Molnupiravir e COVID-19 |
|||||
|
Molecola del mese di ottobre 2021 Molnupiravir è il primo farmaco antivirale che sembra efficace contro COVID-19 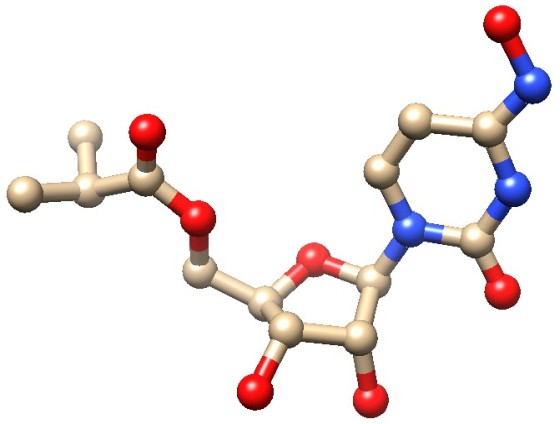 Introduzione IntroduzioneDall'inizio della pandemia da COVID-19 sono stati sperimentati molti farmaci antivirali nel tentativo di evitare che i pazienti andassero incontro alle complicazioni più insidiose della malattia. Nessuno di questi, però, si è rivelato abbastanza efficace da entrare nei protocolli ufficiali di terapia. Molnupiravir è un nuovo farmaco antivirale che in questi giorni è in via di approvazione negli Stati Uniti da parte della Food and Drug Administration con procedura d'emergenza perchè, nella sperimentazione di fase 3 su pazienti di COVID-19, ha dimostrato un'efficacia doppia rispetto al placebo nel ridurre il rischio di sviluppare sintomi gravi e di essere ricoverati in terapia intensiva. 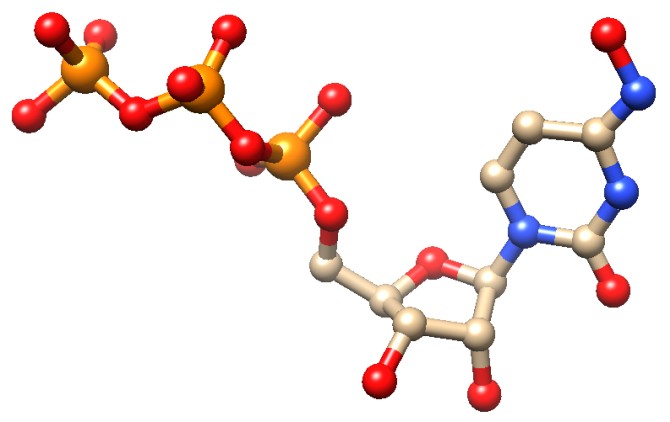 Profarmaco ProfarmacoLa struttura del Molnupiravir è mostrata qui in alto. Contiene un ribosio nella parte centrale legato sulla destra ad una base azotata simile alla Citosina alla quale è stato aggiunto in alto un gruppo OH (rosso) che la trasforma in N4-idrossicitosina NHC. Molnupiravir, in realtà, è un profarmaco cioè è una molecola di per sè inattiva, ma all'interno delle nostre cellule si trasforma nella forma attiva di trifosfato (mostrata qui a destra), pronta per essere incorporata nell'RNA virale. Bersaglio ideale 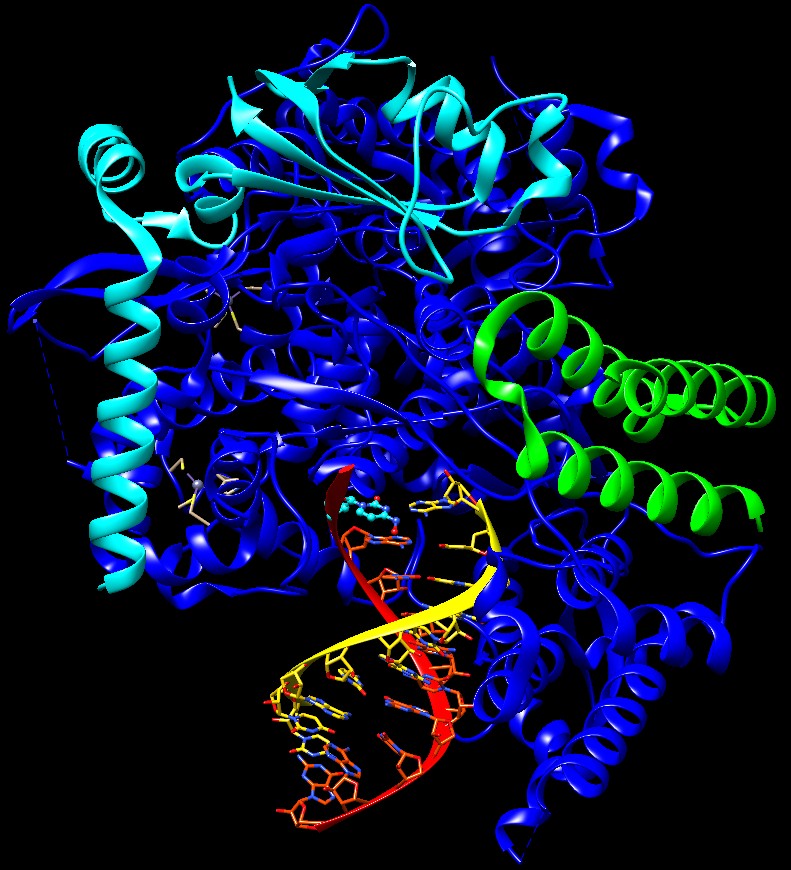 Il
bersaglio del Molnupiravir è la RNA
polimerasi RNA dipendente (Rd-RNApol) (mdm 9-2020) un enzima prodotto
dal COVID-19 indispensabile per il virus per duplicare l'RNA che trasporta.
Il COVID-19, infatti, è un virus a RNA che non ha bisogno del DNA
per duplicarsi. Usa la macchina biochimica della cellula ospite per trascrivere
il proprio RNA in proteine.Tra queste vi è l'enzima Rd-RNApol che
serve a formare molte copie dell'RNA virale. Il
bersaglio del Molnupiravir è la RNA
polimerasi RNA dipendente (Rd-RNApol) (mdm 9-2020) un enzima prodotto
dal COVID-19 indispensabile per il virus per duplicare l'RNA che trasporta.
Il COVID-19, infatti, è un virus a RNA che non ha bisogno del DNA
per duplicarsi. Usa la macchina biochimica della cellula ospite per trascrivere
il proprio RNA in proteine.Tra queste vi è l'enzima Rd-RNApol che
serve a formare molte copie dell'RNA virale.Dato che questo enzima non esiste nelle nostre cellule, è un bersaglio ideale per i farmaci antivirali. Nella molecola del mese del settembre 2020 abbiamo descritto un altro farmaco che attacca la Rd-RNApol, il Remdesivir un farmaco sviluppato contro Ebola che non si è rivelato abbastanza efficace contro COVID-19. Remdesivir agiva provocando terminazioni di catena durante la sintesi dell'RNA virale. Molnupiravir, invece, agisce inducendo l'enzima a commettere errori ripetuti di copiatura. In questo modo l'RNA copiato è del tutto inservibile, il virus non può duplicarsi e così si spegne l'infezione. Nella figura qui a fianco (file PDB 7ozu) si vede l'enzima Rd-RNApol mentre sta duplicando l'RNA virale. Nella parte bassa della figura si vedono le due catene di RNA. La catena rossa è quella del virus, nella quale è stata incorporata la Citosina idrossilata del Molnupiravir evidenziata con i carboni in azzurro. Di fronte alla Citosina idrossilata, sulla catena copiata, gialla, ci può essere la base corretta Guanina, ma anche la base alternativa errata Adenina come accade qui sopra. Accoppiamenti alternativi Grazie alla tecnica innovativa della microscopia crioelettronica, sono già disponibili le prime strutture che mostrano l'enzima Rd-RNApol mentre sta duplicando RNA virale che incorpora il nuovo farmaco Molnupiravir. Qui sotto si possono vedere in primo piano i due possibili accoppiamenti della citosina idrossilata del Molnupiravir. Sulla sinistra vediamo l'accoppiamento con la base azotata errata Adenina (file PDB 7ozu). Sulla destra vediamo l'accoppiamento con la base azotata corretta Guanina (file PDB 7ozv). 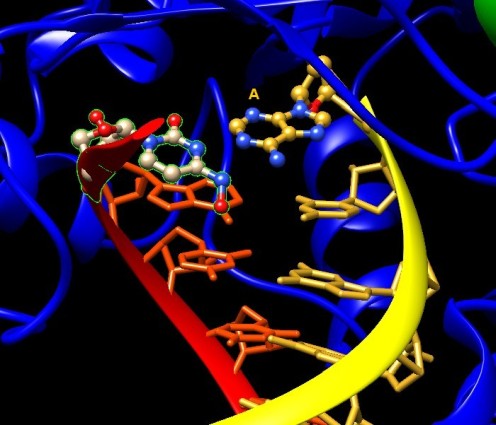
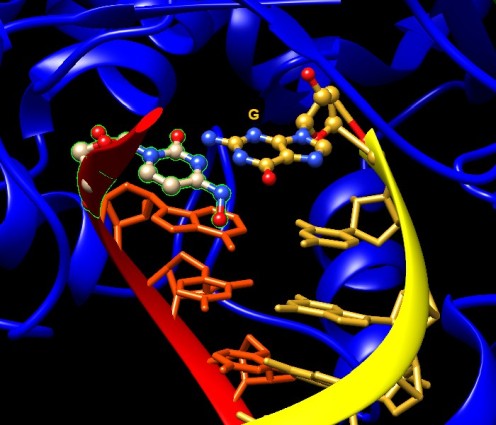 Esplorando la struttura Per capire cosa consente alla citosina idrossilata di accoppiarsi sia con Adenina sia con Guanina bisogna capire su quale logica si basa la differenza tra le basi azotate dell'RNA. Esaminiamo, per esempio Citosina e Uracile (che si accoppiano con Guanina e Adenina). La citosina ha l'azoto (blu) in alto in forma enolica, mentre l'uracile ha l'ossigeno (rosso) in alto in forma chetonica. . . . . . . . . . . . . . . . . 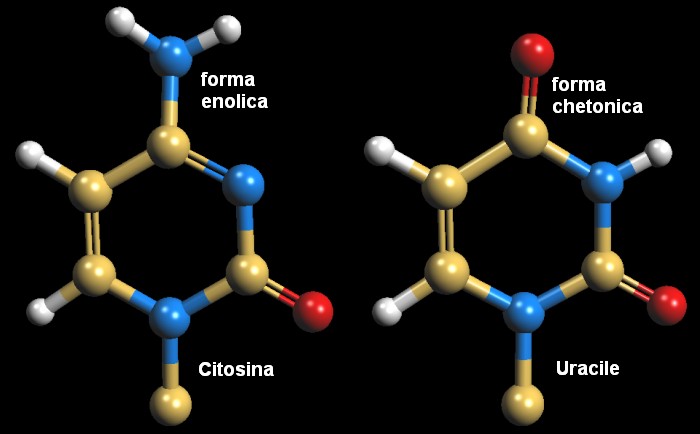
Le due forme enolica e chetonica si possono interconvertire tra loro, ma l'azoto (meno elettronegativo) esiste prevalentemente in forma enolica (1 milione contro 1) mentre l'ossigeno (più elettronegativo) esiste prevalentemente in forma chetonica (1 milione contro 1), questo garantisce una struttura stabile alle basi azotate che si accoppiano in modo errato solo 1 volta ogni milione di basi. Per creare il Molnupiravir, gli scienziati hanno legato all'azoto della citosina un ossigeno (rosso), questo ha aumentato di un po' l'elettronegatività dell'azoto al punto che le due forme enolica e chetonica hanno circa la stessa stabilità. . . . . . . . . . . . . . . . . 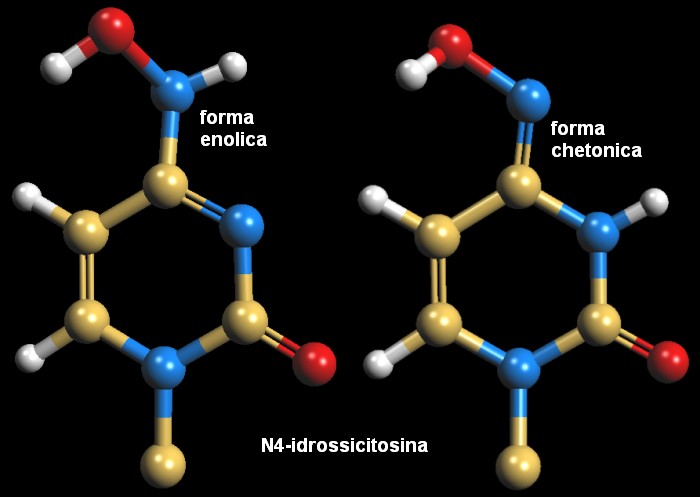 Quando la N-idrossicitosina NHC si trova nella forma enolica, come nell'immagine qui sotto, si accoppia con la Guanina, con la quale realizza tre legami idrogeno (puntini bianchi). . . . . . . . . . . . . . . . . 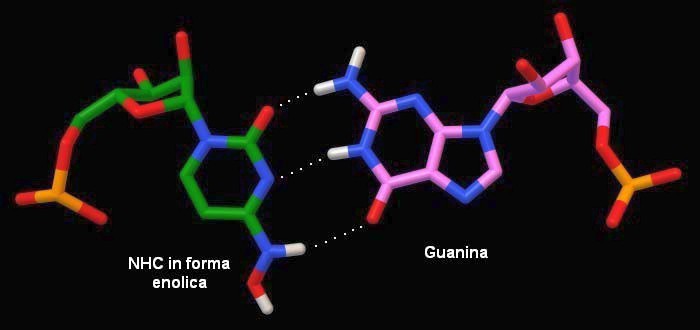 Quando, invece, la N-idrossicitosina NHC si trova nella forma chetonica, come nella prossima immagine, si accoppia con Adenina con la quale realizza due legami idrogeno. Dato che l'accoppiamento diventa del tutto casuale, si verificano continui errori nella catena di RNA copiato. . . . . . . . . . . . . . . . . 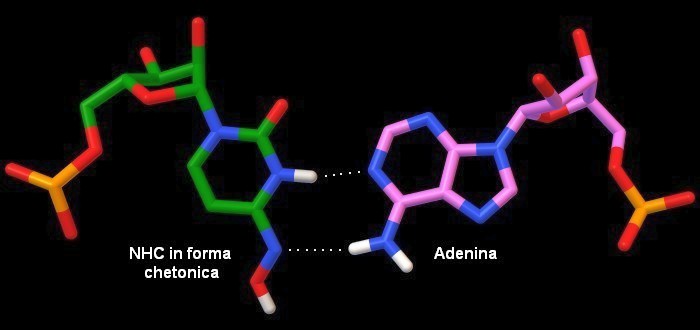 Spunti per ulteriori esplorazioni Per esplorare la struttura delle quattro basi azotate del DNA, cercate il file PDB 2rpd che contiene un singolo filamento di DNA con le quattro basi in sequenza. 7ozu, 7ozv: Kabinger, F., Stiller, C., Schmitzova, J., Dienemann, C., Kokic, G., Hillen, H.S., Hobartner, C., Cramer, P. (2021) Nat Struct Mol Biol 28: 740-746 Yang, X., Huang, Y., Gao, H., Liu, F., Ge, J., Sun, Q., Yang, X., Xu, W., Liu, Z., Yang, H., Lou, Z., Jiang, B., Guddat, L.W., Gong, P., Rao, Z. (2020) Structural Basis for RNA Replication by the SARS-CoV-2 Polymerase. Cell 182: 1-12.
|
||||||
|
|
||||||
