|
|
Nanopori per sequenziare il DNA |
|||||
|
Molecola del mese di settembre 2021 Nanopori sintetici con particolari enzimi realizzano il sequenziamento miniaturizzato del DNA 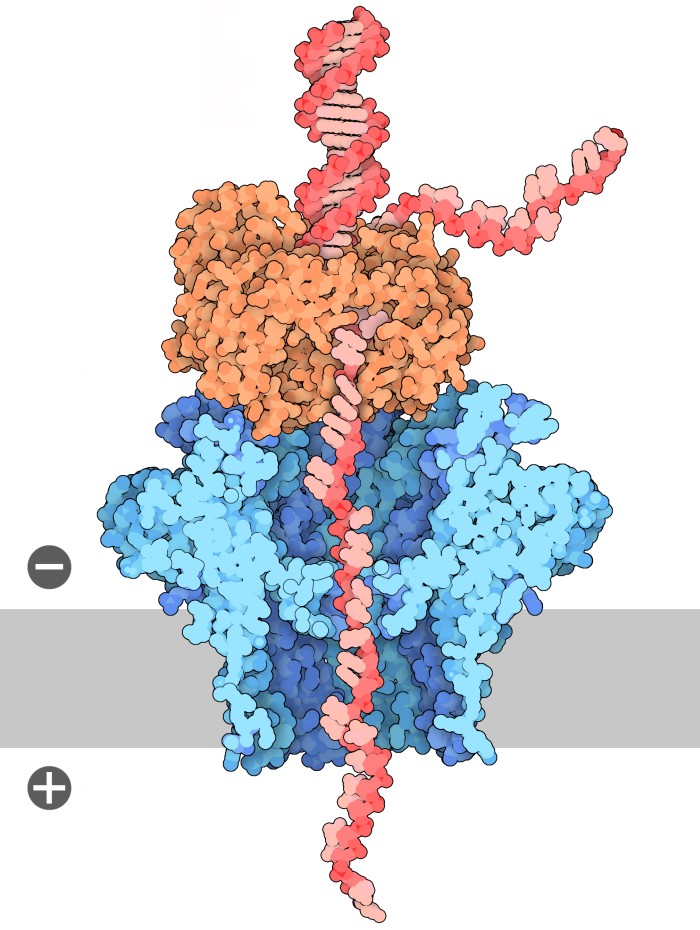 Introduzione IntroduzioneIl sequenziamento del DNA ha rivoluzionato lo studio della biologia rivelando le informazioni genetiche in cui sono codificati i processi vitali. Il Progetto Genoma Umano, che ha richiesto 12 anni di lavoro ed è costato 3 miliardi di dollari, utilizzava la tecnica tradizionale di Sanger di sequenziamento per terminazione di catena. Da allora l'interesse per la genomica è continuato a crescere e ha spinto alla ricerca di nuove tecniche di sequenziamento più veloci, affidabili ed economiche. Il sequenziamento con nanopori è una tecnica che si è evoluta rapidamente negli ultimi tempi, soddisfa a tutte queste esigenze e richiede un apparato che costa meno di 1000 dollari. Oggi questa tecnica è usata ovunque, persino in Antartide e nella Stazione Spaziale Internazionale. Sequenziatori tascabili Il nanoporo di sequenziamento decodifica la catena di DNA man mano che questa attraversa il piccolo poro inserito in una membrana. Paragonata alla tecnica tradizionale di sequenziamento, questa riconosce direttamente i nucleotidi senza bisogno di sintetizzare o amplificare il DNA. Inoltre, questa tecnica può trattare lunghi frammenti di DNA e questo permette di ricostruire la sequenza di tutto il genoma di un organismo in modo rapido e affidabile. Un singolo apparato di sequenziamento contiene migliaia di nanopori, ma rimane semplice, economico e così piccolo da poter essere messo in tasca. I ricercatori lo stanno ancora migliorando per ridurre il numero di errori ed aumentare la lunghezza delle catene che si possono sequenziare. Nanopori in azione I nanopori di sequenziamento sono costituiti da un nanoporo inserito in una membrana che divide una soluzione salina in due camere. Ai due lati della membrana si applica un voltaggio che produce un flusso di ioni che può essere misurato. Quando un filamento di DNA è tirato attraverso il poro, il flusso di ioni si blocca parzialmente e così si riduce la corrente misurata. I quattro tipi di nucleotidi sono associati a quattro diversi livelli di corrente ionica e questo consente di identificarli. I sequenziatori oggi in commercio contengono nanopori formati con la proteina batterica CsgG modificata. La proteina CsgG, mostrata qui sopra in blu (file PDB 4uv3) è una proteina di membrana sempre aperta, non selettiva, con un poro di circa 1 nanometro, che consente il passaggio di un singolo filamento di DNA. Per migliorare la sua capacità di leggere il DNA, CsgG è stata modificata per ridisegnare la forma del nanoporo, come spiegato più avanti, e così si è migliorata la resa e l'accuratezza del sequenziamento. La proteina CsgG è mostrata anche qui sotto vista di fianco e da sopra. 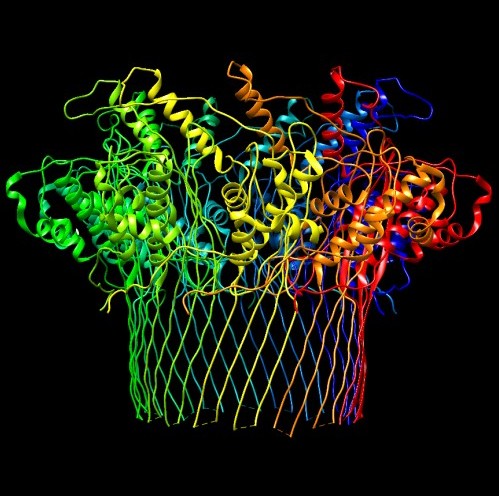 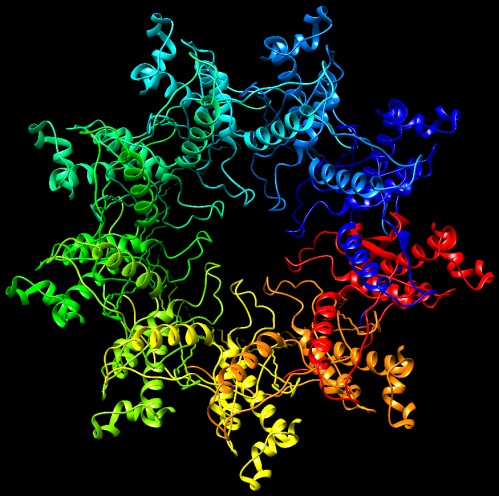
Regolare il passaggio del DNA Per avere un buon sequenziamento, è necessario regolare con precisione la velocità di scorrimento della catena di DNA attraverso il nanoporo. Le catene di DNA libere attraversano il nanoporo troppo velocemente e non ci permettono di leggere in modo chiaro le singole basi. I ricercatori hanno scoperto di poter regolare questa velocità aggiungendo al nanoporo enzimi che si legano al DNA come le polimerasi e le elicasi. Nella figura in alto, al nanoporo (blu) è stata aggiunta la DNA polimerasi del batteriofago phi29 (arancione, file PDB 1xhz). Questa funziona come un motore che fa avanzare un passo alla volta il DNA e così lo rallenta. Il DNA, guidato dalla DNA polimerasi, avanza a scatti attraverso il poro di CsgG, un nucleotide alla volta e così si possono identificare i singoli nucleotidi. 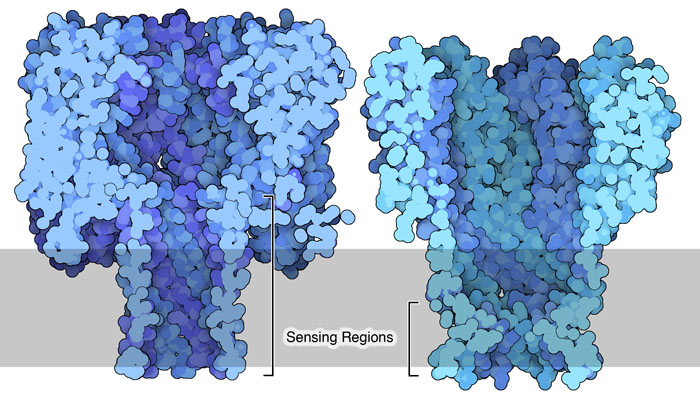 I
pori in natura I
pori in naturaIn natura vi sono molte proteine che formano pori. Le tossine batteriche che formano pori (PFT) sono state tra le prime ad essere considerate dai biologi strutturali perché vengono secrete come proteine solubili in acqua, e in seguito si legano alla membrana formando un complesso transmembrana a forma di poro che causa la morte della cellula aggredita. La tossina batterica alfa-emolisina di Stafilococco Aureo, mostrata qui a fianco sulla sinistra (file PDB 7ahl), è stata la prima proteina usata come nanoporo per il sequenziamento. Possiede un lungo canale con diametro variabile da 1,4 a 2,5 nanometri che però si è rivelato troppo lungo e anche un po' troppo largo per distinguere i singoli nucleotidi. La corrente ionica residua era influenzata da più nucleotidi per volta mentre questi attraversavano i vari punti di restringimento. Un'altra proteina che è stata usata per il sequenziamento è la proteina micobatterica MspA mostrata sulla destra (PDB ID 1uun), un nanoporo naturale con un breve restringimento, che costituisce la regione sensibile. Questo è un vantaggio perché la corrente ionica residua, in un poro con un restringimento breve, è influenzata solo da un nucleotide per volta e questo consente una lettura più accurata della sequenza. Dato che MspA ha un diametro del poro di soli 1,2 nanometri e una regione sensibile più breve, si è dimostrato un sequenziatore migliore dell'alfa-emolisina. Esplorando la struttura: Nanopori con due restringimenti 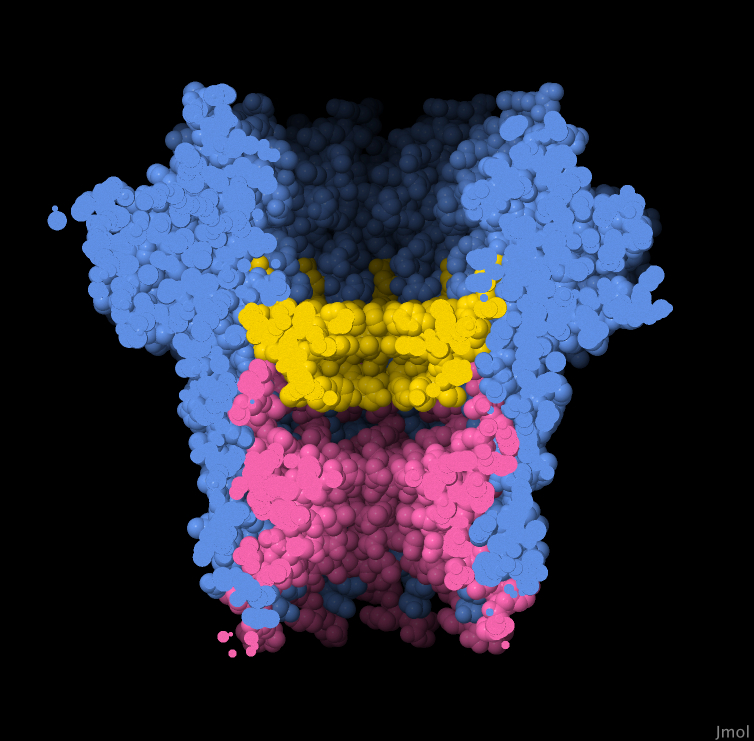 I
ricercatori sono riusciti a produrre nanopori con una maggiore stabilità
del poro e regolati per funzionare in modo ottimale. Per esempio, hanno
creato nanopori con due restringimenti aggiungendo una proteina accessoria
alla CsgG per introdurre un secondo restringimento e controllare meglio
lo scorrimento del DNA. Qui a fianco è mostrato il complesso CsgG-CsgF
(file PDB 6si7) con la proteina CsgG in
blu, la sua regione sensibile che forma il restringimento in giallo, e
la proteina CsgF in rosa. I
ricercatori sono riusciti a produrre nanopori con una maggiore stabilità
del poro e regolati per funzionare in modo ottimale. Per esempio, hanno
creato nanopori con due restringimenti aggiungendo una proteina accessoria
alla CsgG per introdurre un secondo restringimento e controllare meglio
lo scorrimento del DNA. Qui a fianco è mostrato il complesso CsgG-CsgF
(file PDB 6si7) con la proteina CsgG in
blu, la sua regione sensibile che forma il restringimento in giallo, e
la proteina CsgF in rosa.CsgG forma il primo punto di restringimento per il DNA e CsgF ne aggiunge un secondo. Combinando i due segnali che provengono dai due punti di restringimento, si aumenta l'accuratezza della lettura del nucleotide, soprattutto in tratti del DNA in cui vi sono molte copie consecutive della stesso nucleotide. Spunti per ulteriori esplorazioni Per esplorare la struttura delle quattro basi azotate del DNA, cercate il file PDB 2rpd che contiene un singolo filamento di DNA con le quattro basi in sequenza. La Aerolisina, un altro nanoporo attualmente usato per il sequenziamento, è stata ottenuta da un batterio. La forma naturale di questo poro la trovate nel file PDB 5jzt. 6si7: Van der Verren, S.E., Van Gerven, N., Jonckheere, W., Hambley, R., Singh, P., Kilgour, J., Jordan, M., Wallace, E.J., Jayasinghe, L., Remaut, H. (2020) A dual-constriction biological nanopore resolves homonucleotide sequences with high fidelity. Nat Biotechnol 38, 1415-1420. Carter, J.M., Hussain, S. (2017) Robust long-read native DNA sequencing using the ONT CsgG Nanopore system. Wellcome Open Res, 2, 23. Castro-Wallace, S.L., Chiu, C.Y., John, K.K., Stahl, S.E., Rubins, K.H., McIntyre, A.B.R., Dworkin, J.P., Lupisella, M.L., Smith, D.J., Botkin, D.J., Stephenson, T.A., Juul, S., Turner, D.J., Izquierdo, F., Federman, S., Stryke, D., Somasekar, S., Alexander, N., Yu, G., Mason, C.E., Burton, A.S. (2017) Nanopore DNA sequencing and genome assembly on the International Space Station. Sci Rep 7, 18022. Johnson, S.S., Zaikova, E., Goerlitz, D.S., Bai, Y., Tighe, S.W. (2017) Real-time DNA sequencing in the Antarctic dry valleys using the Oxford Nanopore sequencer. J Biomol Tech 28, 2-7. Deamer, D., Akeson, M., Branton, D. (2016) Three decades of nanopore sequencing. Nat Biotechnol 34, 518-524. 4uv3: Goyal, P., Krasteva, P.V., Van Gerven, N., Gubellini, F., Van den Broeck, I., Troupiotis-Tsailaki, A., Jonckheere, W., Pehau-Arnaudet, G., Pinkner, J.S., Chapman, M.R., Hultgren, S.J., Howorka, S., Fronzes, R., Remaut, H. (2014) Structural and mechanistic insights into the bacterial amyloid secretion channel CsgG. Nature 516, 250-253. Lieberman, K.R., Cherf, G.M., Doody, M.J., Olasagasti, F., Kolodji, Y., Akeson, M. (2010) Processive replication of single DNA molecules in a nanopore catalyzed by phi29 DNA polymerase. J Am Chem Soc 132, 17961-17972. 1uun: Faller, M., Niederweis, M., Schulz, G.E. (2004) The structure of mycobacterial outer-membrane channel. Science 303, 1189-1192. 1xhz: Kamtekar, S., Berman, A.J., Wang, J., Lazaro, J.M., de Vega, M., Blanco, L., Salas, M., Steitz, T.A. (2004) Insights into strand displacement and processivity from the crystal structure of the protein-primed DNA polymerase of bacteriophage phi29. Mol Cell 16, 609-618. 7ahl: Song, L., Hobaugh, M.R., Shustak, C., Cheley, S., Bayley, H., Gouaux, J.E. (1996) Structure of staphylococcal alpha-hemolysin, a heptameric transmembrane pore. Science 274, 1859-1866.
|
||||||
|
|
||||||
