|
Molecola del mese di dicembre 2019
HIF-alfa è un interruttore molecolare che risponde alle variazioni
dei livelli di ossigeno
 Introduzione Introduzione
Per noi l'ossigeno è essenziale, senza di esso le nostre cellule
morirebbero rapidamente. Proprio per questo, abbiamo sviluppato un sistema
dedicato che controlla il livello di ossigeno e induce le opportune
risposte quando questo si abbassa troppo cioè in caso di ipossia.
Le cellule in carenza di ossigeno mandano un segnale che induce la produzione
di globuli rossi e vasi sanguigni. Inoltre le cellule in carenza di
ossigeno riprogrammano il loro metabolismo spostando la produzione di
energia verso vie diverse che non richiedano ossigeno. Riducono quindi
l'attività della piruvato deidrogenasi che porta alla respirazione
cellulare e aumentano l'attività della lattato deidrogenasi e
così realizzano la fermentazione lattica. Il premio Nobel per
la medicina e la fisiologia di quest'anno è stato attribuito
a tre ricercatori che hanno scoperto i dettagli di questo processo sensibile
all'ossigeno chiarendo il ruolo della sua molecola centrale, il fattore
di trascrizione HIF (fattore inducibile da ipossia).
Alti livelli di ossigeno
Il fattore di trascrizione alfa inducibile da ipossia (HIF-alfa) è
l'interruttore centrale che permette alle cellule di rispondere alla
carenza di ossigeno. E' una proteina di circa 800 amminoacidi con parti
che svolgono diverse funzioni.
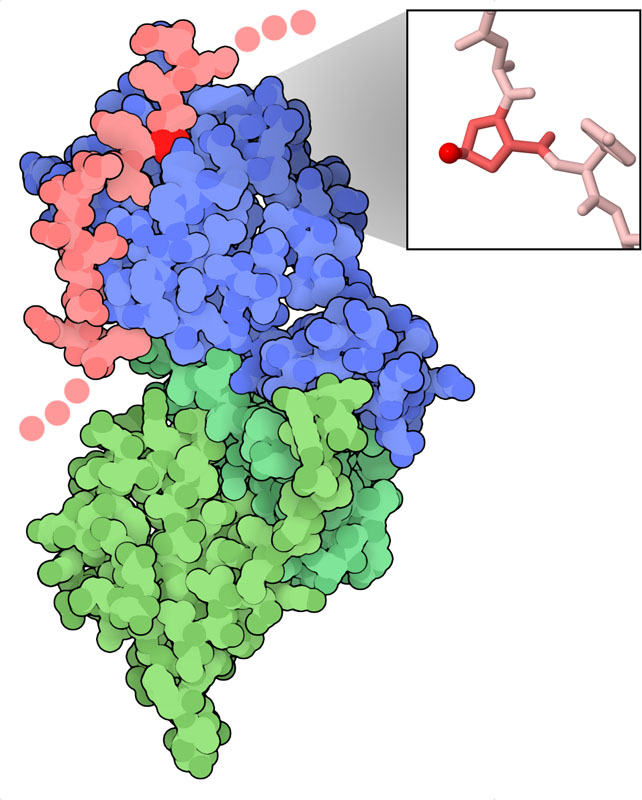
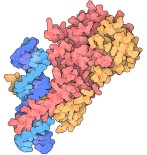
La struttura mostrata qui a fianco (file PDB 1lqb
e 1lm8) include una piccola porzione della
regione centrale di HIF-alfa (rosa) che contiene due proline chiave
(una è evidenziata in rosso). Quando l'ossigeno è abbondante,
queste proline sono idrossilate da enzimi PHD (HIF prolil idrossilasi)
e quindi contengono l'ossigeno mostrato in figura come sferetta rossa.
La idrossiprolina viene poi riconosciuta da pVHL (blu, von Hippel-Lindau
soppressore di tumore) in complesso con elonghina B e C (verde). Questo
complesso lega ubiquitina ad HIF-alfa per degradarlo nei proteasomi.
Quindi, se i livelli di ossigeno sono normali, HIF-alfa è continuamente
degradata e le cellule si comportano in modo normale.
Sentire l'ossigeno
Gli enzimi PHD hanno il compito di sentire i livelli di ossigeno. Inseriscono
un atomo di ossigeno in due proline chiave in HIF-alfa usando uno ione
metallico e acido alfachetoglutarico. Quando l'ossigeno è scarso,
la reazione di PHD è rallentata e le proline non vengono più
idrossilate. Un altro enzima, FIH (fattore di inibizione di HIF), realizza
un altro tipo di idrossilazione su una asparagina in HIF-alfa e modifica
il modo con cui questo interagisce con gli enzimi della trascrizione
(file PDB 1h2n, non mostrato qui).
Bassi livelli di ossigeno
Quando vi è poco ossigeno, HIF-alfa non viene idrossilato,
non è degradato dai proteasomi e così entra in azione.
Va nel nucleo si associa con una proteina simile, chiamata HIF-beta.
Insieme si legano a molti siti del DNA e promuovono la trascrizione
di geni coinvolti nel metabolismo in carenza di ossigeno e nella rimodellazione
del sistema circolatorio per migliorare il trasporto di ossigeno. Questo
promuove la crescita di molti tumori, per questo HIF-alfa è diventato
un bersaglio nella terapia del cancro.
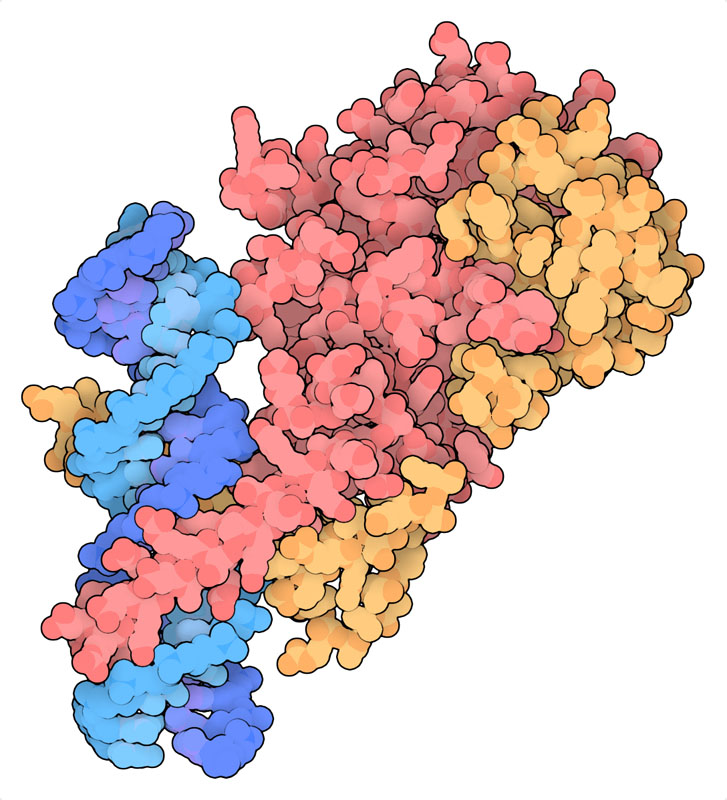
La struttura illustrata qui a fianco (file PDB 4zpr)
ci mostra come questo complesso si lega al DNA (azzurro e blu) usando
due tratti di alfa elica uno di
HIF-alfa (rosa) e l'altro di HIF-beta (gialla). Questi interagiscono
con la scanalatura maggiore del DNA avvolgendolo a forbice.
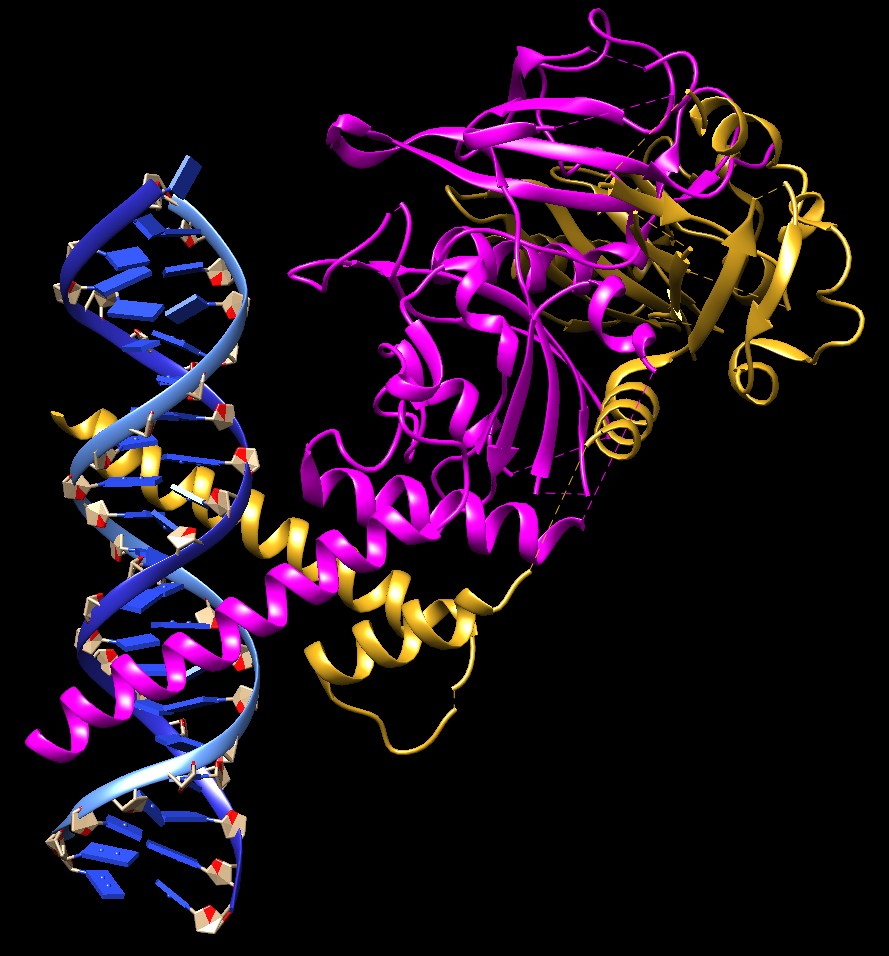
Questo dettaglio si vede bene anche nell'immagine qui sotto realizzata
con Chimera.
. . . . . . .
Esplorando la struttura
Recentemente, per il trattamento dell'anemia e dell'ischemia,
si stanno studiando farmaci capaci di legarsi agli enzimi PHD. Bloccando
questi enzimi, il farmaco induce le cellule a credere di aver bisogno
di più ossigeno e così le induce a mandare segnali per
produrre una quantità maggiore di globuli rossi. Quando i ricercatori
hanno ottenuto le prime strutture 3D dell'enzima PHD2, hanno cominciato
subito a progettare (con la tecnica del docking molecolare) degli inibitori
di questo enzima come i due che sono presenti nei file PDB 2g1m
e 2g19 (non mostrati qui).
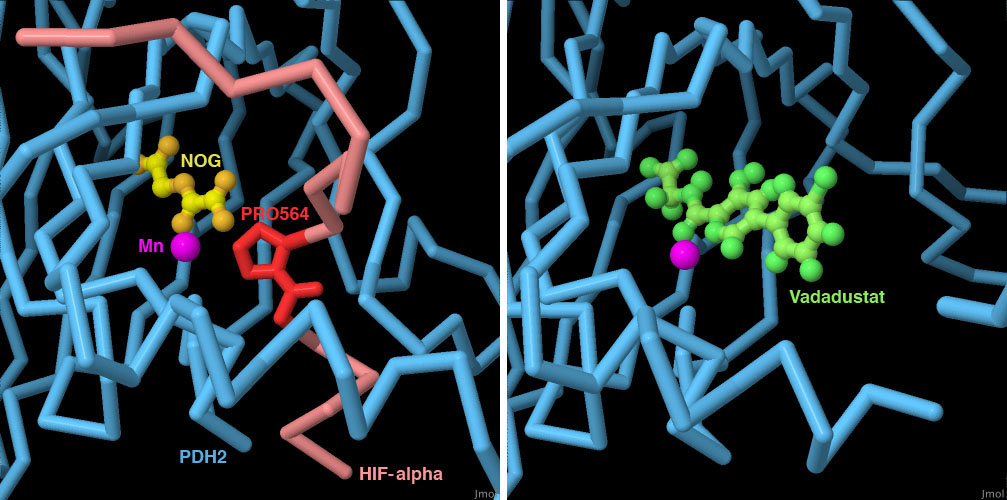
Qui sotto a destra è mostrato uno dei più promettenti
inibitori vadadustat (file PDB 5ox6)
che ha superato le prime fasi di test in vitro, ma che è ancora
un farmaco in valutazione. Confrontando questo farmaco (verde) con la
struttura qui sotto a sinistra di PHD2 legata ad un piccolo frammento
di HIF-alfa (rosa) (file PDB 3hqr), possiamo
vedere che il nuovo farmaco si lega all'enzima nello stesso punto dell'acido
alfachetoglutarico (qui è presente un suo analogo NOG, giallo)
ed inoltre è abbastanza grande da bloccare il legame con la prolina-564
(rossa) di HIF-alfa.
Spunti per ulteriori esplorazioni
Vi sono molte strutture di FIH negli archivi PDB che vi permettono di
esplorare le sue interazioni con altri substrati e inibitori
Il dominio del complesso FIH che lega il DNA è simile ad una
forbice e viene chiamato "basic helix-loop-helix". Potete
trovarne altri esempi cercando "bHLH" nel sito PDB.
 Bibliografia Bibliografia
5ox6: Yeh, T.L., Leissing, T.M., Abboud, M.I., Thinnes, C.C.,
Atasoylu, O., Holt-Martyn, J.P., Zhang, D., Tumber, A., Lippl, K., Lohans,
C.T., Leung, I.K.H., Morcrette, H., Clifton, I.J., Claridge, T.D.W.,
Kawamura, A., Flashman, E., Lu, X., Ratcliffe, P.J., Chowdhury, R.,
Pugh, C.W., Schofield, C.J. (2017) Molecular and cellular mechanisms
of HIF prolyl hydroxylase inhibitors in clinical trials. Chem Sci 8:
7651-7668.
4zpr: Wu, D., Potluri, N., Lu, J., Kim, Y., Rastinejad, F. (2015)
Structural integration in hypoxia-inducible factors. Nature 524: 303-308.
Semenza, G..L (2012) Hypoxia-inducible factors in physiology
and medicine. Cell 148: 399-408.
3hqr: Chowdhury, R., McDonough, M.A., Mecinovic, J., Loenarz,
C., Flashman, E., Hewitson, K.S., Domene, C., Schofield, C.J. (2009)
Structural basis for binding of hypoxia-inducible factor to the oxygen-sensing
prolyl hydroxylases. Structure 17: 981-989.
2g1m, 2g19: McDonough, M.A., Li, V., Flashman, E., Chowdhury,
R., Mohr, C., Lienard, B.M.R., Zondlo, J., Oldham, N.J., Clifton, I.J.,
Lewis, J., McNeill, L.A., Kurzeja, R.J.M., Hewitson, K.S., Yang, E.,
Jordan, S., Syed, R.S., Schofield, C.J. (2006) Cellular oxygen sensing:
crystal structure of hypoxia-inducible factor prolyl hydroxylase (PHD2).
Proc.Natl.Acad.Sci.USA 103: 9814-9819.
1h2n: Elkins, J.M., Hewitson, K.S., McNeill, L.A., Seibel, J.F.,
Schlemminger, I., Pugh, C., Ratcliffe, P., Schofield, C.J. (2003) Structure
of factor-inhibiting hypoxia-inducible factor (Hif) reveals mechanism
of oxidative modification of Hif-1Alpha. J.Biol.Chem. 278: 1802-1806.
1lqb: Hon, W.C., Wilson, M.I., Harlos, K., Claridge, T.D., Schofield,
C.J., Pugh, C.W., Maxwell, P.H., Ratcliffe, P.J., Stuart, D.I., Jones,
E.Y. (2002) Structural basis for the recognition of hydroxyproline in
HIF-1 alpha by pVHL. Nature 417: 975-978.
1lm8: Min, J.H., Yang, H., Ivan, M., Gertler, F., Kaelin Jr.,
W.G., Pavletich, N.P. (2002) Structure of an HIF-1alpha -pVHL complex:
hydroxyproline recognition in signaling. Science 296: 1886-1889
|
|
|