|
|
Ribonucleotide
riduttasi
|
|||||
|
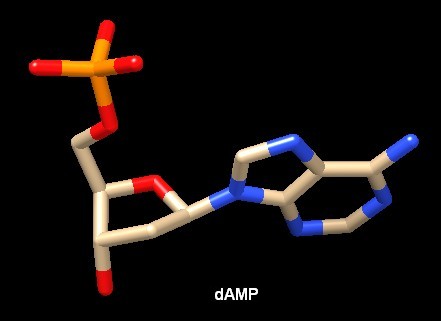
Molecola del mese di ottobre 2019 La ribonucleotide riduttasi crea i desossiribonucleotidi, i mattoni fondamentali per la sintesi e la riparazione del DNA 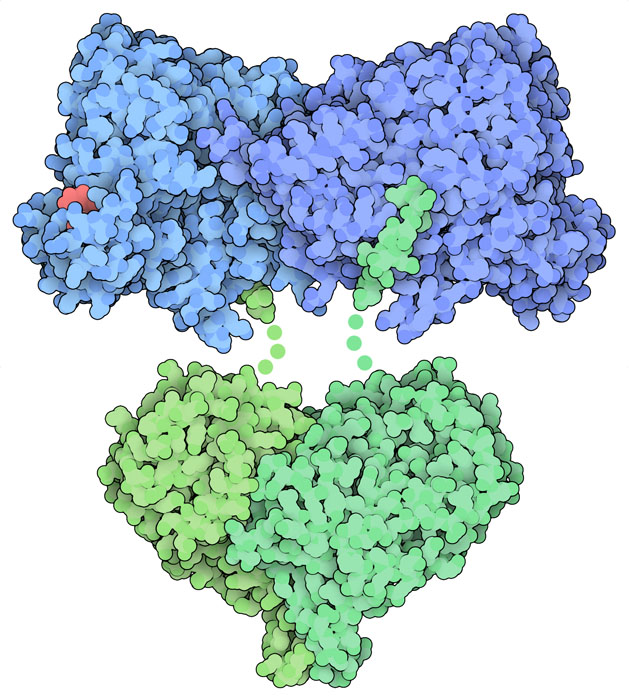 Introduzione IntroduzioneDNA ed RNA hanno strutture quasi identiche che si differenziano per un solo gruppo OH. Il DNA non possiede un OH sul ribosio nella posizione 3', come ci ricorda il suo nome: Acido Desossiribo Nucleico. Questa piccola differenza, però, ha una grande influenza sul comportamento chimico del DNA e lo rende molto più stabile e adatto a conservare l'informazione genetica per lungo tempo. D'altra parte l'RNA è meno stabile perchè l'OH in più sul ribosio lo rende più suscettibile all'idrolisi e così l'RNA viene usato spesso dalla cellula come un flash drive biologico, un supporto dove conservare dati biologici per un breve periodo di tempo per poi cancellarli quando non servono più. Qui sotto sono mostrati un ribonucleotide (AMP) e un desossiribonucleotide (dAMP), in quest'ultimo si nota la mancanza del gruppo OH in basso sull'anello. Nel nostro corpo, il solo modo per sintetizzare i desossiribonucleotidi, essenziali per la sintesi e la riparazione del DNA, è quello di ridurre il corrispondente ribonucleotide per opera dell'enzima ribonucleotide riduttasi. . . . . 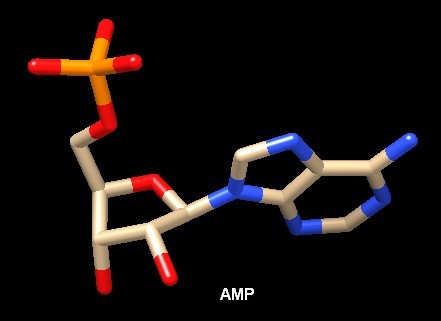 . . . .
. . . . 
Un enzima radicale Il meccanismo catalitico della ribonucleotide riduttasi è interessante perché coinvolge dei radicali liberi. Mentre, in generale, i radicali liberi sono dannosi per il nostro corpo, nella ribonucleotide riduttasi questi giocano un ruolo essenziale nella sintesi dei blocchi costruttivi del DNA. La ribonucleotide riduttasi è un enzima altamente conservato in tutte le forma di vita, ma vi sono più classi di questo enzima che usano metalli diversi per generare i radicali liberi. Nell'enzima ribonucleotide riduttasi di Classe I di Escherichia coli, mostrato in alto, un radicale libero è prodotto nella subunità beta (verde, chiamata anche R2) e da qui migra verso la subunità alfa (blu, R1) dove viene sintetizzato il deossiribonucleotide. Questo enzima è un tetramero formato da due subunità alfa (blu chiaro e scuro, file PDB 1mrr) e due subunità beta (verde chiaro e scuro, file PDB 3r1r) collegate da una catena flessibile (mostrata con puntini). Un nucleotide (rosso) è legato nel sito regolatorio della subunità alfa. Bersaglio per la chemioterapia Una delle vie per fermare la crescita incontrollata delle cellule cancerose consiste nel fermare gli enzimi coinvolti nella sintesi del DNA. Per questo, la ribonucleotide riduttasi è diventata un importante bersaglio per i farmaci contro il cancro. Il metodo più ovvio per inibire questo enzima è quello di creare una molecola che somigli ad un nucleoside e si leghi all'enzima impedendo ai normali nucleosidi difosfato di legarsi nel sito attivo. Dato, però, che gli analoghi dei nucleosidi sono molto simili ai normali nucleosidi, potrebbero essere incorporati nel DNA delle cellule sane oltre che in quello delle cellule cancerose provocando dei seri effetti collaterali nei pazienti. Una via per risolvere questo problema consiste nell'usare piccole molecole che non siano analoghi dei nucleosidi, ma che si leghino comunque nel sito attivo bloccando l'attività della ribonucleotide riduttasi. Più sotto, nel paragrafo Esplorando la struttura, sono mostrati esempi di entrambi questi approcci. 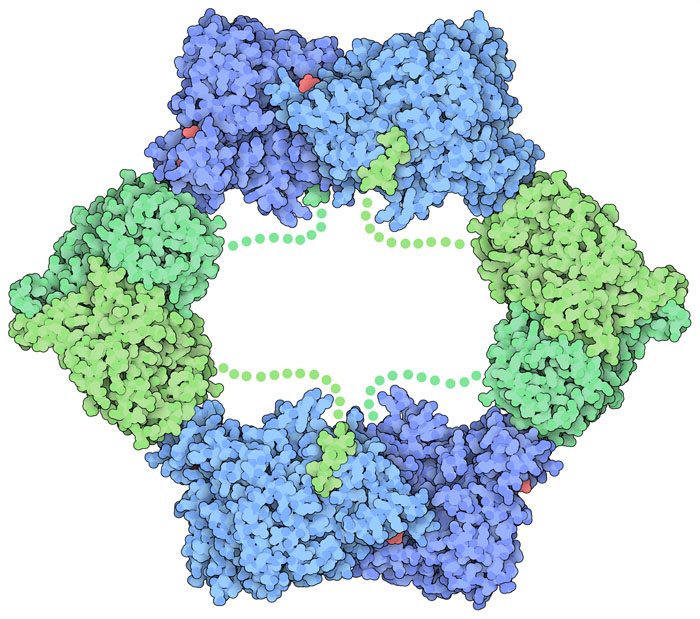 Controlli e bilanciamenti Dato che la ribonucleotide riduttasi è essenziale per la sintesi del DNA, la sua azione è altamente regolata per consentirle di lavorare solo quando è necessario. Quando la concentrazione di ATP nella cellula è alta, l'ATP si lega al sito di controllo della ribonucleotide riduttasi come segnale per sintetizzare più desossiribonucleotidi. Questo accade, per esempio, durante la divisione cellulare, quando si deve sintetizzare molto nuovo DNA. Accanto a questo sito di controllo, la ribonucleotide riduttasi ne ha un altro che sente quali desossiribonucleotidi sono necessari. Per impedire che la ribonucleotide riduttasi sintetizzi livelli troppo alti e tossici di desossiribonucleotidi, il desossiATP, dATP, si lega all'enzima e ne spegne l'azione. Qui a lato è mostrata la forma inibita dell'enzima (file PDB 3uus). Il modo in cui l'enzima si disattiva è particolarmente interessante: due tetrameri dell'enzima si legano tra loro formando una struttura ad anello che impedisce il passaggio dei radicali liberi dalla subunità beta alla alfa. Esplorando la struttura 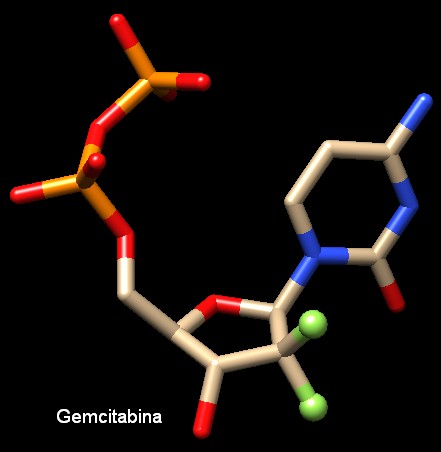 Di
seguito sono mostrate due strutture di ribonucleotide riduttasi inibite
da due diversi farmaci sintetici. Di
seguito sono mostrate due strutture di ribonucleotide riduttasi inibite
da due diversi farmaci sintetici. Il primo farmaco è gemcitabina difosfato, un analogo della citosina usato in chemioterapia contro vari tipi di cancro, che contiene due atomi di fluoro (verde chiaro) nella posizione 3' sul ribosio. Nella figura qui sotto (file PDB 2eud) l'enzima ribonucleotide riduttasi sta legando gemcitabina difosfato nel suo sito catalitico. Il farmaco inibisce l'enzima perchè non può essere ridotto dato che contiene due atomi di fluoro (verde chiaro) nella posizione 3' dove i normali nucleosidi hanno un idrogeno e un gruppo OH. L'uso terapeutico della gemcitabina, però, non è molto ampio a causa della sua tossicità perchè, tra l'altro, provoca terminazioni di catena nella sintesi del DNA. . . 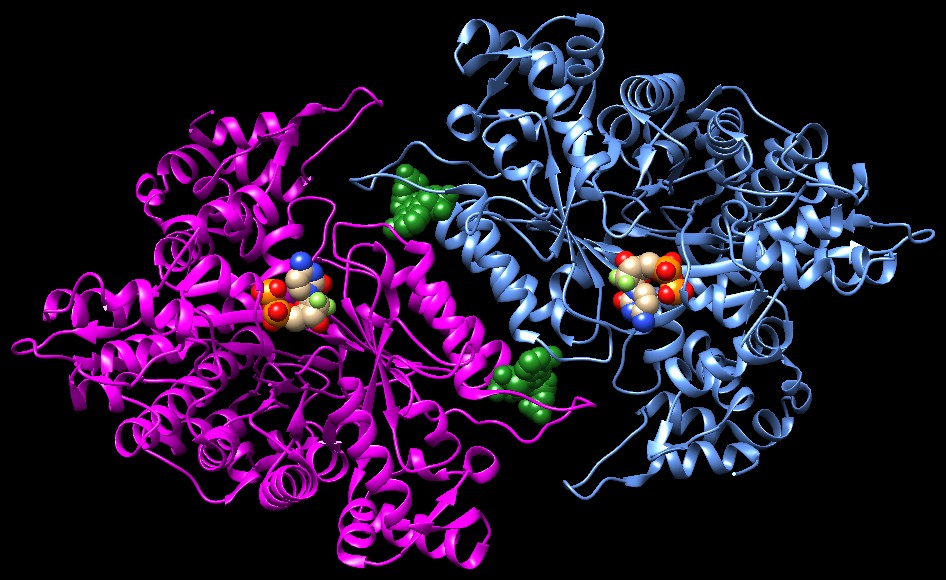 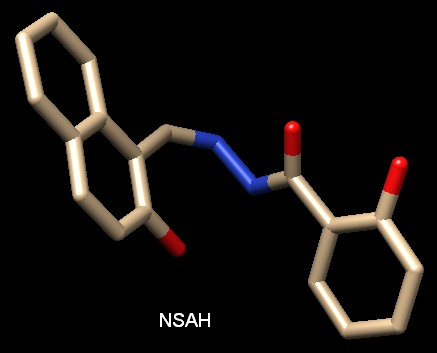 Il
secondo farmaco è Naftil salicil acil idrazone (NSAH),
un inibitore non nucleotidico scoperto attraverso tecniche di docking
molecolare, che permettono di progettare un farmaco valutando, al
computer, la sua interazione con il sito attivo dell'enzima, usando modelli
molecolari ad alta risoluzione del farmaco e dell'enzima. Il
secondo farmaco è Naftil salicil acil idrazone (NSAH),
un inibitore non nucleotidico scoperto attraverso tecniche di docking
molecolare, che permettono di progettare un farmaco valutando, al
computer, la sua interazione con il sito attivo dell'enzima, usando modelli
molecolari ad alta risoluzione del farmaco e dell'enzima.Nella figura qui sotto (file PDB 5tus) si vede che anche NSAH inibisce l'enzima legandosi nel sito catalitico. Questo farmaco, però, si è dimostrato due volte più efficace della gemcitabina nell'inibire la crescita delle cellule tumorali e nello stesso tempo ha mostrato una bassa tossicità verso le altre cellule. La sua azione fa diminuire in modo sostanziale la produzione di dGTP e dATP. . . . 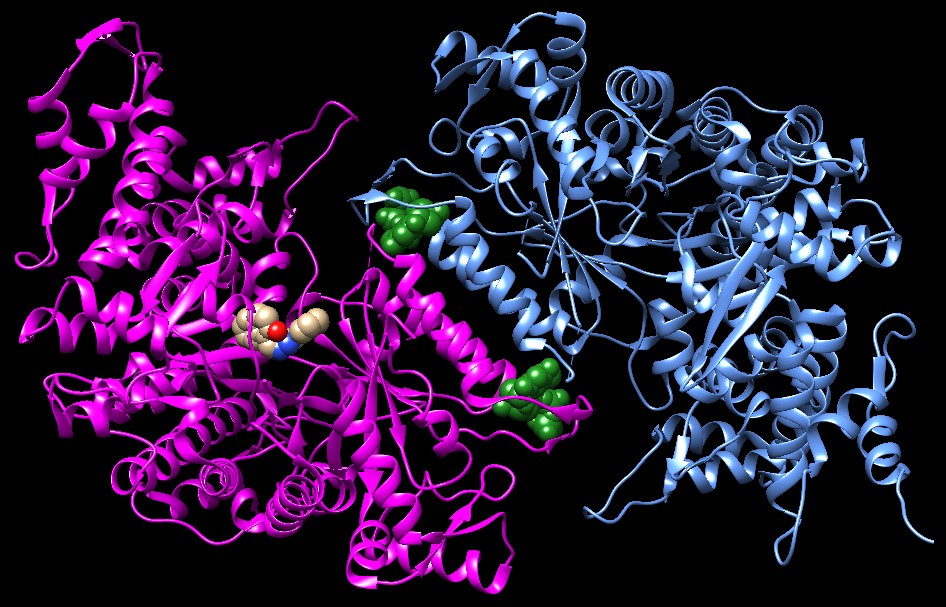 Spunti per ulteriori esplorazioni Molte proteine sono un bersaglio per la chemioterapia del cancro. Cercate nel sito PDB-101 nel menù Category la parola cancer (cancro). Negli archivi PDB vi sono molte molecole di DNA e RNA. Cercate su ogni ribosio di RNA il gruppo OH che fa la differenza con il DNA. Potete cominciare dal file PDB 1g4q che include una corta doppia elica ibrida tra DNA e RNA. 5tus: Ahmad, M.F., Alam, I., Huff, S.E., Pink, J., Flanagan, S.A., Shewach, D., Misko, T.A., Oleinick, N.L., Harte, W.E., Viswanathan, R., Harris, M.E., Dealwis, C.G. (2017) Potent competitive inhibition of human ribonucleotide reductase by a nonnucleoside small molecule. Proc. Natl. Acad. Sci. U.S.A. 114: 8241-8246 3uus: Ando, N., Brignole, E.J., Zimanyi, C.M., Funk, M.A., Yokoyama, K., Asturias, F.J., Stubbe, J., Drennan, C.L. (2011) Structural interconversions modulate activity of Escherichia coli ribonucleotide reductase. Proc.Natl.Acad.Sci.USA 108: 21046-21051 2eud: Xu, H., Faber, C., Uchiki, T., Racca, J., Dealwis, C. (2006) Structures of eukaryotic ribonucleotide reductase I define gemcitabine diphosphate binding and subunit assembly. Proc.Natl.Acad.Sci.Usa 103: 4028-4033 3r1r: Eriksson, M., Uhlin, U., Ramaswamy, S., Ekberg, M., Regnstrom, K., Sjoberg, B.M., Eklund, H. (1997) Binding of allosteric effectors to ribonucleotide reductase protein R1: reduction of active-site cysteines promotes substrate binding. Structure 5: 1077-1092 1mrr: Atta, M., Nordlund, P., Aberg, A., Eklund, H., Fontecave, M. (1992) Substitution of manganese for iron in ribonucleotide reductase from Escherichia coli. Spectroscopic and crystallographic characterization. J.Biol.Chem. 267: 20682-20688
|
||||||
|
|
||||||
