|
|
Antibiotici amminoglicosidici |
||||||
|
Molecola del Mese di Ottobre 2018 I batteri diventano resistenti agli antibiotici amminoglicosidici sia distruggendoli che modificando il loro bersaglio 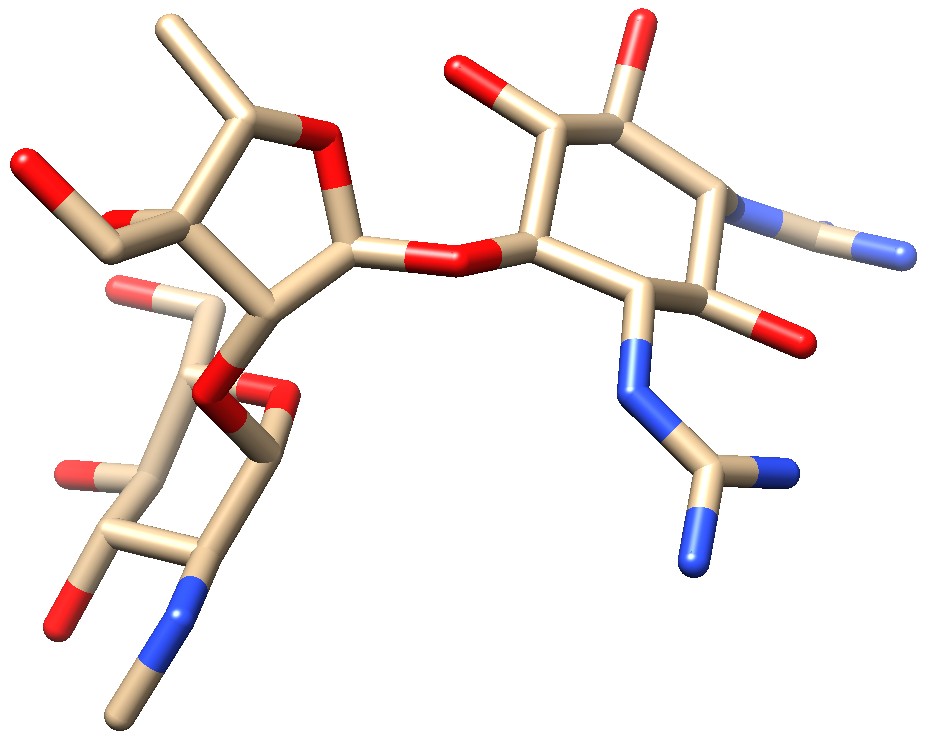 Introduzione IntroduzioneGli scienziati sono dei principianti nella lotta contro i batteri mentre gli organismi viventi hanno combattuto le infezioni per milioni di anni. Per questo la comunità scientifica ha potuto scoprire nuovi antibiotici semplicemente cercandoli in natura. Negli anni 1940, nei batteri del terreno, fu scoperto un antibiotico amminoglicosidico, la streptomicina (mostrata qui a lato), che ci ha permesso di combattere con successo la tubercolosi. Nelle decadi successive, però, gli scienziati sono stati costretti a sintetizzare nuove versioni di questo antibiotico come la neomicina e la paromomicina per combattere i batteri patogeni che rapidamente erano diventati resistenti prima alla streptomicina e poi ad altre sue varianti. 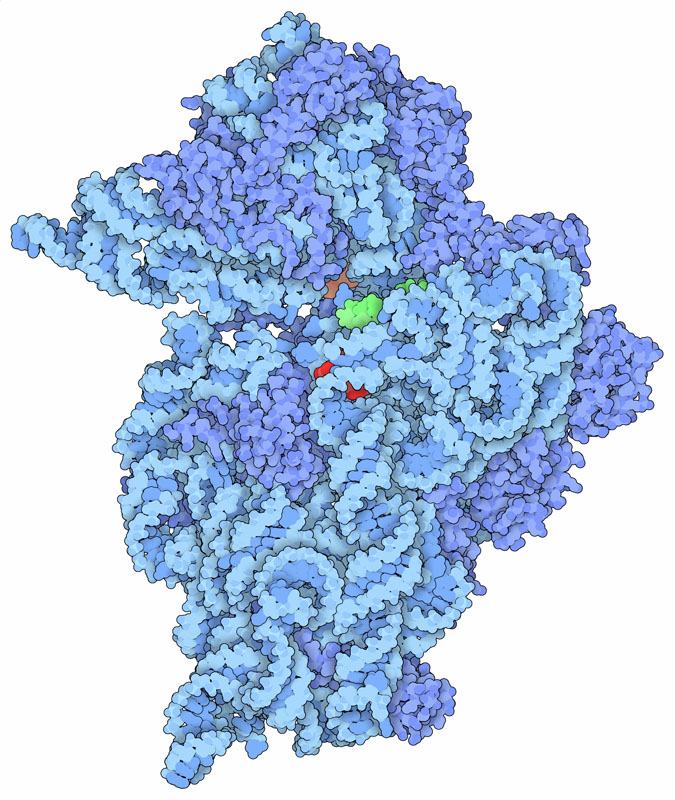 Vedere
il sito Vedere
il sitoStudiando la struttura del ribosoma (mdm 1-2010) batterico, nella subunità più piccola 30S, i ricercatori hanno individuato il sito dove si legano gli antibiotici amminoglicosidici. La struttura mostrata qui a fianco (file PDB 1fjg) è la subunità ribosomiale 30S di thermus thermophilus (composta da RNA, azzurro, e proteine, blu) che lega una piccola porzione di mRNA (verde) e tre diversi antibiotici. La paromomicina (rossa) è legata ad una lunga doppia elica di RNA che è coinvolta nel verificare che il transfer RNA (mdm 3-2001) sia legato correttamente all'RNA messaggero, un processo chiamato decodifica. La streptomicina (non visibile) è legata subito dietro in un sito confinante. La spectinomicina (arancione) è legata in un sito diverso ed interviene in una fase successiva della sintesi delle proteine, quando il fattore di allungamento G (mdm 9-2006) muove in avanti l'mRNA per esporre il codone successivo, un processo chiamato traslocazione. 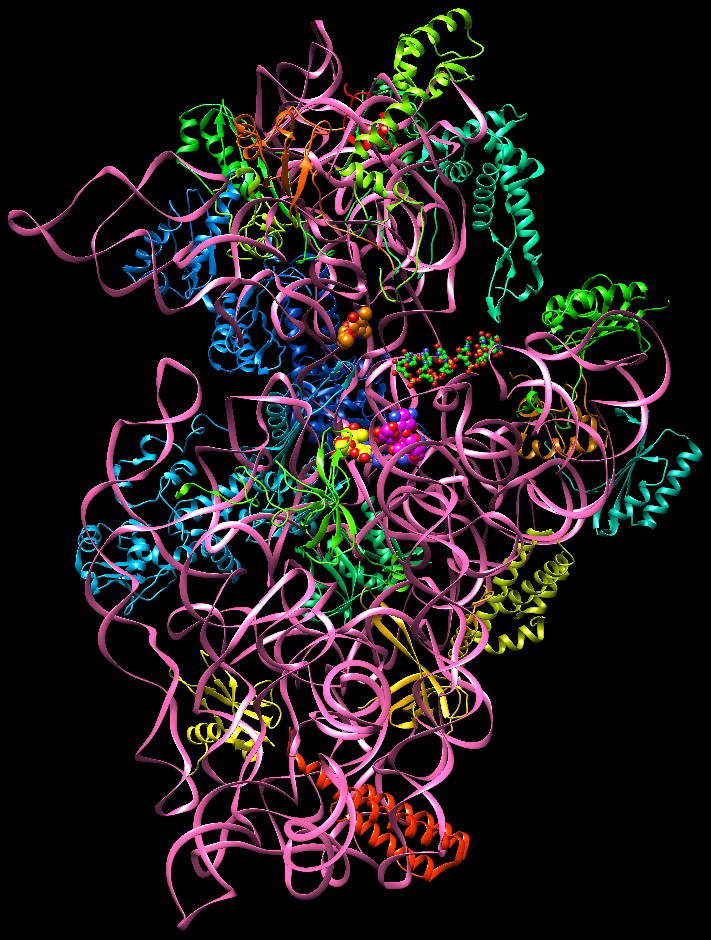 Nella
figura qui a fianco (realizzata con Chimera), la catena di RNA del ribosoma
30S è rappresentata con un nastro rosa, mentre le proteine ribosomiali
sono rappresentate con colori diversi. Nella
figura qui a fianco (realizzata con Chimera), la catena di RNA del ribosoma
30S è rappresentata con un nastro rosa, mentre le proteine ribosomiali
sono rappresentate con colori diversi. Nella struttura sono presenti tutti e tre gli antibiotici ammino-glicosidici e anche una piccola catena di mRNA. L'mRNA si trova al centro rappresentato con piccole sfere con carboni verdi. La paronomicina (sfere con carboni magenta) e la streptomicina (sfere con carboni gialli) si trovano in due siti vicini tre loro sotto l'mRNA del quale vogliono ostacolare la decodifica. La spectinomicina (sfere con carboni arancioni) si trova in un sito più in alto, sopra l'mRNA, dove ostacola la traslocazione. Qui sotto è mostrato un ingrandimento della porzione centrale della figura. 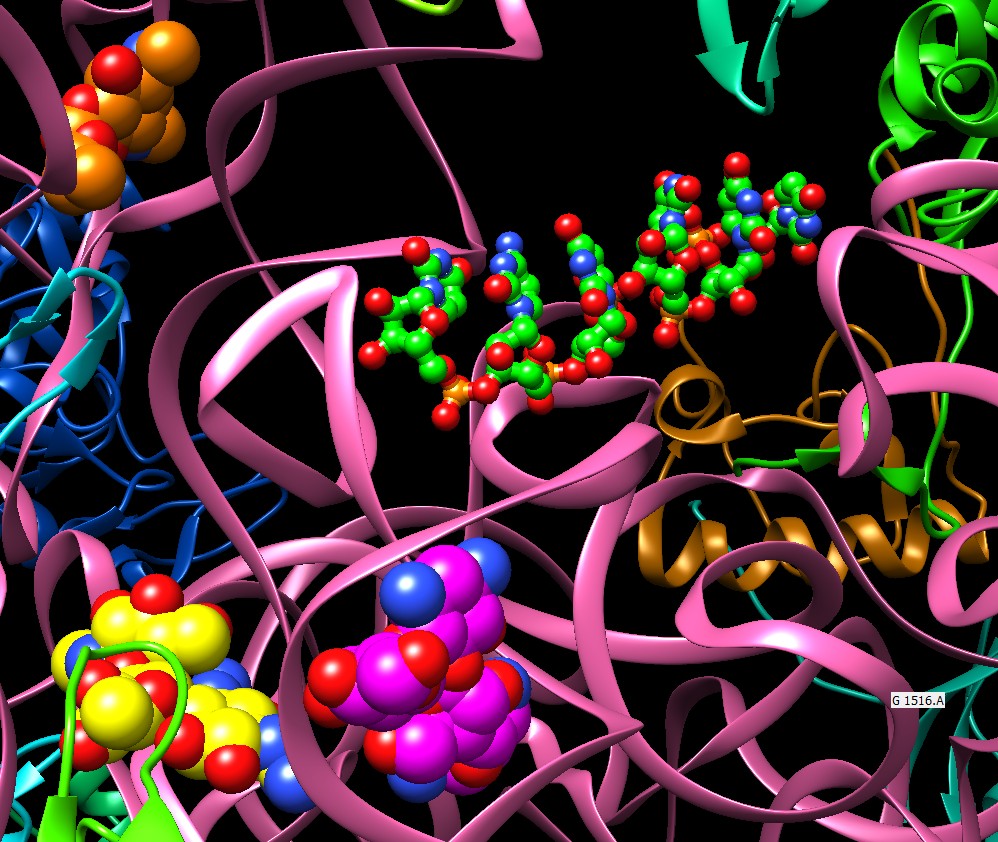 Errori forzati Gli antibiotici amminoglicosidici si legano ad una regione del ribosoma coinvolta nella traduzione del codice genetico, dove l'RNA transfer si appaia con il codone dell'RNA messaggero (decodifica). Gli antibiotici amminoglicosidici ostacolano i leggeri movimenti che sono necessari per garantire che si realizzino solo gli appaiamenti corretti, e quindi provocano appaiamenti errati che introducono amminoacidi diversi nella proteina sintetizzata. Queste proteine mutanti non sono in grado di funzionare correttamente e compromettono alcune funzioni vitali del batterio che alla fine muore. 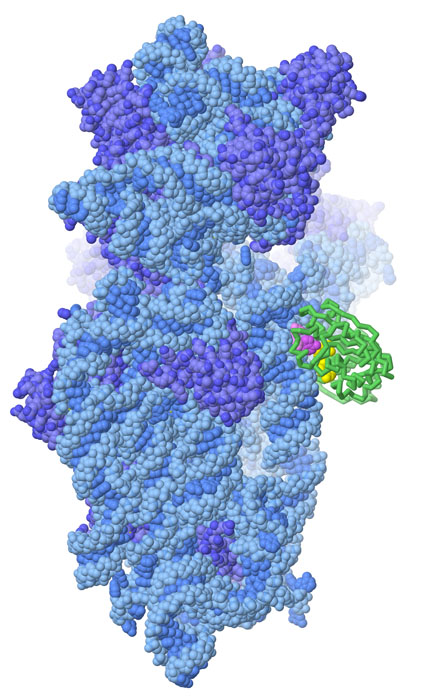 Proteggere
il sito Proteggere
il sitoCome accade anche con altri antibiotici, i batteri hanno sviluppato (e condiviso tra loro) molte strategie per diventare resistenti agli antibiotici amminoglicosidici. Una particolare adenina del ribosoma, A1408, è importante nel processo di riconoscimento codone-anticodone (decodifica) mRNA-tRNA ed è il bersaglio degli amminoglicosidi come la paromomicina e la streptomicina. I batteri possono diventare resistenti modificando questa adenina aggiungendole un gruppo metile e così la paromomicina non si può più legare, ma il ribosoma continua a funzionare correttamente nella sintesi proteica. La struttura qui a fianco (file PDB 4ox9) mostra come agisce uno specifico enzima metiltransferasi (verde): l'enzima piega in fuori l'adenina A1408 (magenta) e vi lega un metile trasferendolo da un cofattore donatore di metili (giallo). Esplorando la struttura Gli amminoglicosidi hanno molti gruppi amminici e idrossilici che sono indispensabili per legarsi all'RNA. Un'altra via con la quale i batteri diventano resistenti consiste nell'aggredire il farmaco stesso con un particolare enzima che modifica chimicamente gli amminoglicosidi aggiungendo altri gruppi chimici ai gruppi amminici o idrossilici in modo che il farmaco perda la sua capacità di legarsi all'RNA per indurre errori nella sintesi proteica. L'enzima mostrato qui sotto, usando ATP, aggiunge un nucleotide al farmaco, tobramicina, formando adenilil-tobramicina Le due strutture PDB 5cfs e 5cfu mostrano l'enzima prima e dopo la reazione. 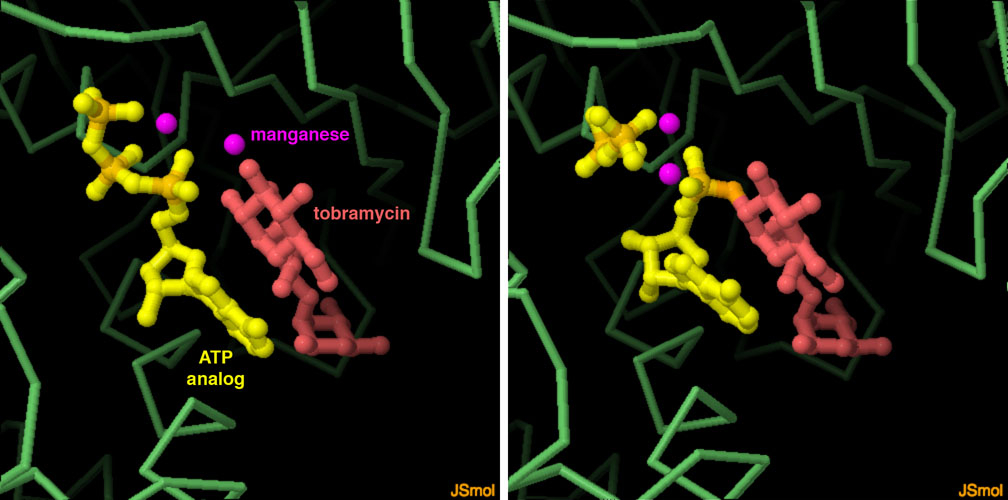 Spunti per ulteriori esplorazioni 1 - Per cercare nuovi amminoglicosidi, i chimici hanno spesso studiato la catena bersaglio sul ribosoma. Potete trovare alcune di queste strutture cercando "neomycin" o "paromomycin" nel sito RCSB PDB. 2 - Un'altra strategia usata dai batteri per diventare resistenti agli antibiotici è quella di pomparli fuori dalla loro cellula. Per vedere alcune di queste pompe cercate "efflux pump". 3 - Per vedere la struttura degli antibiotici amminoglicosidici (come quella mostrata all'inizio di questa pagina) cercate sulla pagina "ligand" di "streptomycin" o "paromomycin". 5cfs: Bassenden, A.V., Rodionov, D., Shi, K., Berghuis, A.M. (2016) Structural analysis of the tobramycin and gentamicin clinical resistome reveals limitations for next-generation aminoglycoside design. ACS Chemical Biology 11: 1339-1346 4ox9: Dunkle, J.A., Vinal, K., Desai, P.M., Zelinskaya, N., Savic, M., West, D.M., Conn, G.L., Dunham, C.M. (2014) Molecular recognition and modification of the 30S ribosome by the aminoglycoside-resistance methyltransferase NpmA. Proceedings of the National Academy of Science USA 111: 6275-6280 B Becker, MA Cooper (2013) Aminoglycoside antibiotics in the 21st century. ACS Chemical Biology 8, 105-115. 1fjg: Carter, A.P., Clemons Jr., W.M., Brodersen, D.E., Morgan-Warren, R.J., Wimberly, B.T., Ramakrishnan, V. (2000) Functional insights from the structure of the 30S ribosomal subunit and its interactions with antibiotics Nature 407: 340-348
|
|||||||
|
|
|||||||
