|
Molecola del Mese di Dicembre 2015
La vancomicina è un antibiotico glicopeptidico che impedisce la
costruzione della parete batterica di peptidoglicano
 Introduzione Introduzione
La maggior parte dei nostri antibiotici deriva da
batteri e funghi, i più grandi esperti al mondo nel combattere
le infezioni. Questi organismi si proteggono usando una incredibile
varietà di molecole esotiche che si sono evolute per attaccare
ogni punto debole dei loro competitori. La maggior parte di questi antibiotici,
come la penicillina
(mdm 5/2002), attaccano enzimi batterici, ne bloccano l'azione, e in
questo modo uccidono il batterio infettante. La vancomicina,
invece, attacca la molecola che deve essere elaborata dall'enzima batterico.
Fermare la sintesi
I batteri costruiscono una robusta parete cellulare
formata da catene di zuccheri intrecciate con piccoli peptidi che costituisce
una struttura a rete chiamata peptidoglicano. Questa rete è
costruita in più passaggi: all'inizio vengono formate catene
di zuccheri e poi queste vengono unite da piccole catene di peptidi.
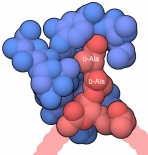
La vancomicina agisce nella seconda parte di questo processo. Un'estremità
del peptide di reticolazione termina con due amminoacidi strani, due
D-alanine che possiedono un centro stereochimico opposto (D, destro)
a quello dei normali amminoacidi che sono tutti L (sinistri). Per realizzare
la reticolazione, l'alanina terminale deve essere eliminata e così
il peptide viene legato al peptide vicino.
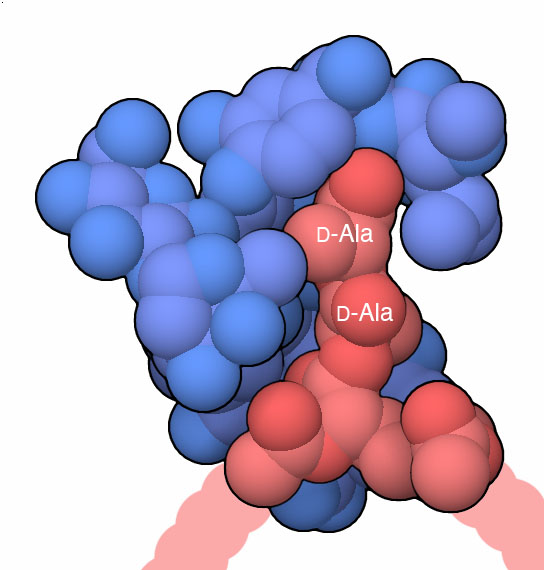
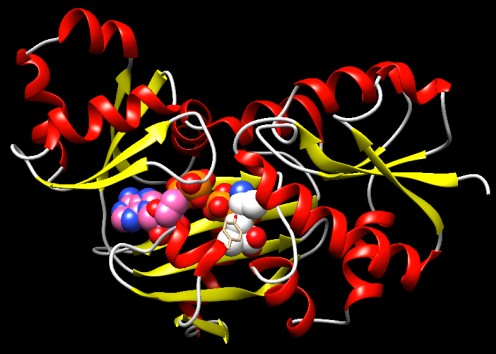
Come si può vedere nell'immagine qui a fianco (file PDB 1fvm),
la vancomicina si lega strettamente alla sequenza dei due amminoacidi
D-alanina-D-alanina, impedendo così al peptidoglicano di reticolarsi.
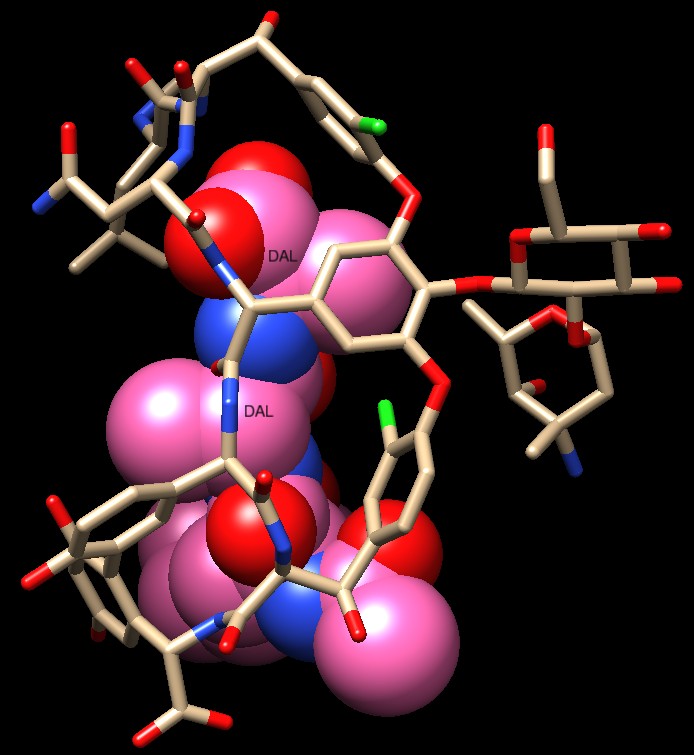
Nell'immagine qui sotto, realizzata con Chimera, la stessa struttura
è vista da dietro per evidenziare che la vancomicina è
un antibiotico glicopeptidico. Sulla sinistra è costituita
da una piccola sequenza di sette amminoacidi lievemente modificati che
formano alcuni anelli che rendono rigida la struttura e sono legati
ad uno zucchero (glucosio) sulla destra e ad un ultimo anello che termina
la molecola..
. . . . . . . . . . . . . . . . .
Gram positivi e gram negativi
Solo alcuni batteri sono sensibili alla vancomicina.
Batteri come lo Stafilococco hanno uno spesso strato di peptidoglicano
che circonda la cellula e sono chiamati gram-positivi per il
fatto che assorbono il primo dei coloranti di Gram e appaiono viola
al microscopio. La vancomicina è efficace nel bloccare la formazione
di questo tipo di parete cellulare e quindi viene usata come ultima
risorsa per curare le infezioni dei batteri gram-positivi resistenti
agli altri antibiotici.
I batteri gram-negativi come Escherichia coli, invece,
costruiscono il loro peptidoglicano al di sotto di una membrana protettiva
più esterna dove rimane inacessibile al colorante viola di Gram
e anche alla vancomicina. In questi casi, per entrare all'interno di
questi batteri, è più utile usare piccoli antibiotici
come la penicillina.
 Antica
resistenza Antica
resistenza
I batteri sono diventati resistenti alla vancomicina
liberandosi del suo bersaglio, la coppia di D-alanine.
La proteina VanA, con l'aiuto di altre due proteine, sostituisce
un gruppo lattato al posto della D-alanina alla fine della catena
peptidica di peptidoglicano. Questo peptide modificato è ancora
capace di fare la reticolazione, ma non è riconosciuto dall'antibiotico.
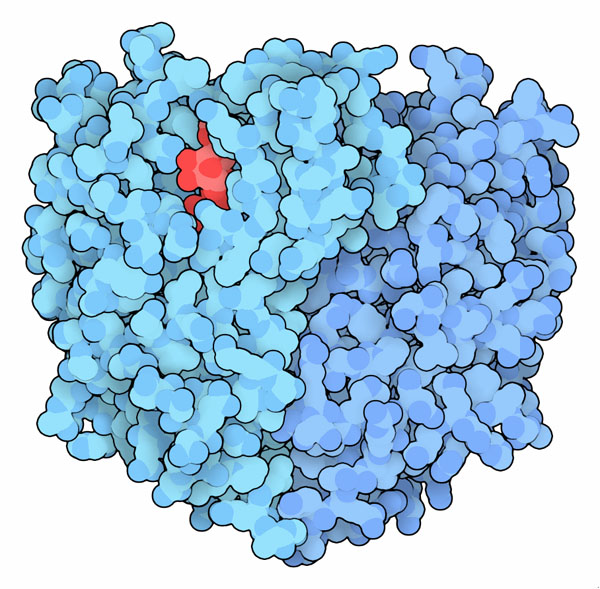
La struttura mostrata qui è quella della proteina VanA di un
antico batterio, ricostruita dal DNA recuperato da sedimenti di permafrost
vecchi di 30.000 anni (file PDB 3se7).
In rosso si vede la molecola di ATP necessaria all'enzima per svolgere
la sua azione. E' incredibilmente simile alla proteina VanA dei batteri
moderni e questo dimostra che la guerra tra antibiotici e resistenza
è cominciata molto prima che la moderna medicina scoprisse l'utilità
degli antibiotici. Potete confrontare voi stessi le strutture della
proteina antica e di quella moderna usando lo strumento "Structure
Comparison Tool" nel sito PDB.
Esplorando la struttura
Possiamo comprendere meglio l'evoluzione della proteina
VanA osservando altre proteine batteriche.
VanA è una D-alanina-D-lattato ligasi, infatti aggiunge
D-lattato alla D-alanina di una catena in crescita del peptidoglicano.
L'enzima che sintetizza il normale peptidoglicano, invece, è
una D-alanina-D-alanina ligasi perchè aggiunge D-alanina
alla D-alanina di una catena in crescita del peptidoglicano.
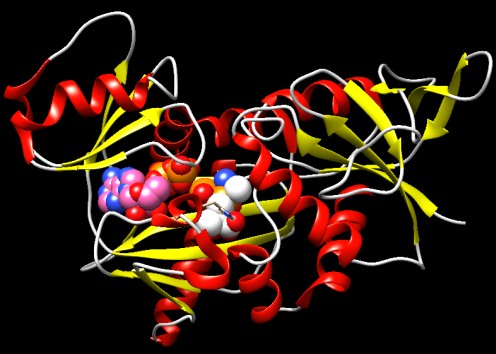
Confrontando le due strutture, (file PDB 1e4e
e 2dln), vediamo che sono molto simili.
Questa è una prova che la resistenza si è evoluta a partire
dall'enzima normale.
Notate che entrambi gli enzimi hanno bisogno di ATP (rosa). La molecola
con sfere bianche occupa il centro del sito attivo.
Nella molecola di sinistra (Van-A) nel sito attivo dove deve
legarsi acido lattico (più grande) è presente una istidina.
Nella molecola di destra (D-Ala-D-Ala ligasi) nello stesso punto
dove deve legarsi D-Alanina (più piccola) è presente una
tisosina (entrambi questi amminoacidi sono mostrati con sticks al centro
della figura).
Spunti per ulteriori esplorazioni
Potete esplorare una varietà di antibiotici
simili e di enzimi coinvolti nella loro sintesi cercando "glycopeptide
antibiotics".
Esaminate la proteina VanX (file PDB 1r44),
anche questa è necessaria per la resistenza alla vancomicina.
Si tratta di un piccolo enzima che rompe ogni peptide che possiede la
sequenza D-alanina - D-alanina nella parete cellulare. In questo modo
consente alla VanA di costruire i suoi peptidi modificati.
 Bibliografia Bibliografia
3se7: V. M. D'Costa, C. E. King, L. Kalan,
M. Morar, W. W. L. Sung, C. Schwarz, D. Froese, G. Zazula, F. Calmels,
R. Debruyne, G. B. Golding, H. N. Poinar & G. D. Wright (2011)
Antibiotic resistance is ancient. Nature 477, 457-461.
1fvm: Y. Nitanai, T. Kikuchi, K. Kakoi, S. Hanamaki, I. Fujisawa
& K. Aoki (2009) Crystal structures of the complexes between vancomycin
and cell-wall precursor analogs. Journal of Molecular Biology
385, 1422-1432.
1e4e: D. I. Roper, T. Huyton, A. Vagin & G. Dodson (2000)
The molecular basis of vancomycin resistance in clinically relevant
Enterococci: crystal structure of D-alanyl-D-lactate ligase (VanA).
Proceedings of the National Academy of Sciences USA 97,
8921-8925.
2dln: C. Fan, P. C. Moews, C. T. Walsh & J. R. Knox (1994)
Vancomycin resistance: structure of D-alanine:D-alanine ligase at 2.3
A resolution. Science 266, 439-443.
J. C. J. Barna, D. H. Williams (1984) The structure and mode
of action of glycopeptide antibiotics of the vancomycin group. Annual
Reviews of Microbiology 38, 339-357.
|
|
|
|