|
|
NewDelhi |
||||||
|
Molecola del Mese di Luglio 2015 Nuovi ceppi batterici che usano la NewDelhi metallo-beta-lattamasi si stanno rivelando altamente resistenti agli antibiotici beta-lattamici 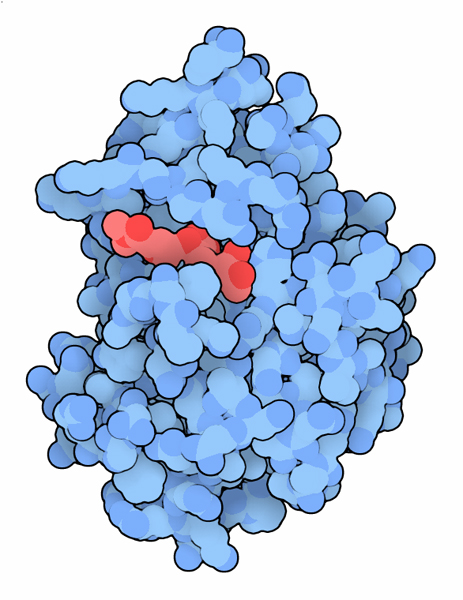 Introduzione IntroduzioneLa scoperta della penicillina (mdm 5/2002) e di altri antibiotici è stata una pietra miliare nella storia dell'umanità, perchè ha permesso finalmente di tenere sotto controllo le malattie infettive che prima spesso si rivelavano mortali. Dopo pochi anni da questa scoperta, però, i batteri hanno cominciato a reagire e sono emerse nuove generazioni di batteri resistenti agli antibiotici. Negli anni seguenti e fino ad oggi, i ricercatori hanno lavorato senza sosta per scoprire nuovi antibiotici, sia naturali che sintetici con i quali combattere questi batteri resistenti. Superbatteri Negli ultimi anni abbiamo dovuto affrontare batteri altamente resistenti in grado di distruggere tutti i farmaci che abbiamo per combatterli. Uno dei ceppi più resistenti è emerso in sud Asia e da qui si è diffuso in tutto il mondo tanto che, da poco, ha causato la morte di molti pazienti anche nei centri medici di Los Angeles. Questi batteri usano l'enzima NDM-1 (New Delhi metallo-ß-lattamasi) per disattivare una grande varietà di antibiotici della famiglia delle penicilline. La struttura qui a destra (file PDB 4eyl) mostra l'enzima dopo che ha distrutto il carbapenem, un antibiotico altamente avanzato nel quale si riponevano grandi speranze. Scambiare informazioni Per rendere le cose ancora più gravi, i batteri resistenti ai farmaci sono in grado di scambiare i loro enzimi di resistenza agli antibiotici con altri batteri. Le informazioni per la sintesi di questi enzimi sono contenute in piccoli tratti di DNA circolare a doppia elica chiamati plasmidi che possono essere trasferiti con facilità da una cellula all'altra. Quindi, quando un nuovo batterio incorpora il plasmide che include il gene che codifica per NDM-1, acquista la resistenza ad una grande varietà di antibiotici. Questa triste realtà sta costringendo i ricercatori a cercare sempre nuovi bersagli per la terapia antibiotica perchè questi superbatteri ormai sono diventati insensibili anche ai migliori antibiotici ß-lattamici, cioè della famiglia delle penicilline. 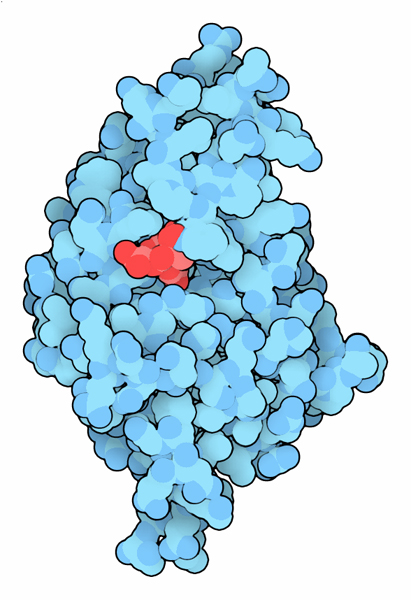 Metodi
diversi Metodi
diversiI batteri resistenti agli antibiotici usano molti metodi diversi per distruggere gli antibiotici. NDM-1 è un metallo-enzima che usa due atomi di zinco per realizzare la sua reazione. Una recente varietà di Klebsiella pneumoniae, che è anche resistente all'antibiotico carbapenem, usa un approccio del tutto diverso: attacca l'antibiotico con un amminoacido serina particolarmente reattivo. I ricercatori stanno già cercando nuove strategie per controbatterlo. La struttura mostrata qui a fianco (file PDB 3rxw) include un farmaco sperimentale che si lega a questa serina e la disattiva. Esplorando la struttura NDM-1 è particolarmente efficace perchè è meno specifico dei normali enzimi. Di solito gli enzimi inglobano completamente il loro substrato in modo da riconoscere solo quella specifica molecola e quindi sono molto selettivi. NDM-1, invece, lega l'antibiotico in superficie e riconosce solo l'anello ß-lattamico, la porzione dell'antibiotico che è attiva contro il batterio ed ignora tutto il resto della molecola. In questo modo può distruggere tutti gli antibiotici ß-lattamici, annullando gli sforzi fatti dai ricercatori negli anni passati di introdurre piccole varianti nelle molecole di penicillina per ingannare le difese dei batteri. L'immagine qui sotto mostra l'enzima legato all'antibiotico Ampicillina (file PDB 3q6x), subito dopo che l'anello ß-lattamico è stato rotto. Nella figura si vedono anche i due atomi di Zinco (magenta) ed una molecola di acqua che partecipa alla reazione (ciano). Passando con il mouse sull'immagine si vede l'enzima legato ad un altro antibiotico, Meticillina. (file PDB 4exs) Notate come le parti variabili degli antibiotici vengono alloggiate senza problemi dato che l'antibiotico si trova sulla superficie esterna dell'enzima. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 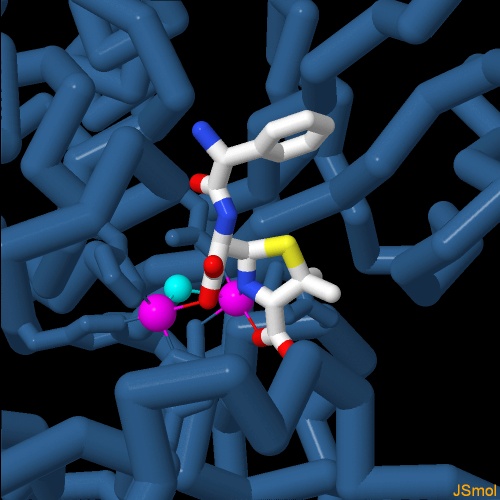 Nella figura successiva si vede l'enzima legato ad altri due antibiotici ß-lattamici Benzylpenicillin (file PDB 4ey2) e Oxacillin (file PDB 4eyf). . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 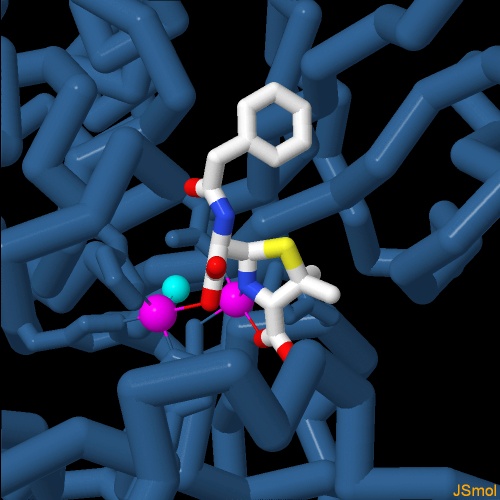
Nella figura seguente si vede l'enzima legato ad altri due antibiotici ß-lattamici Meropenem (file PDB 4eyl) e Faropenem (file PDB 4eyb). . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 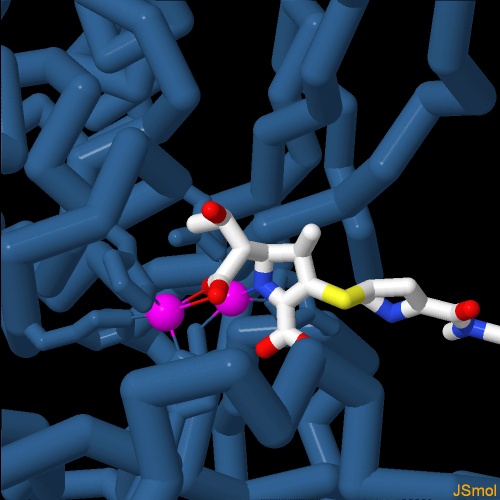 Nella figura qui sotto si vede l'enzima legato ad altri due antibiotici ß-lattamici Cefuroxime (file PDB 4hky) e Cephalexin (file PDB 4rl0). Notate la grande quantità di variazioni introdotte nelle molecole di antibiotico. Sono state prodotte negli anni nel tentativo di superare le resistenza agli antibiotici che i batteri ottenevano producendo enzimi capaci di rompere l'anello ß-lattamico della penicillina. Si sperava che non riconoscessero e quindi non distruggessero i nuovi antibiotici dalla forma modificata. Questo è stato vero fino a quando i batteri non hanno prodotto enzimi a largo spettro come NDM-1, in grado di riconoscere tutti gli antibiotici ß-lattamici. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 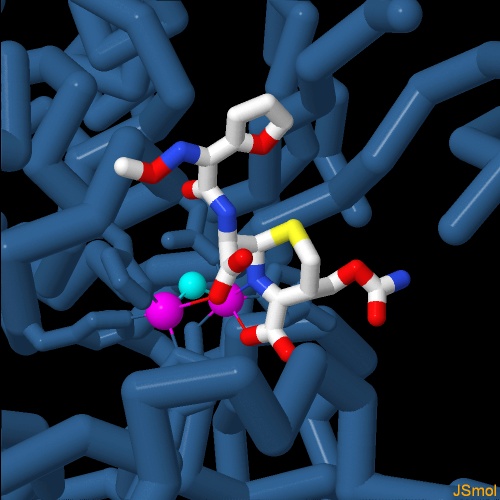 Infine in quest'ultima immagine si vede l'enzima legato ad un inibitore sperimentale, un farmaco progettato per vincere questa resistenza agli antibiotici, L-Captopril (file PDB 4rl2) . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 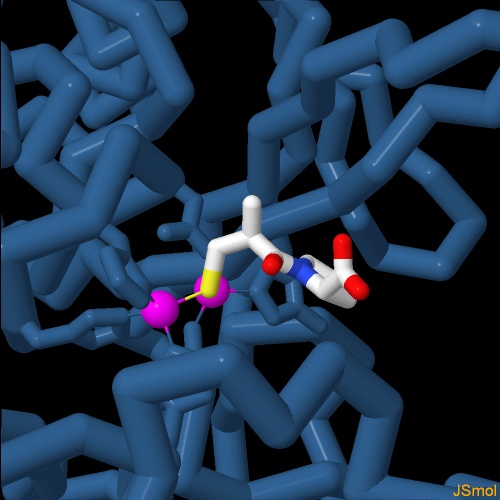 Spunti per ulteriori esplorazioni Negli archivi PDB ci sono molte strutture legate alla resistenza agli antibiotici ß-lattamici. Provate ad esplorare alcune di queste strutture cercando il "Protein Feature View" per TEM-1, KPC-2, NDM-1. Potete esplorare le strutture che coinvolgono diversi antibiotici usando il "Ligand Summary page" e cercando, per esempio "ampicillin". 4rl0, 4rl2: H. Feng, J. Ding, D. Zhu, C. Liu, X. Xu, Y. Zhang, S. Zang, D. C. Wang & W. Liu (2014) Structural and mechanistic insights into NDM-1 catalyzed hydrolysis of cephalosporins. Journal of the American Chemical Society 136, 14694-14697. 3rxw: W. Ke, C. R. Bethel, K. M. Papp-Wallace, S. R. Pagadala, M. Nottingham, D. Fernandez, J. D. Buynak, R. A. Bonomo & F. van den Akker (2012) Crystal structures of KPC-2 ß-lactamase in complex with 3-nitrophenyl boronic acid and the penam sulfone PSR-3-226. Antimicrobial Agents and Chemotherapy 56, 2713-2718. 4exs, 4ey2, 4eyf, 4eyl, 4eyb: D. T. King, L. J. Worrall, R. Gruninger & N. C. Strynadka (2012) New Delhi metallo-ß-lactamase: structural insights into ß-lactam recognition and inhibition. Journal of the American Chemical Society 134, 11362- 11365. 3se7: V. M. D'Costa, C. E. King, L. Kalan, M. Morar, W. W. Sung, C. Schwarz, D. Froese, G. Zazula, F. Calmels, R. Debruyne, G. B. Golding, H. N. Poinar & G. D. Wright (2011) Antibiotic resistance is ancient. Nature 477, 457-461. 3q6x: H. Zhang & Q. Hao (2011) Crystal structure of NDM-1 reveals a common ß-lactam hydrolysis mechanism. FASEB Journal 25, 2574-2582. 1e4e: D. I. Roper, T. Huyton, A. Vagin & G. Dodson (2000) The molecular basis of vancomycin resistance in clinically relevant Enterococci: crystal structure of D-alanyl-D-lactate lyase (VanA). Proceedings of the National Academy of Science USA 97, 8921-8925.
|
|||||||
|
|
|||||||
