|
|
Fototropina |
||||||
|
Molecola del Mese di Marzo 2019 La fototropina sente i livelli di luce blu, consentendo alle piante di rispondere ai cambiamenti delle condizioni ambientali 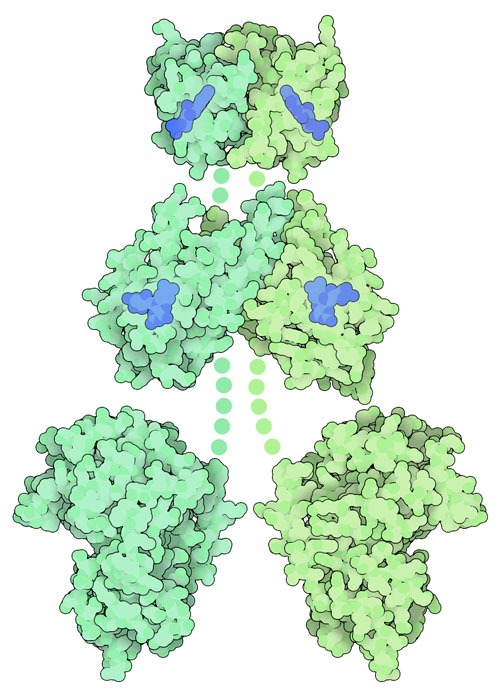 Introduzione IntroduzioneGli scienziati sanno da molti anni che le piante reagiscono alla luce, per esempio crescono verso la sorgente luminosa o rifuggono dalla luce se questa è troppo intensa. In realtà le piante rispondono ai livelli di luce blu. Questa infatti offre molti vantaggi rispetto alla luce di altri colori. E' più ricca di energia e quindi ha un effetto più grande sulle molecole fotosensibili. Per le piante acquatiche, inoltre, la luce blu è quella che penetra più in profondità nell'acqua. La sensibilità delle piante (e di altri organismi) alla luce dipende dal fatto che possiedono delle proteine sensibili alla luce blu che scatenano in esse una risposta. Vedere la luce Qui a fianco è mostrata la fototropina, una proteina fotosensibile che si trova in molti tipi di piante. E' importante per massimizzare l'efficienza della fotosintesi, infatti stimola l'orientamento verso la luce, induce il riposizionamento dei cloroplasti nella foglia verso le zone più illuminate e l'apertura degli stomi. La fototropina è composta di molti domini. Due di questi, chiamati domini LOV1 (in alto) e LOV2 (al centro) (luce, ossigeno o voltaggio), entrambi dimeri, contengono dei cromofori flavinici (blu) che assorbono luce blu. LOV2 è connesso a una chinasi alla serina-treonina (in basso) che propaga il segnale alla cellula quando LOV2 è attivato. Non vi è ancora una struttura completa di questa proteina, così l'immagine qui a fianco è un collage di più strutture. I domini LOV1 e LOV2 sono stati ottenuti dai file PDB 2z6c e 4hhd, e il dominio di chinasi è stato preso da una chinasi simile, di bue (file PDB 1atp). Fatto con amore (with LOV) I domini fotosensibili LOV sono modulari così gli scienziati li hanno ingegnerizzati in vari modi, li hanno legati ad altre proteine per variare leggermente la luce che assorbono. Per esempio, nel PDB sono presenti molecole sensibili alla luce ottenute unendo un dominio LOV con una diversa chinasi sensoria (non mostrata qui, PDB entry 4gcz ) e vi è un dominio LOV ingegnerizzato per emettere fluorescenza in condizioni anaerobiche (mostrato qui sotto, PDB entry 4eeu). Questo si è rivelato utile per marcare molecole nelle cellule viventi in modo più efficace della proteina fluorescente verde GFP (mdm 6-2003) perchè FOV è di più piccole dimensioni. Alcuni amminoacidi della catena di FOV sono stati modificati per ancorare meglio il gruppo flavinico (mostrato con sfere colorate) e questo gli consente di assorbire luce ultravioletta senza degradarsi e di emettere fluorescenza. 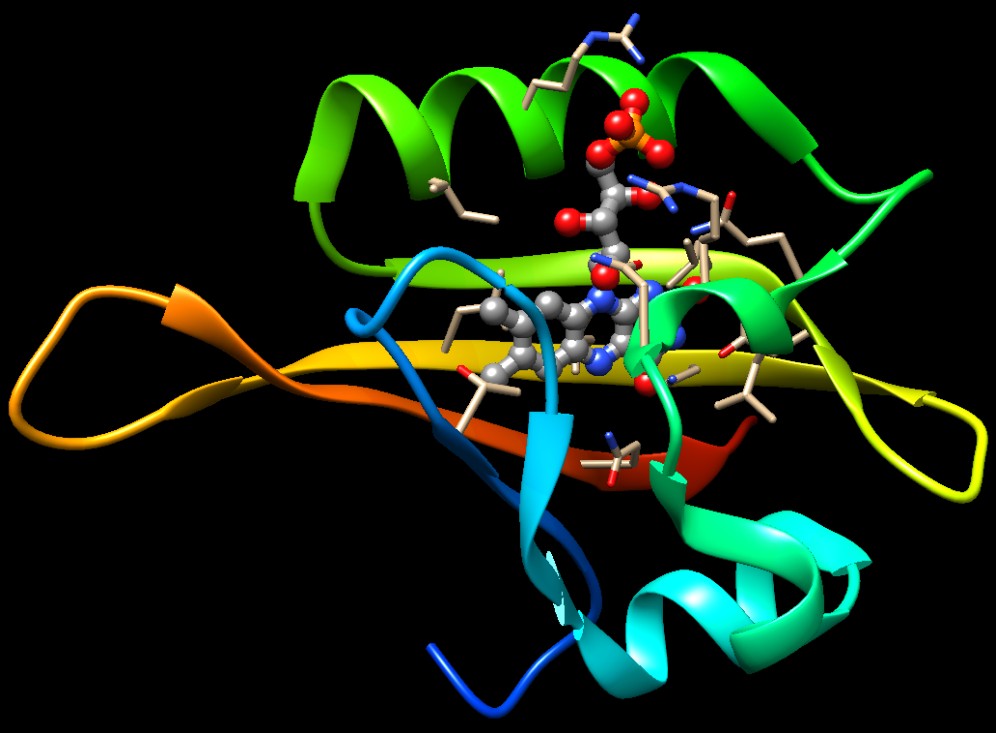 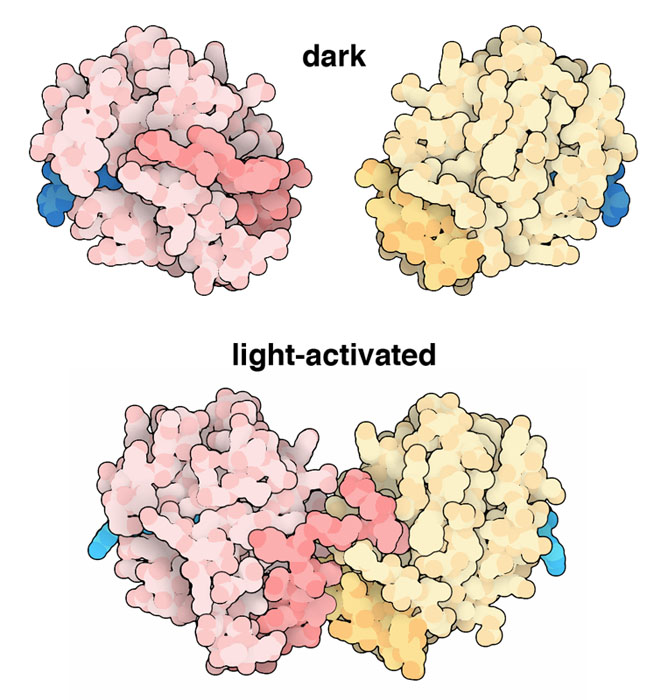 Mandare
il segnale Mandare
il segnaleQuando il cromoforo flavinico assorbe luce, la proteina circostante manda oltre il segnale e questo provoca la risposta della pianta. Nella fototropina, il segnale passa ad un dominio di chinasi che modifica, fosforilandole, altre proteine di segnalazione della cellula. I dettagli di questo processo sono ancora oggetto di studi, ma alcuni indizi sono stati scoperti nel sensore di luce blu Vivid che si trova nei funghi. E' un sensore più semplice della fototropina e possiede un solo dominio LOV. Al buio questo esiste come monomero come si vede qui a fianco in alto (file PDB 2pd7). Quando il cromoforo (blu) assorbe luce blu, costringe un'alfa elica della proteina a spostarsi (mostrata con colori più rossi a sinistra e più gialli sulla destra). Questo induce la proteina a formare dei dimeri come si vede qui a fianco in basso (file PDB 3rh8). Questo meccanismo di dimerizzazione di Vivid fornisce i dettagli molecolari che spiegano come una classe di fotorecettori converte la risposta alla luce in nuove interazioni proteina-proteina. Esplorando la struttura Gli scienziati hanno determinato la struttura dei domini LOV della fototropina, e di altre proteine sensibili alla luce blu, sia al buio sia dopo che hanno assorbito la luce. Nella fototropina, il gruppo flavinico foto attivato dalla luce reagisce con una cisteina adiacente (grigia e gialla) formando un legame covalente. Questo distorce i tre anelli flavinici e anche la porzione di proteina circostante. Le due strutture sono mostrate qui sotto (file PDB 1g28 e 1jnu). . . . 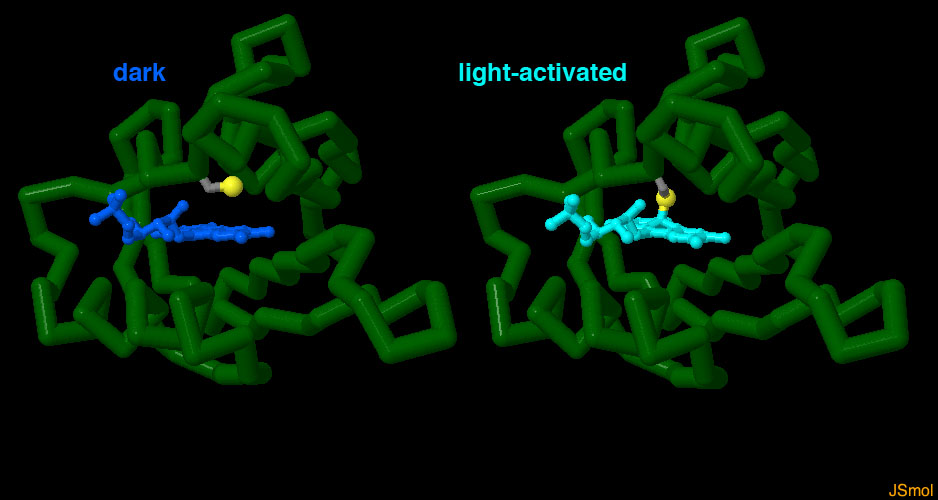 Spunti per ulteriori esplorazioni Vi sono molte altre proteine sensibili alla luce. Per cercare proteine simili alla fototropina cercate nel sito PDB per "LOV", oppure, per cercare proteine diverse, cercate "cryptochrome" or "BLUF". I gruppi flavinici si trovano anche nei coenzimi trasportatori di elettroni. Cercate nel sito PDB per "FAD". Usate la "Protein Feature View" per confrontare tra loro i domini diversi di fototropina, per esempio nella pagina della fototropina di Arabidopsis. K. S. Conrad, C. C. Manahan & B. R. Crane (2014) Photochemistry of flavoprotein light sensors. Nature Chemical Biology 10, 801-809. 4gcz: R. P. Diensthuber, M. Bommer, T. Gleichmann & A. Moglich (2013) Full-length structure of a sensor histidine kinase pinpoints coaxial coiled coils as signal transducers and modulators. Structure 21, 1127-1136. 4hhd: A. S. Halavaty & K. Moffat (2013) Coiled-coil dimerization of the LOV2 domain of the blue-light photoreceptor phototropin 1 from Arabidopsis thaliana. Acta Crystallographica F69, 1316-1321. 4eeu: J. M. Christie, K. Hitomi, A. S. Arvai, K. A. Hartfield, M. Mettlen, A. J. Pratt, J. A. Tainer & E. D. Getzoff (2012) Structural tuning of the fluorescent protein iLOV for improved photostability. Journal of Biological Chemistry 287, 22295-22304. A. Losi and W. Gartner (2011) Old chromophores, new photoactivation paradigms, trendy applications: flavins in blue light-sensing photoreceptors. Photochemistry and Photobiology 87, 491-510. 3rh8: A. T. Vaidya, C. H. Chen, J. C. Dunlap, J. J. Loros & B. R. Crane (2011) Structure of a light-activated LOV protein dimer that regulates transcription. Science Signaling 4, ra50. 2z6c: M. Nakasako, K. Zikihara, D. Matsuoka, H. Katsura & S. Tokutomi (2008) Structural basis of the LOV1 dimerization of Arabidopsis phototropins 1 and 2. Journal of Molecular Biology 381, 718-733. 2pd7: B. D. Zoltowski, C. Schwerdtfeger, J. Widom, J. J. Loros, A. M. Bilwes, J. C. Dunlap & B. R. Crane. (2007) Conformational switching in the fungal light sensor Vivid. Science 316, 1054-1057. 1jnu: S. Crosson & K. Moffat (2002) Photoexcited structure of a plant photoreceptor domain reveals a light-driven molecular switch. The Plant Cell 14, 1067-1075. 1g28: S. Crosson & K. Moffat (2001) Structure of a flavin-binding plant photoreceptor domain: insights into light-mediated signal transduction. Proceedings of the National Academy of Sciences USA 98, 2995-3000.
|
|||||||
|
|
|||||||
