|
|
Cascade e CRISPR |
||||
|
Molecola del Mese di Gennaio 2015 La tecnica CRISPR-Cas9 permette di riconoscere e tagliare un tratto specifico di DNA usando come sonda un semplice tratto di RNA 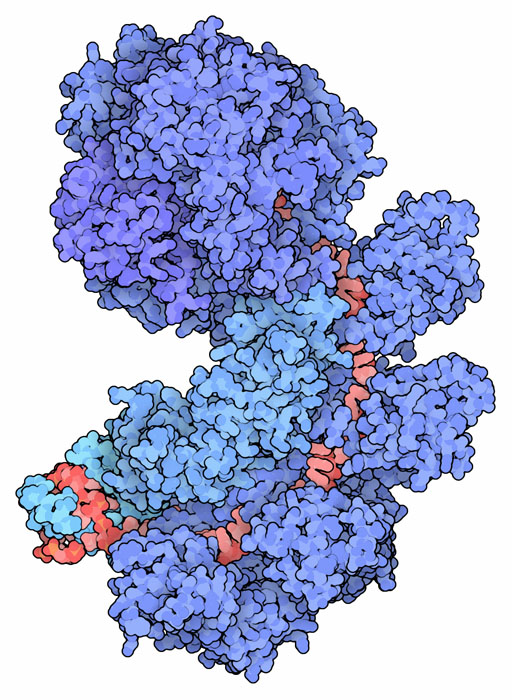 Introduzione IntroduzioneCome ogni altro organismo vivente, anche i batteri e gli archea sono continuamente attaccati dai virus e quindi hanno sviluppato molte armi per difendersi. La loro prima linea di difesa è un sistema immunitario innato che usa gli enzimi di restrizione per tagliare il DNA dei virus invasori. Per proteggersi ulteriormente contro i virus che li attaccano, hanno anche un sistema immunitario adattativo, simile al nostro. Questo sistema, chiamato CRISPR-Cas, contiene le informazioni di tutte le minacce passate e fornisce le armi per distruggere l'aggressore in caso di un nuovo attacco. Archivio delle infezioni I batteri usano le sequenze CRISPR, conservate nel loro genoma, per identificare i virus che li attaccano. La sigla sta per "Clustered Regularly Interspaced Short Palindromic Repeats," (brevi ripetizioni palindrome raggruppate e spaziate con regolarità). Il DNA CRISPR è composto di piccoli tratti di DNA virale ottenuto da frammenti dei virus che li hanno attaccati nel passato (tratti spaziatori), separati da una sequenza ripetuta e caratteristica usata per creare l'archivio (tratti ripetenti). La cosa importante è che le nuove sequenze sono aggiunte all'inizio dell'archivio e così, leggendo la sequenza CRISPR, possiamo conoscere la storia delle infezioni virali subite da una popolazione batterica nel passato. Sorveglianza continua Un sistema di proteine Cas (CRISPR associated proteins) usa questo archivio di informazioni per combattere i virus quando cercano di infettare di nuovo il batterio. Il centro di questo sistema è il grande complesso proteico Cascade. Questo lega un RNA trascritto dalla sequenza CRISPR e cerca nella cellula i DNA virali complementari, le tracce dell'infezione. Se trova un DNA virale corrispondente, la proteina si apre e mette in azione delle nucleasi per tagliarlo. La struttura mostrata qui a fianco include il complesso di sorveglianza Cascade composto di 6 proteine diverse (mostrate nei vari toni del blu) insieme con un frammento di RNA trascritto (rosso) (file PDB 4tvx e 4u7u). 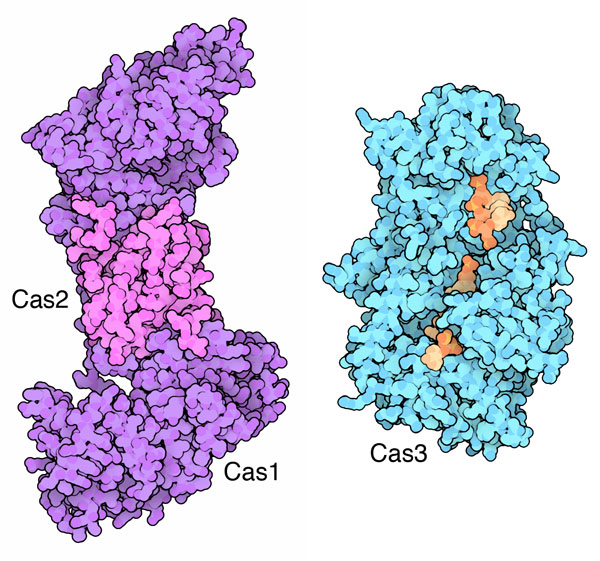 Proteine Cas Il complesso Cascade lavora con un gruppo di proteine per costruire l'archivio CRISPR che poi usa per proteggere i batteri dai virus. In un primo momento, proteine reclutanti come Cas1 e Cas2 (file PDB 4p6i) vengono usate per tagliare i virus durante l'infezione e per archiviare frammenti di lunghezza opportuna nel CRISPR. L'RNA complementare trascritto da questi pezzi viene poi esibito da Cascade per combattere eventuali infezioni future di quel virus. Se il virus viene individuato, entra in azione Cas3, il demolitore, una endonucleasi che prende il DNA virale infettante individuato da Cascade e lo taglia. 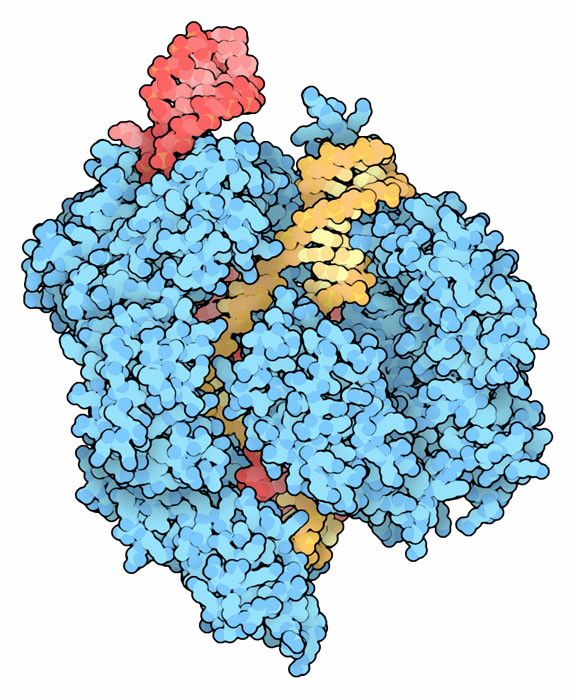 Cas9 e nuove terapie Tipi diversi di batteri e di archea hanno sviluppato almeno tre variazioni su questo tema di base. Il complesso Cascade di tipo I esibisce l'RNA trascritto dal CRISPR e utilizza come demolitore Cas3 per tagliare il DNA virale. Il complesso Cascade di tipo III usa la stessa strategia per individuare il DNA virale, ma l'enzima che lo taglia fa parte dello stesso complesso proteico. Il complesso Cascade di tipo II, come Cas9 mostrato qui a fianco (file PDB 4un3 e 4oo8), ha la proteina di sorveglianza e quella demolitrice unite in una sola catena proteica. Il complesso Cas9, mostrato qui a fianco, include l'RNA ottenuto dal CRISPR (rosso) insieme con un pezzo di DNA virale catturato (giallo). Questa molecola è stata usata di recente in una cura sperimentale contro l'infezione latente da HIV. E' stato usato un virus ingegnerizzato per inserire Cas9 e il CRISPR anti-HIV nelle cellule infettate, questo complesso ha poi tagliato a pezzi il DNA virale che vi si era legato. La tecnica CRISPR-Cas9 permette di riconoscere e tagliare un tratto specifico di DNA usando come sonda un semplice tratto di RNA complementare. Questo è molto più facile da ottenere rispetto alle sequenze di proteina che servono come sonda nella tecnica delle dita di zinco (mdm 3-2007) o delle proteine TALEN (mdm 12-2014). Per questo la tecnica CRISPR-Cas9 sta trovando grande diffusione e sta aprendo nuovi orizzonti anche nell'editing del DNA o nel controllo dell'espressione dei geni. Nell'immagine seguente dello stesso complesso Cas9 (file PDB 4un3), la proteina è mostrata in modo più schematico, così si vede meglio che l'RNA ottenuto dal CRISPR (rosso) è costituito da una parte ripiegata su sè stessa, il tratto ripetente, e una parte complementare al DNA virale, lo spaziatore, di 20 nucleotidi. Nella figura qui sotto si vede che una delle catene del DNA virale si è appaiata al tratto spaziatore di RNA ottenuto dal CRISPR. A questo punto i due filamenti di DNA virale devono essere tagliati. Uno dei due è già stato tagliato, mentre l'altro filamento è ancora intero e quindi stiamo osservando il complesso CRISPR-Cas9 nel mezzo della sua azione. 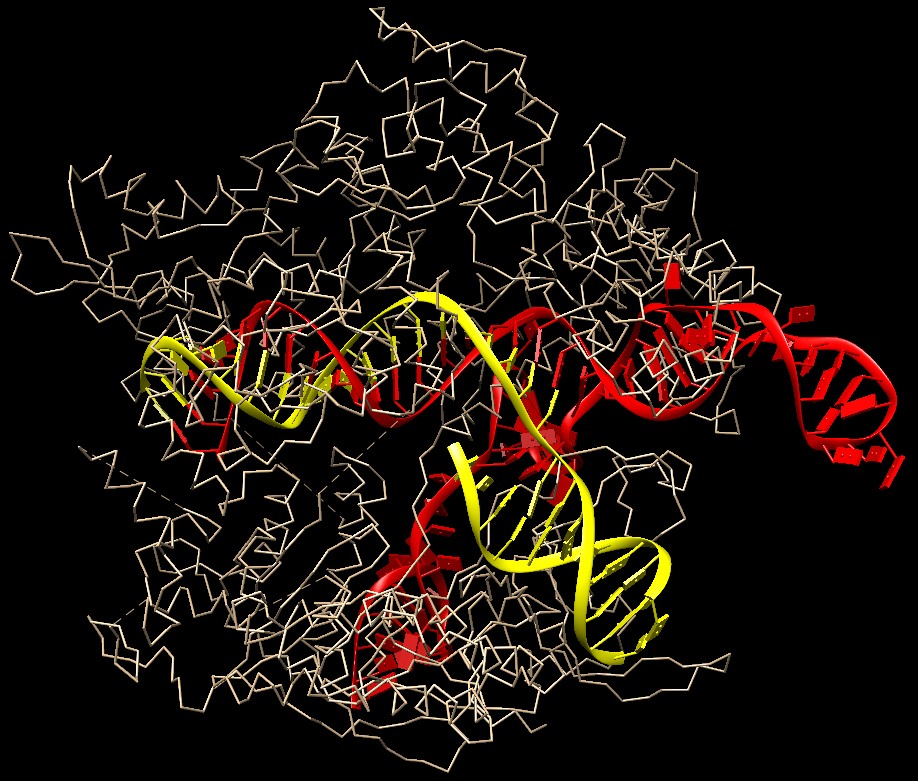 Nel DNA virale (giallo), il tratto riconosciuto da Cas9 è sempre preceduto da una sequenza caratteristica 5'-NGG-3' chiamata PAM che in questo caso è TGG come si vede nell'immagine più sotto. Questa sequenza aiuta Cas9 a separare i due filamenti con facilità ed è riconosciuta da alcuni amminoacidi di Cas9 che interagiscono con le scanalature del DNA virale. 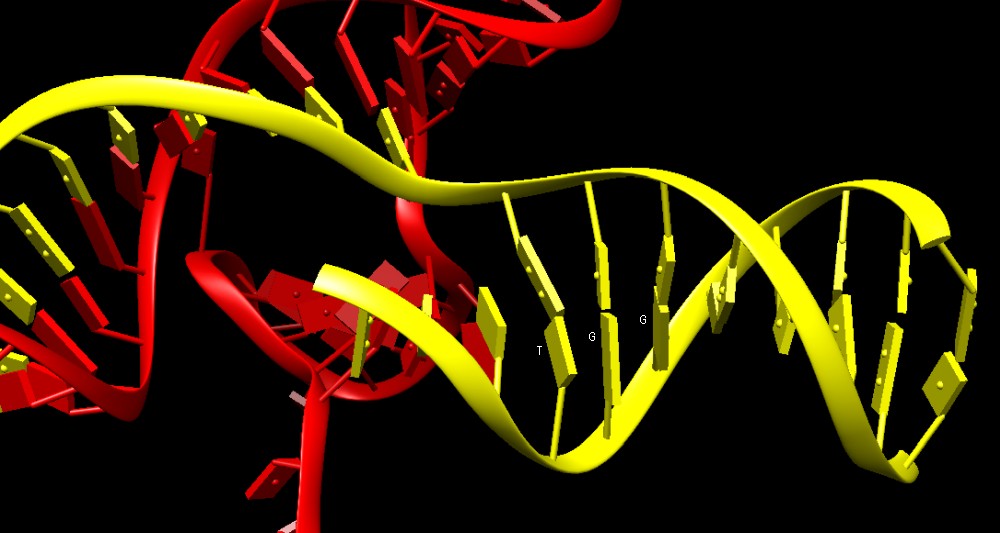 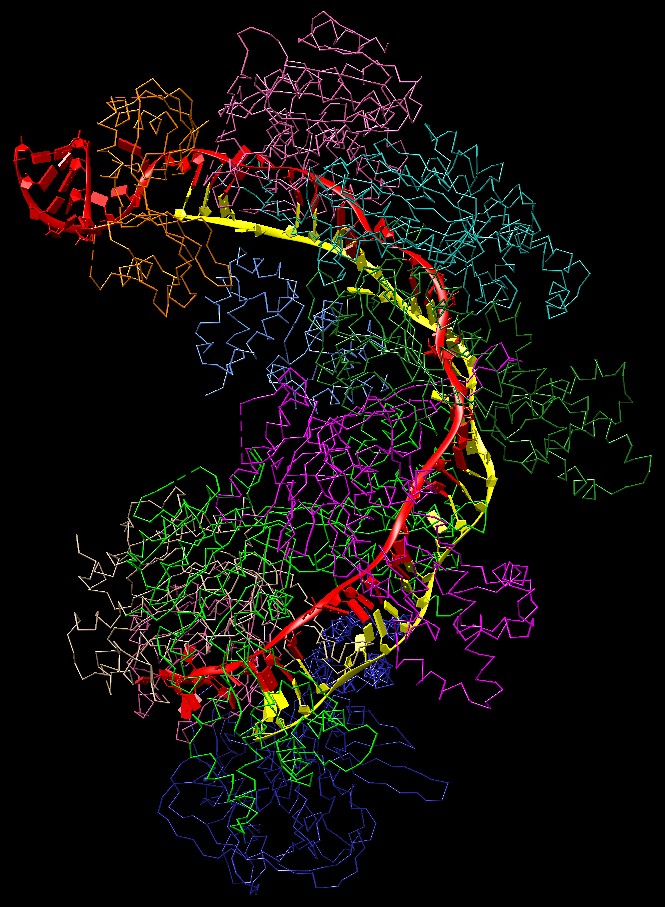 Esplorando
la struttura Esplorando
la strutturaIl file PDB 4qyz mostra un complesso Cascade di tipo III in azione. La struttura include la catena di un CRISPR-RNA (rosso) ed un piccolo pezzo di DNA virale (giallo) dopo che è stato svolto e riconosciuto. Nel CRISPR RNA (rosso) si riconosce, in alto, una parte ripiegata su se stessa, il tratto ripetente, e una parte complementare al DNA virale, il tratto spaziatore, allungata verso il basso. Questo complesso ha una struttura sorprendente, ma logica del complesso DNA-RNA. L'RNA è aperto e stirato all'interno di una lunga tasca curva di Cascade, mentre il DNA virale (giallo) si è legato fianco a fianco all'RNA CRISPR invece di formare la normale doppia elica. Notate infine che il complesso Cascade non è formato da una sola catena proteica, ma da ben undici catene diverse associate tra loro. Queste ultime tre immagini sono state ottenute con Chimera (vedi Chimica al Computer). Spunti per ulteriori esplorazioni Quando leggete articoli sulle sequenze CRISPR, fate attenzione alla terminologia perchè può trarre in inganno. Per esempio il termine "spacer" è usato per i corti pezzi di DNA virale che sono custoditi in CRISPR e "repeat" è usato per riferirsi alle brevi sequenze ripetute che separano i vari pezzi di DNA virale. Molti di questi grandi complessi CRISPR-Cas sono stati caratterizzati con la microscopia elettronica. Per esempio, per vedere la struttura del complesso di tipo III (che è diverso dai complessi di tipo I e di tipo II) visitate EMDataBank. J. van der Oost, E. R. Westra, R. N. Jackson & B. Wiedenheft (2014) Unravelling the structural and mechanistic basis of CRISPR-Cas systems. Nature Reviews Microbiology 12, 479-492. H. Ebina, N. Misawa, Y. Kanemura & Y. Koyanagi (2013) Harnessing the CRISPR/Cas9 system to disrupt latent HIV-1 provirus. Scientific Reports 3, 2510. 4qqw: Y. Huo, K. H. Nam, F. Ding, H. Lee, L. Wu, Y. Xiao, M. D. Farchione, S. Zhou, K. Rajashankar, I. Kurinov, R. Zhang & A. Ke (2014) Structures of CRISPR Cas3 offer mechanistic insights into Cascade-activated DNA unwinding and degradation. Nature Structural & Molecular Biology 21, 771-777. 4un3: C. Anders, O. Niewoehner, A. Duerst & M. Jinek (2014) Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature 513, 569-573. 4tvx: R. N. Jackson, S. M. Golden, P. B. G. van Erg, J. Carter, E. R. Westra, S. J. J. Brouns, J. van der Oost, T. C. Terwilliger, R. J. Read & B. Wiedenheft. (2014) Crystal structure of the CRISPR RNA-guided surveillance complex from Escherichia coli. Science 345, 1473-1479. 4qyz: S. Mulepati, A. Heroux & S. Bailey (2014) Crystal structure of a CRISPR RNA-guided surveillance complex bound to a ssFNA target. Science 345, 1479-1484. 4p6i: J. K. Nunez, P. J. Kranzusch, J. Noeske, A. V. Wright, C. W. Davies & J. A. Doudna (2014) Cas1-Cas2 complex formation mediates spacer acquisition during CRISPR-Cas adaptive immunity. Nature Structural & Molecular Biology 21, 528-534.
|
|||||
|
|
|||||
