|
|
Piruvato deidrogenasi |
|||||
|
Molecola del Mese di Settembre 2012 La decarbossilazione ossidativa dell'acido piruvico avviene in tre reazioni consecutive realizzate da un grande complesso enzimatico 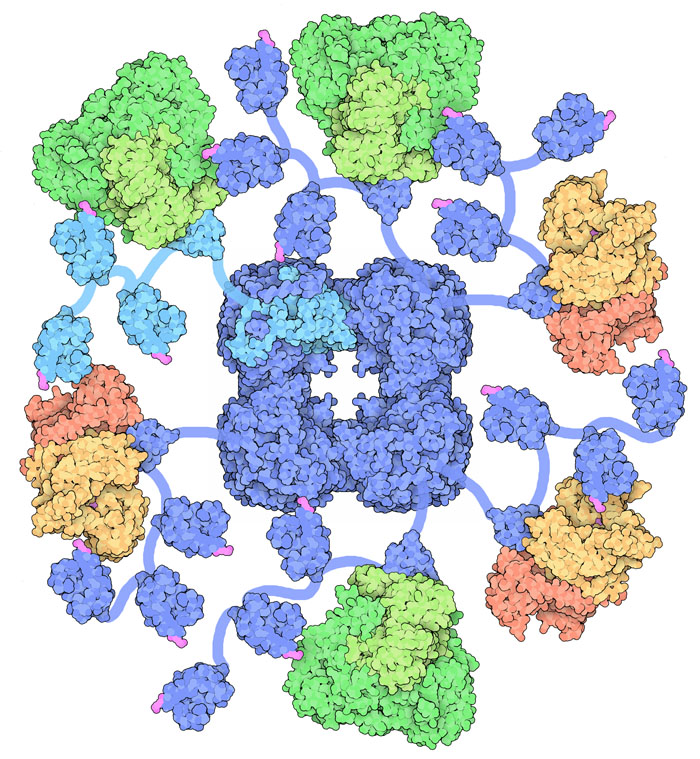 Introduzione Una combinazione di più tecniche, cristallografia ai raggi X, spettroscopia NMR, e microscopia elettronica, sta rivelando i segreti dell'enzima piruvato deidrogenasi. Questo enzima si trova nei mitocondri, organelli cellulari specializzati nelle reazioni ossidative, e qui realizza la decarbossilazione ossidativa, una tappa centrale della produzione di energia nella cellula, che collega la glicolisi al ciclo di Krebs. Questa reazione viene realizzata in tre passaggi da tre enzimi diversi (E1, E2, E3 rappresentati qui a fianco in verde, blu e giallo-arancione), legati tra loro in un grande complesso multienzimatico, chiamato piruvato deidrogenasi. Anatomia di un complesso multienzimatico Il complesso ha molte parti mobili che lavorano insieme per realizzare la reazione globale. E' costruito attorno ad un nocciolo simmetrico (E2) mostrato in blu. Tutte le catene proteiche che compongono il nocciolo terminano con una lunga coda flessibile che si avvolge per realizzare molti altri domini funzionali. Uno di questi domini lega gli altri enzimi del complesso, mostrati in verde (E1) e in giallo-aramcione (E3). Vi sono poi molti altri piccoli domini blu che agiscono da trasportatori. Una amminoacido lisina su questi trasportatori, mostrato in magenta, è legato ad una speciale molecola, l'acido lipoico (non mostrato nella struttura). Questo trasporta le molecole parzialmente reagite da E1 (verde, dove il piruvato viene decarbossilato) ad E2 (nocciolo blu, dove il residuo viene ossidato dall'acido lipoico e trasferito al coenzima A). Alla fine il trasportatore si porta in E3 (giallo-arancione) dove l'acido lipoico ridotto viene ossidato ad opera di FAD e NAD+. Nell'immagine qui sopra il complesso enzimatico è stato rappresentato in modo semplificato mostrando solo 6 delle 24 code proteiche che in realtà possiede. Nel complesso vero il nocciolo centrale è completamente circondato da catene proteiche formando una grossa palla. Scoprire il complesso Dato che il complesso è flessibile, è stato molto difficile da studiare, infatti solo le proteine che hanno una forma ben definita si possono vedere con la tecnica della diffrazione ai raggi X, o con la spettroscopia NMR. Per questo motivo i chimici strutturisti hanno scelto la via del "divide et impera" e hanno diviso il complesso in piccoli frammenti, questi, avendo una struttura più rigida, sono stati determinati con facilità. Infine, per capire come andavano assemblati i vari frammenti, hanno usato immagini a bassa risoluzione ottenute con il miscroscopio elettonico. Nell'immagine qui sopra sono state incluse le strutture di più frammenti: il nocciolo centrale E2 (blu, file PDB 1eaa), i domini di trasporto (blu, file PDB 1lac), e gli enzimi legati alle code proteiche E1 (verde, file PDB 1w85) ed E3 (giallo-arancione, file PDB 1ebd). L'importanza di assumere vitamine 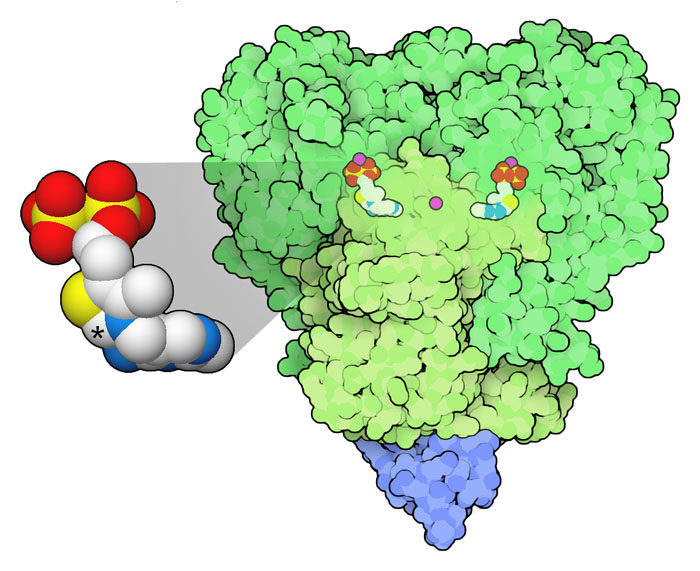 La
reazione di decarbossilazione ossidativa che viene compiuta dalla piruvato
deidrogenasi è complessa, e quindi utilizza diverse molecole di
supporto. L'enzima E1 che realizza il primo passaggio, la decarbossilazione,
è mostrato qui a destra (file PDB 1w85)
e usa tiamina pirofosfato (vitamina B1) per estrarre anidride carbonica,
CO2, dal piruvato.
La molecola di tiamina ha un atomo di carbonio particolarmente reattivo,
mostrato qui con un asterisco, che compie l'attacco al piruvato. Anche
altre molecole sono indispensabili per realizzare la reazione complessiva:
i piccoli domini trasportatori usano acido lipoico per legare il
piruvato (dopo che ha perso CO2)
e ossidarlo ad acetile e alla fine (in E2) cedono l'acetile ad un'altra
molecola strana, il coenzima A (che contiene acido pantotenico,
vitamina B5). L'enzima che realizza l'ultimo passaggio (E3) usa FAD
(che contiene riboflavina, vitamina B2) e NAD (che contiene niacina,
vitamina B3 o PP) per riportare l'acido lipoico nella sua forma iniziale
ridotta. Le nostre cellule non sono in grado di sintetizzare molte di
queste molecole complesse e quindi le dobbiamo assumere con la dieta,
per questo si chiamano vitamine, ammine della vita. La
reazione di decarbossilazione ossidativa che viene compiuta dalla piruvato
deidrogenasi è complessa, e quindi utilizza diverse molecole di
supporto. L'enzima E1 che realizza il primo passaggio, la decarbossilazione,
è mostrato qui a destra (file PDB 1w85)
e usa tiamina pirofosfato (vitamina B1) per estrarre anidride carbonica,
CO2, dal piruvato.
La molecola di tiamina ha un atomo di carbonio particolarmente reattivo,
mostrato qui con un asterisco, che compie l'attacco al piruvato. Anche
altre molecole sono indispensabili per realizzare la reazione complessiva:
i piccoli domini trasportatori usano acido lipoico per legare il
piruvato (dopo che ha perso CO2)
e ossidarlo ad acetile e alla fine (in E2) cedono l'acetile ad un'altra
molecola strana, il coenzima A (che contiene acido pantotenico,
vitamina B5). L'enzima che realizza l'ultimo passaggio (E3) usa FAD
(che contiene riboflavina, vitamina B2) e NAD (che contiene niacina,
vitamina B3 o PP) per riportare l'acido lipoico nella sua forma iniziale
ridotta. Le nostre cellule non sono in grado di sintetizzare molte di
queste molecole complesse e quindi le dobbiamo assumere con la dieta,
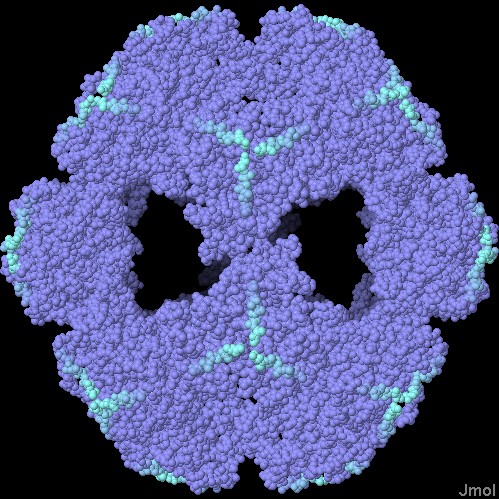
per questo si chiamano vitamine, ammine della vita.Esplorando la struttura Come accade spesso in biochimica, le cose si rivelano ancora più complesse quando vengono osservate con maggior dettaglio. Esistono molte varianti della piruvato deidrogenasi nei vari tipi di cellule. Alcune cellule hanno uno o due domini di trasporto sulle code proteiche flessibile invece dei tre mostrati qui. Il complesso nelle nostre cellule include un'altra proteina nel nocciolo che possiede dei propri domini di trasporto. Inoltre lo stesso nocciolo può avere geometrie diverse: un nocciolo a forma di cubo composto di 24 subunità (mostrato qui sotto a sinistra PDB 1eaa) o un nocciolo a forma di dodecaedro composto di 60 subunità (mostrato qui sotto a destra, file PDB 1b5s). Il nocciolo qui sotto sulla sinistra (a cubo) è stato preso da Azobacter, quello sulla destra (a dodecaedro) è di Geobacillus. 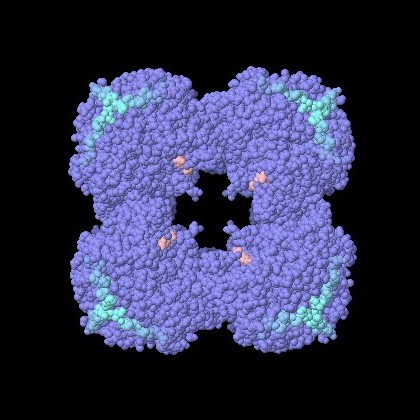 . . .
. . . 
Qui sotto sono mostrati gli stessi due noccioli lievemente ruotati per apprezzare la loro complessità tridimensionale. La fine di ogni catena è stata colorata in ciano, da qui escono le code proteiche che costituiscono ulteriori domini e che legano gli altri due enzimi del complesso. 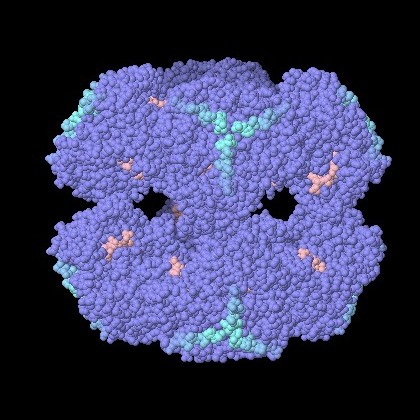 . . .
. . . 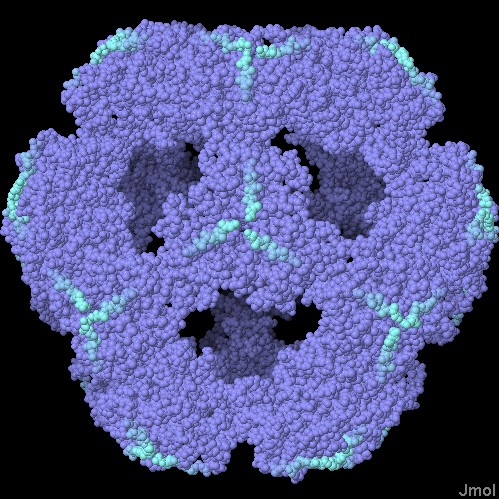 Nel nocciolo di sinistra sono anche state evidenziate in rosa le molecole di coenzima A legate nei siti attivi dell'enzima. Spunti per ulteriori esplorazioni - Vengono scoperte sempre più proteine che sono composte da molti domini funzionali collegati da braccia flessibili. Ne volete cercare qualcuna negli archivi PDB? - Vi sono molti enzimi che usano coenzimi ottenuti da vitamine come la tiamina (B1), la niacina (B3), la riboflavina (B2). Questi coenzimi svolgono funzioni simili oppure hanno funzioni ben distinte tra loro? D. L. Nelson and M. M. Cox (2008) Lehninger Principles of Biochemistry. Worth Publishers, New York. X. Yu, Y. Hiromasa, H. Tsen, J. K. Stoops, T. E. Roche and Z. H. Zhou (2008) Structures of the human pyruvate dehydrogeanse complex cores: a highly conserved catalytic center with flexible N-terminal domains. Structure 16, 104-114. J. L. S. Milne, X. Wu, M. J. Borgnia, J. S. Lengyel, B. R. Brooks, D. Shi, R. N. Perham and S. Subramaniam (2006) Molecular structure of a 9-MDa icosahedral pyruvate dehydroegnase subcomplex containing the E2 and E3 enzymes using cryoelectron microscopy. Journal of Biological Chemistry 281. 4364-4370. T. Izard, A. Aevarsson, M. D. Allen, A. H. Westphal, R. N. Perham, A. deKok and W. G. J. Hol (1999) Principles of quasi-equivalence and Euclidean geometry govern the assembly of cubic and dodecahedral cores of pyruvate dehydrogenase complexes. Proceedings of the National Academy of Sciences USA 96, 1240-1245.
|
||||||
|
|
||||||
