|
|
Concanavalina A |
|||||
|
Molecola del Mese di Aprile 2010 Alcune proteine mantengono la stessa struttura tridimensionale anche se iniziano e finiscono in punti diversi della catena 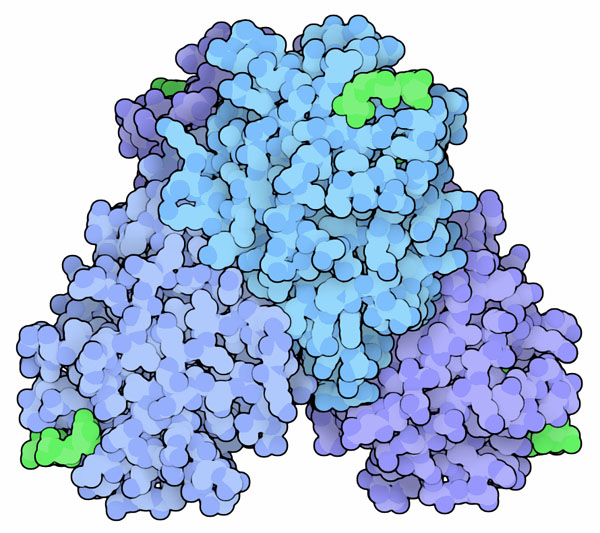 Introduzione IntroduzioneL'evoluzione, quando trova un progetto di successo, lo usa ancora e ancora introducendo, se possibile, variazioni e miglioramenti. Gli esempi di questo tipo sono moltissimi. La maggior parte dei mammiferi ha quattro zampe che hanno assunto le forme più diverse e che si sono evolute in braccia e gambe e persino in pinne e ali. La maggior parte delle piante sono coperte di foglie, che hanno assunto forme molto diverse, da quella ad ago delle conifere a quella a lamina espansa delle latifoglie. Anche nella struttura e nella sequenza delle proteine troviamo grandi diversità generate da variazioni. Le proteine, in generale, si evolvono attraverso una lenta mutazione di singoli amminoacidi, ma si possono anche evolvere attraverso cambiamenti più grandi che coinvolgono interi tratti proteici quando i geni delle proteine vengono tagliati e riassemblati in un nuovo ordine. Permutazione circolare La permutazione circolare è uno dei più strani cambiamenti scoperti nell'evoluzione delle proteine. Per capire da dove viene il nome "permutazione circolare" immaginate una proteina ripiegata che abbia le due porzioni terminali una vicino all'altra. Se legate chimicamente le due estremità ottenete una proteina costituita da una catena circolare, che non ha amminoacidi terminali. Se ora tagliate la proteina circolare in un punto diverso, ottenete una proteina ripiegata molto simile a quella originale, a parte il fatto che gli amminoacidi terminali sono in un punto diverso. 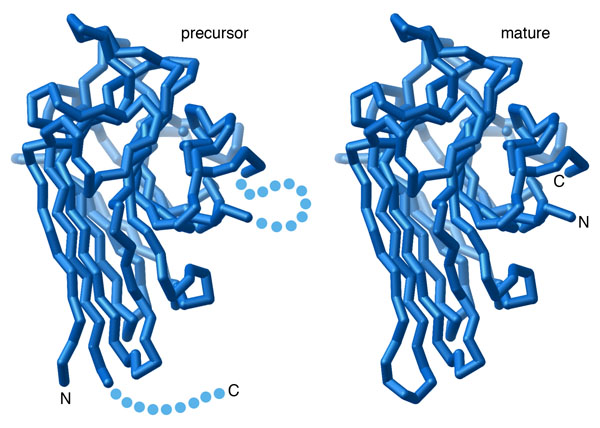 Proteine
permutate Proteine
permutateQuesto è proprio quello che succede con la concanavalina A, una proteina che lega i carboidrati, che si trova in un fagiolo tropicale (Canavalia ensiformis o Jack Bean), mostrata qui a fianco (file PDB 1cvn). Quando la sequenza di amminoacidi di questa proteina è stata determinata negli anni '70, si è scoperto che aveva una sequenza simile a quella della favina, una proteina che si trova nella pianta della fava (Vicia faba). La sequenza però è permutata come se la catena proteica fosse stata tagliata a metà e i due frammenti invertiti. Ulteriori studi hanno mostrato che, nella cellula, la concanavalina A è sintetizzata in modo simile alla favina, ma poi si verifica una permutazione circolare come quella illustrata qui a fianco (file PDB 3cna). La proteina precursore, formata direttamente dai ribosomi, ha un tratto di coda in più e un tratto centrale in più, mostrati con puntini in figura. La proteina matura si ottiene tagliando via la coda e il tratto centrale per formare due nuovi terminali di catena (C e N) e unendo chimicamente gli amminoacidi terminali della catena originale per formare un tratto continuo. Permutare i geni Dopo quello della concanavalina A, sono stati scoperti molti altri esempi di permutazione circolare. Nella maggior parte dei casi, però, la permutazione circolare avviene nel genoma e non attraverso il laborioso taglia e cuci sulla proteina già formata. Si sono osservati, infatti, dei riarrangiamenti genetici: la porzione iniziale del gene viene prima rimossa e poi inserita alla fine del gene. Nella proteina codificata da geni permutati, qualche volta risulta spostata solo una singola elica (come nelle proteine dei file PDB 1ui9 e 1h0r), in altri casi sono invertiti interi domini. Permutazioni intenzionali Appena gli scienziati hanno scoperto il processo della permutazione circolare, hanno subito cercato di riprodurlo. La proteina glucanasi è stata permutata in vari modi tagliando il gene in punti diversi e poi invertendo i due frammenti. La figura qui sotto mostra la proteina glucanasi nativa e alcune di queste varianti (file PDB 2ayh, 1ajk, 1ajo e 1cpm), in ogni catena le due estremità sono state colorate una di blu e l'altra di rosso mentre i tratti intermedi sono arcobaleno. Notate che tutte e quattro le proteine sono ripiegate nello stesso modo, la sola differenza è che le catene iniziano e finiscono in punti diversi, come è evidenziato dai colori blu e rosso. Questo dimostra che il ripiegamento delle proteine è un processo robusto ed è determinato dalla sequenza di amminoacidi della proteina anche se questi sono un po' rimescolati. . . . . . . . . . . 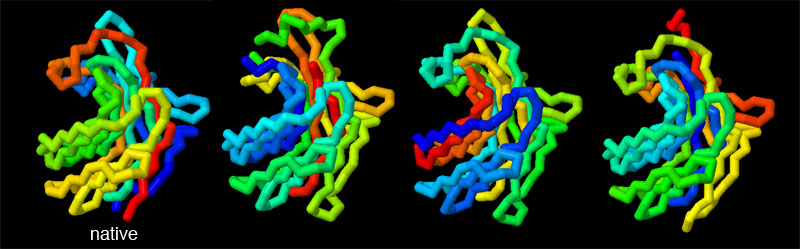 Esplorando la struttura La permutazione circolare è stata originariamente scoperta comparando la sequenza di amminoacidi della concanavalina A (file PDB 3cna) con la sequenza della lectina degli arachidi (peanut lectin) mostrate qui sotto (file PDB 2pel). Quando, dopo alcuni anni, la struttura di queste proteine è stata risolta con la tecnica della diffrazione dei raggi x, si è visto che le loro forme tridimensionali erano molto simili nonostante la permutazione della catena. Anche qui i tratti iniziale e finale delle catene sono mostrati rispettivamente in rosso e in blu. . . . . . . . . . . . . . . . . . . . . 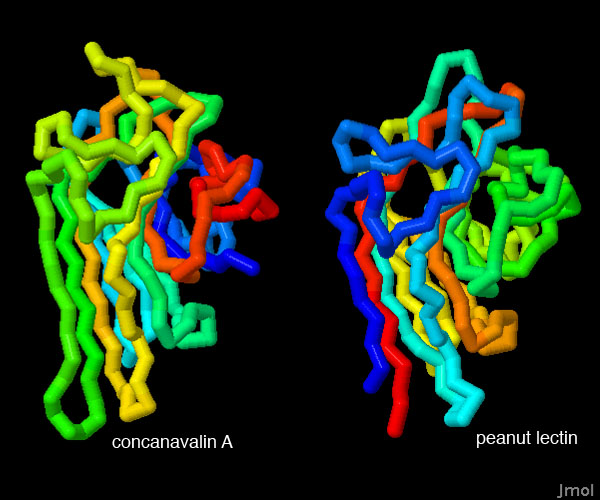
Spunti per ulteriori esplorazioni
|
||||||
|
|
||||||
