|
|
Proteina attivatrice di cataboliti |
|||||
|
Molecola del Mese di Dicembre 2003 Quando il glucosio scarseggia, la proteina attivatrice di cataboliti CAP mobilita le proteine necessarie per degradare altri nutrienti 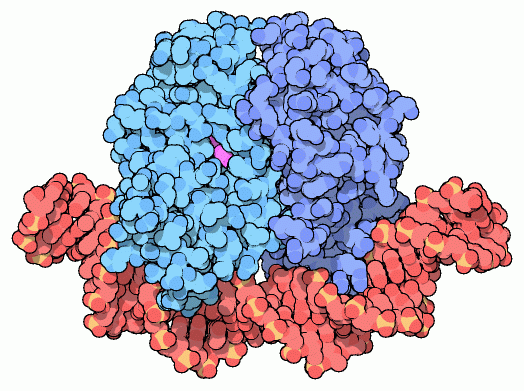 Golosi
di dolci Golosi
di dolci I batteri amano lo zucchero. In particolare, i batteri amano il glucosio che è facilmente digeribile e può essere rapidamente trasformato in energia chimica. Quando il glucosio è abbondante, i batteri ignorano gli altri alimenti che si trovano nel loro ambiente e si nutrono solo con il loro cibo favorito. Ma, quando il glucosio scarseggia, cambiano atteggiamento e mettono in moto gli strumenti necessari per utilizzare altre fonti di energia. 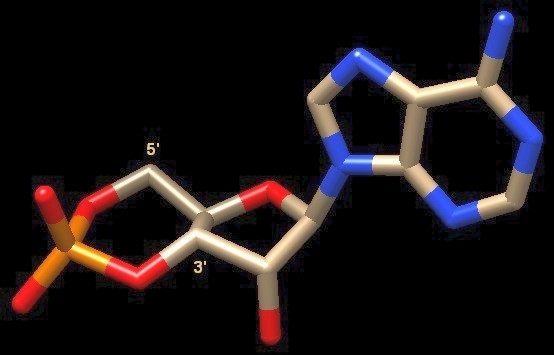 Messaggero
secondario Messaggero
secondario I batteri usano AMP ciclico, mostrato qui a lato (file PDB cAMP), per informare il loro apparato sintetico della concentrazione di glucosio disponibile in quel momento. Quando i livelli di glucosio scendono, viene attivata la adenil ciclasi, un enzima che si trova sulla superficie della cellula. Questo enzima afferra le molecole di ATP, taglia via due fosfati, e riconnette la parte terminale libera del fosfato rimasto in 5' con l'OH in posizione 3', creando un nuovo anello a 6 atomi, illustrato sulla sinistra dell'immagine qui a lato. La molecola che si forma è AMP ciclico (cAMP, adenosina monofostato ciclico). Quando viene rilasciato, cAMP diffonde attraverso la cellula stimolando la produzione degli enzimi che trasformano molecole di cibo diverse dal glucosio. A causa del suo ruolo nel trasportare messaggi dal sensore primario del glucosio (adenil ciclasi) all'apparato sintetico, AMP ciclico è noto come messaggero secondario. Leggere il messaggio La proteina attivatore di cataboliti (CAP), anche nota come proteina recettore dell'AMP ciclico (CRP), è attivata dall'AMP ciclico e stimola la sintesi degli enzimi che degradano molecole nutrienti diverse dal glucosio. È composta da due subunità identiche, mostrate in blu nell'immagine iniziale (file PDB 1cgp). Quando AMP ciclico (magenta) si lega, fa cambiare leggermente la conformazione della proteina, rendendola perfetta per legarsi al DNA (rosso). La proteina CAP si lega ad una specifica sequenza di DNA che si trova vicino ai geni che devono essere attivati. Come vedremo più sotto, quando CAP si lega al DNA, induce la RNA polimerasi a legarsi in quel luogo e a cominciare la trascrizione. Trovare un appiglio La proteina CAP è piuttosto rude quando si lega al DNA: lo afferra e lo piega di circa 90 gradi. Questa non è una piegatura leggera. Dato che il DNA è una doppia elica con due scanalature che scorrono a spirale lungo la catena, si piega più facilmente in alcune direzioni. La proteina CAP piega il DNA nei due punti dove le alfa eliche della proteina toccano la scanalatura principale del DNA che è comprimibile. 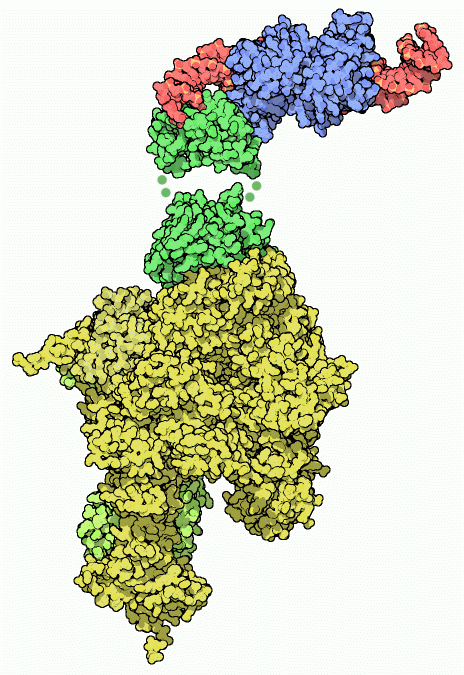 CAP
in azione CAP
in azione La proteina CAP attrae la RNA polimerasi (mdm 4-2003) sul DNA, stimolando la trascrizione dei geni che si trovano a valle di quel punto. Nella figura qui a fianco si vede la RNA polimerasi (gialla, file PDB 1iw7) che, all'estremità superiore, presenta due subunità (verdi) che interagiscono con CAP (blu). Queste subunità sono composte di due parti connesse da un braccio flessibile, mostrato con piccoli punti verdi. In questa struttura si può vedere solo metà di ognuna di queste subunità. Mentre il file PDB 1lb2 contiene le altre parti di queste subunità della RNA polimerasi, legate strettamente alla proteina CAP. Nella figura qui a lato, CAP è mostrata in alto (blu) ed è legata ad un piccolo pezzo di DNA (rosso). Naturalmente, nelle cellule questo DNA, fa parte di una lunga catena. Le braccia flessibili permettono alla RNA polimerasi di raggiungere le regioni vicine del DNA e iniziare la trascrizione. 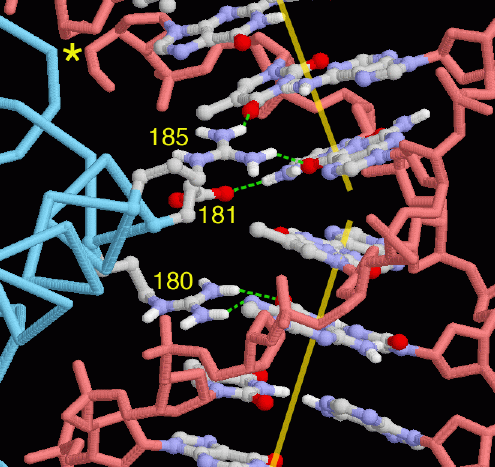 Esplorando
la struttura Esplorando
la struttura CAP riconosce una specifica sequenza di DNA, legandosi solamente a speciali siti vicini ai geni coinvolti nella produzione di energia. Il riconoscimento è dovuto a interazioni tra amminoacidi della proteina CAP e basi del DNA nella scanalatura principale. Nella figura qui a destra (file PDB 1cgp) si vedono due arginine (180 e 185) ed un acido glutammico (181) della proteina CAP che arrivano a sfiorare le basi del DNA formando specifici legami idrogeno (trattini verdi). Osservate, al centro, la grande curvatura nel DNA in corrispondenza del paio di basi timina-adenina . La proteina CAP interagisce anche con molti gruppi fosfato nella catena principale del DNA, rinforzando così il suo legame. Il DNA illustrato qui ha un'interruzione in un punto chiave della catena principale (asterisco). Se volete, potete vedere come CAP interagisce con questo sito esaminando un'altra struttura nel file PDB 1j59. A. Kolb, S. Busby, H. Buc, S. Garges and S. Adhya (1993): Transcriptional regulation by cAMP and its receptor protein. Annual Review of Biochemistry 62, pp. 749-795. S. Busby and R. H. Ebright (1999): Transcription activation by catabolite activator protein. Journal of Molecular Biology 293, pp. 199-213.
|
||||||
|
|
||||||
