|
Molecola del mese di settembre 2019
I nanodischi consentono di impaccare in modo efficiente un piccolo tratto
di membrana per studi sperimentali
 Introduzione Introduzione
I nanodischi e la microscopia crioelettronica stanno rivoluzionando
la biologia strutturale. Nuovi sviluppi nella microscopia crioelettronica
consentono ai ricercatori di determinare la struttura di macchine molecolari
molto più grandi e complesse che in passato. Tra queste, quelle
più sofisticate sono le proteine legate alla membrana che sono
di per sé difficili da studiare perché sono inserite in
un ambiente lipidico. In questo i nanodischi si sono rivelati utili.
Sono formati da un sottile disco di lipidi, grande solo quanto basta
per ospitare una singola proteina di membrana, tenuto insieme da una
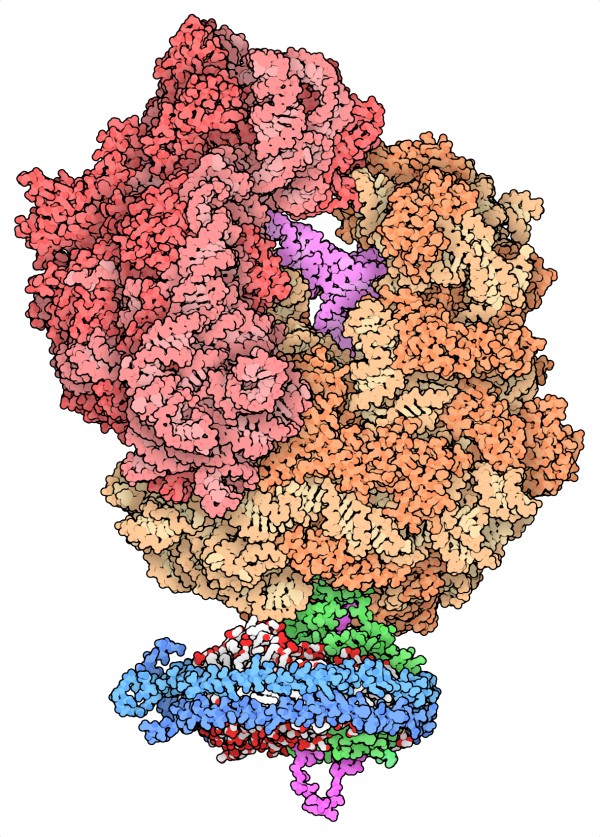
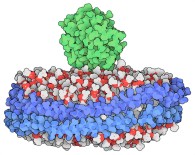
cintura proteica. Nella figura qui a lato si vede un ribosoma (rosso
e arancione) che sta sintetizzando una proteina (magenta, ancora legata
al tRNA in alto, magenta) che viene espulsa in basso attraverso il canale
formato dal complesso proteico di secrezione SecYE (verde) che è
inserito in un nanodisco di membrana (apolipoproteine in blu, fosfolipidi
in bianco e rosso).
Colesterolo buono
I nanodischi sono stati costruiti usando come modello strutture che
già esistono in natura.
Il colesterolo e altri lipidi vengono trasportati in tutto il corpo
in piccoli globuli circondati e tenuti in forma da speciali proteine
chiamate apolipoproteine. Una classe particolare di questi globuli
è chiamata HDL (high-density lipoproteins), lipoproteine ad alta
densità, dato che contengono una grande percentuale di proteine
e quindi sono più dense di altre lipoproteine più ricche
di lipidi come le LDL low-density lipoproteins.
Le HDL sono spesso chiamate colesterolo buono perché hanno il
compito di trasportare l'eccesso di colesterolo dalla periferia del
nostro corpo al fegato dove viene rimosso dal sangue.
Le HDL si sono rivelate buone anche per la scienza perché una
di loro ha una forma a disco che è stata di ispirazione per la
progettazione dei nanodischi.
Nanodischi ovunque
Cercando strutture di nanodischi negli archivi PDB se ne possono trovare
decine, ma sono pochissime quelle che contengono le coordinate dei lipidi
del nanodisco. Questo perché i lipidi sono molto flessibili e
i ricercatori sono più interessati alla proteina che è
inserita nel nanodisco e così i lipidi circostanti restano sfuocati
e non si vedono nella mappa della microscopia crioelettronica. La struttura
enorme mostrata qui sopra (file PDB 4v6m)
è un'eccezione, i ricercatori hanno usato la microscopia crioelettronica
combinata con simulazioni di dinamica molecolare per generare un modello
strutturale dell'intero complesso, compreso il nanodisco e i lipidi
che lo costituiscono. Contiene un ribosoma che sta sintetizzando una
proteina e la sta facendo uscire dalla membrana attraverso il canale
formato dalla proteina esportatrice SecYE.
 Nanodischi
ed NMR Nanodischi
ed NMR
Dato che i nanodischi sono piccoli e solubili in acqua, sono stati una
manna per i biologi strutturali che usano la tecnica NMR per studiare
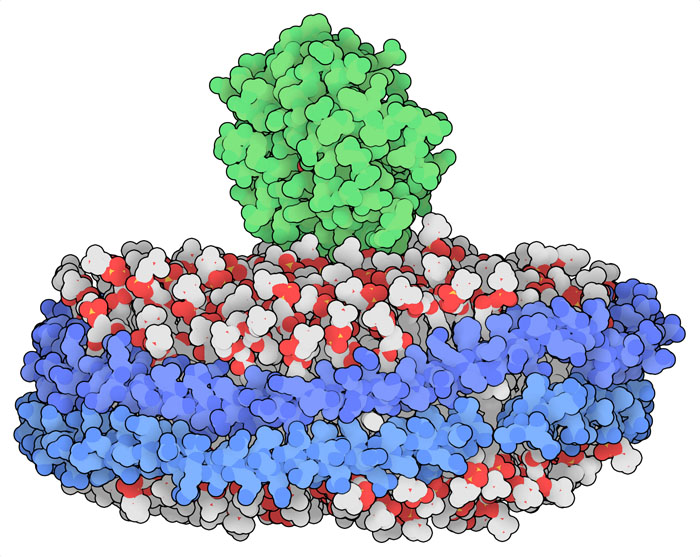
le proteine. La struttura mostrata qui a lato (file PDB 6clz)
mostra il dominio legato alla membrana della proteina MT1-MMP (metallo
tipo 1, metalloproteinasi di matrice), un enzima che taglia il collagene
ed è necessario per consentire alle cellule di migrare attraverso
la matrice extracellulare quando vengono costruiti nuovi vasi sanguigni.
Questa proteina è anche un bersaglio per la chemioterapia del
cancro perché le cellule tumorali che danno metastasi spesso
usano questo enzima per diffondersi nel corpo. Questa struttura ha permesso
ai ricercatori di esplorare due modi di legarsi del dominio e il loro
ruolo nel regolare l'azione dell'enzima.
 Esplorando
la struttura Esplorando
la struttura
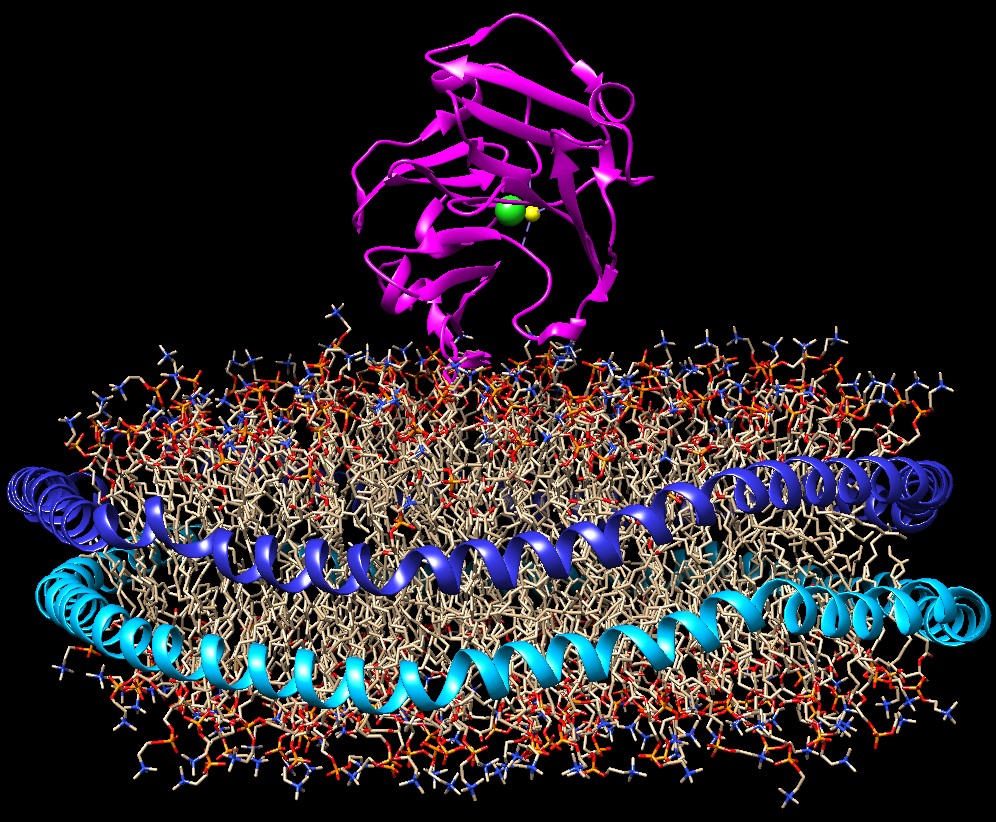
I nanodischi vengono realizzati con tecniche di ingegneria genetica
a partire da apolipoproteine naturali, cambiando la lunghezza delle
catene per circondare un disco delle dimensioni desiderate.
Le proteine ingegnerizzate sono chiamate MSP (membrane scaffolding proteins)
proteine che supportano una membrana e, come le apolipoproteine, hanno
una struttura ad alfa elica che dirige verso l'interno dell'anello una
serie di amminoacidi apolari evidenziati nella figura qui a fianco
con sferette (file PDB 6clz). Questi amminoacidi
stabilizzano le code idrofobiche dei fosfolipidi di membrana.
Nell'immagine qui sotto, realizzata con Chimera, si possono apprezzare
meglio le strutture tridimensionali delle catene proteiche e dei fosfolipidi
di questa struttura (file PDB 6clz).
 E'
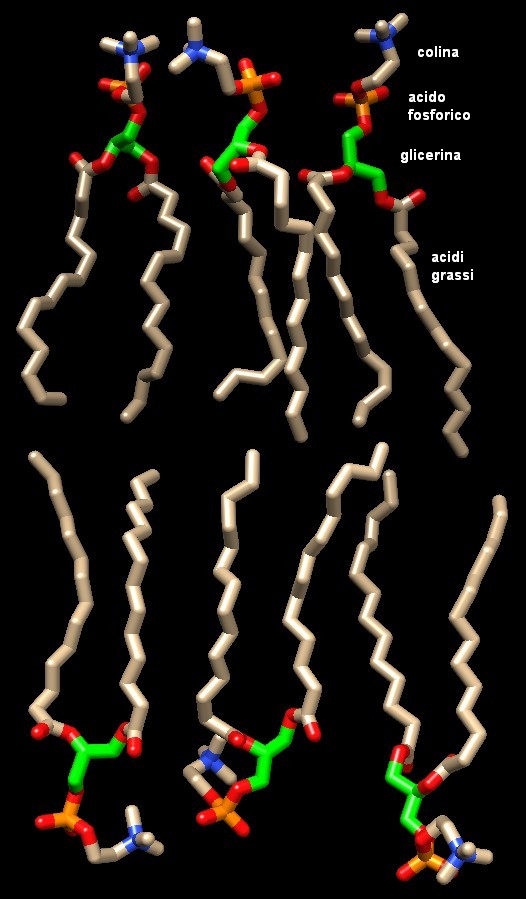
interessante anche osservare nel dettaglio la struttura dei fosfolipidi
che costituiscono il doppio strato lipidico della membrana. Qui a fianco
sono mostrati sei fosfolipidi presi dalla struttura complessa del file
PDB 6clz esaminato sopra. E'
interessante anche osservare nel dettaglio la struttura dei fosfolipidi
che costituiscono il doppio strato lipidico della membrana. Qui a fianco
sono mostrati sei fosfolipidi presi dalla struttura complessa del file
PDB 6clz esaminato sopra.
Si tratta di sei lecitine, molecole composite che hanno al centro
una molecola di glicerina (con i tre carboni evidenziati in verde, ognuno
dei quali regge un ossigeno rosso).
La glicerina lega, sugli ossigeni del primo e del secondo carbonio,
due acidi grassi con le catene idrocarburiche affiancate tra loro. Sull'ossigeno
del terzo carbonio è legata una molecola di acido fosforico (arancione
e rossa) a sua volta legata ad una colina (ossigeno rosso e azoto blu)
in cui l'azoto è legato a tre metili e così diventa uno
ione ammonio positivo.
La parte polare dei fosfolipidi è costituita dagli ossigeni dell'acido
fosforico (negativi) e dall'azoto dello ione ammonio (positivo). Questa
porzione polare è rivolta all'esterno verso l'ambiente acquoso
polare.
La porzione apolare dei fosfolipidi è formata dalle code degli
acidi grassi che si affiancano una all'altra formando una superficie
apolare che aderisce alla superficie sottostante apolare del secondo
strato di fosfolipidi.
Spunti per ulteriori esplorazioni
Cercate "nanodisc" al EMDataResource per esaminare
la mappe EM di queste proteine. Vedrete che il nanodisco è spesso
non ben risolto.
Cercate "apolipoprotein" nel sito RCSB PDB per vedere la struttura
di queste proteine quando non sono legate ai lipidi.
 Bibliografia Bibliografia
6clz: Marcink, T.C., Simoncic, J.A., An, B., Knapinska,
A.M., Fulcher, Y.G., Akkaladevi, N., Fields, G.B., Van Doren, S.R. (2019)
MT1-MMP Binds Membranes by Opposite Tips of Its beta Propeller to Position
It for Pericellular Proteolysis. Structure 27: 281-292.e6.
Denisov, I.G., Sligar, S.G. (2016) Nanodiscs for structural and
functional studies of membrane proteins. Nature Structural and Molecular
Biology 6, 481-486.
4v6m: Frauenfeld, J., Gumbart, J., Sluis, E.O., Funes, S., Gartmann,
M., Beatrix, B., Mielke, T., Berninghausen, O., Becker, T., Schulten,
K., Beckmann, R. (2011) Cryo-EM structure of the ribosome-SecYE complex
in the membrane environment. Nature Structural and Molecular Biology
18: 614-621.
|
|
|