|
|
Sirtuine |
|||||
|
Molecola del Mese di Settembre 2017 L'attivazione della sirtuina sembra essere collegata ad un più lento invecchiamento 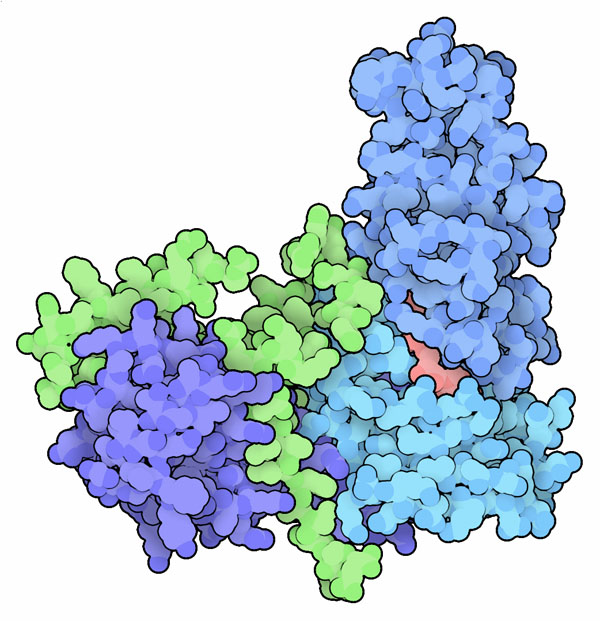 Introduzione IntroduzioneMentre studiavano una piccola proteina che regolava l'espressione dei geni, chiamata sir2, i ricercatori hanno fatto una scoperta sorprendente. Le cellule di lievito ingegnerizzate per produrre una maggior quantità di sir2 vivevano il 30% in più del normale. Naturalmente si è acceso subito un grande interesse su questa proteina per scoprire come agisce e come possa influenzare l'invecchiamento del lievito e magari anche quello delle nostre cellule. I ricercatori hanno scoperto che sir2 è un enzima che rimuove i gruppi acetilici dagli istoni. Gli istoni sono proteine basiche, ricche di lisine e arginine con carica positiva, attorno alle quali si avvolge il DNA, ricco di gruppi fosfato negativi, per assumere una struttura più compatta, detta cromatina, che serve anche a silenziare i geni. Gli istoni, con le lisine deacetilate da sir2, potevano formare delle regioni più compatte di cromatina che silenziavano i geni in modo più efficiente. Sir2 (silent information regulator 2) appartiene ad una classe di proteine che si trovano in tutti gli organismi eucarioti e sono state chiamate sirtuine. Nell'immagine qui a lato si vede sir2 (in vari toni del blu) legata al NAD (rosso) e a un dominio di sir4 (verde). Una famiglia di sirtuine Le nostre cellule costruiscono sette diverse sirtuine (SIRT1 - SIRT7) che realizzano una varietà di funzioni. Alcune svolgono la normale funzione di modificare gli istoni e di regolare la trascrizione dei geni. Altre si legano a proteine nel citoplasma e nei mitocondri e sono implicate nel regolare una grande varietà di processi dal metabolismo alla neurodegenerazione. Alcune sono specializzate nel rimuovere i gruppi acetilici, mentre altre rimuovono gruppi diversi come lipidi o fanno altre modifiche. Lo studio intensivo delle sirtuine ha rivelato che in molti organismi sono legate all'invecchiamento. La struttura delle sirtuine Le sirtuine usano un cofattore molto comune, il NAD, per realizzare una reazione insolita. Prendono il NAD, un cofattore impiegato nelle reazioni di ossidoriduzione, e rimuovono il suo anello caratteristico di nicotinammide. Poi estraggono il gruppo acetilico da una lisina acetilata della proteina bersaglio e lo trasferiscono al frammento rimasto della molecola del NAD. Tutte le sirtuine, come sir2 mostrata qui sopra (file PDB 4iao), hanno una caratteristica strutturale in comune. Hanno due domini, uno può legare il NAD e gli toglie l'anello di nicotinammide, l'altro posiziona la lisina acetilata vicino al NAD e catalizza il trasferimento del gruppo acetile. Alcune sirtuine hanno anche altri domini che interagiscono con proteine regolatorie come per esempio sir4 mostrata in verde nella figura. 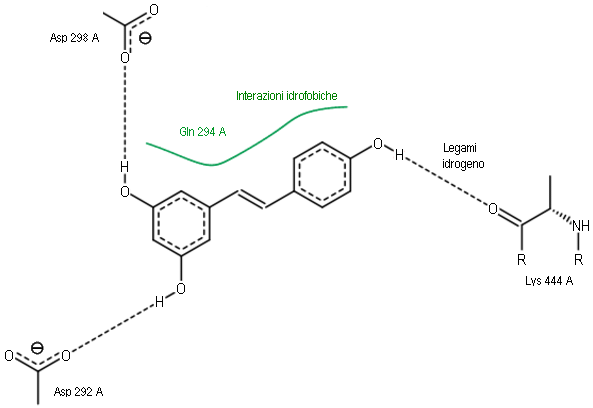 Molecole
anti-invecchiamento Molecole
anti-invecchiamentoLa scoperta di un legame tra sirtuine ed invecchiamento ha dato il via ad una intensa ricerca di composti che possano aumentare l'attività delle sirtuine. Il resveratrolo, un polifenolo naturale presente in alcuni frutti, ma soprattutto nella buccia dell'uva e quindi nel vino rosso, è stato uno dei primi ad essere individuato ed è stato (con troppo entusiasmo) propagandato dai media. La struttura della SIRT1 che contiene resveratrolo (file PDB 5btr) mostra che questo agisce in modo allosterico cioè legandosi in un altro sito della sirtuina e aiuta la proteina ad avvicinare i due siti attivi potenziando l'azione enzimatica. Nella figura qui a lato si vede come il resveratrolo si lega all'interno dell'enzima: i suoi tre gruppi OH formano legami idrogeno con tre amminoacidi mentre gli anelli aromatici interagiscono con un altro amminoacido. 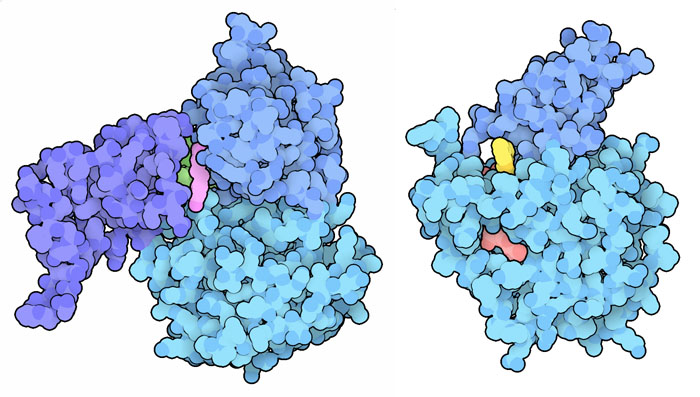 Oltre al resveratrolo, sono stati testati migliaia di altri composti e si è scoperto, per esempio, che un composto che contiene un pirrolo e una piridina (UBCS) è in grado di attivare la SIRT6 (file PDB 5mf6) con un meccanismo allosterico simile a quello del resveratrolo. Nelle immagini qui a lato si vede, sulla sinistra, SIRT1 legato al resveratrolo (magenta), mentre sulla destra, si vede SIRT6 legato all'attivatore allosterico UBCS (giallo). Nella stessa struttura si vede anche un analogo del NAD (rosso). Esplorando la struttura Le due strutture qui sotto mostrano una sirtuina batterica prima e dopo la sua reazione su una proteina bersaglio (verde). Nell'immagine sulla sinistra (file PDB 2h4f) si vede il NAD (arancione con l'anello di nicotinammide evidenziato in magenta) legato nel sito attivo insieme ad un piccolo peptide (verde) del soppressore di tumore P53 (mdm 7-2002). Notate che la lisina acetilata (con il gruppo acetile in bianco e rosso) è trattenuta molto vicino all'anello della nicotinammide (magenta). Nell'immagine sulla destra (file PDB 2h59) si vede la sirtuina alla fine della reazione. Il gruppo acetile (bianco e rosso) è stato trasferito sul NAD nel punto dove prima era legata la nicotinammide. 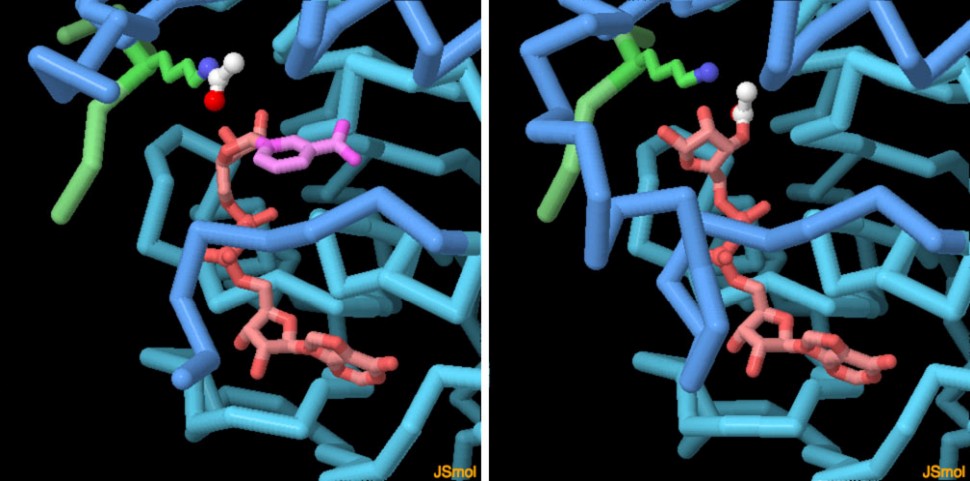
Spunti per ulteriori esplorazioni Fino ad oggi sono state determinate molte strutture di sirtuine umane: trovatele nel PDB cercando “sirtuin” e poi limitando la ricerca a “Homo sapiens” usando l'opzione che si trova nella parte sinistra del menù. Le sirtuine sono flessibili, infatti si aprono e chiudono durante la loro reazione. Usate lo strumento di confronto delle strutture per esplorare questa flessibilità, per esempio con le due forme di SIRT2 1j8f e 3zgv. 5mf6: You W, Rotili D, Li TM, Kambach C, Meleshin M, Schutkowshi M, Chua KF, Mai A & Steegborn C (2017) Structural basis of Sirtuin 6 activation by synthetic small molecules. Angewandte Chemie 56, 1007-1011. Bonkowski MS, Sinclair DA (2016) Slowing ageing by design: the rise of NAD+ and sirtuin-activating compounds. Nature Reviews Molecular Cell Biology 17, 679-690. Sacconnay L, Carrupt PA & Nurisso A (2016) Human sirtuins: structures and flexibility. Journal of Structural Biology 196, 534-542. 5btr: Cao D, Wang M, Qiu X, Liu D, Jiang H, Yang N & Xu RM (2015) Structural basis for allosteric, substrate-dependent stimulation of SIRT1 activity by resveratrol. Genes Development 29, 1316-1325. 4iao: Hsu HC, Wang CL, Wang M, Yang N, Chen Z, Sternglanz R, Xu RM (2013) Structural basis for allosteric stimulation of Sir2 activity by Sir4 binding. Genes Development 27, 64-73. 2h4f, 2h59: Hoff KG, Avalos JL, Sens K & Wolberger C (2006) Insights in the sirtuin mechanism from ternary complexes containing NAD(+) and acetylated peptide. Structure 14, 1231-1240.
|
||||||
|
|
||||||
