|
|
Selenocisteina sintasi |
|||||
|
Molecola del Mese di Agosto 2008 Talvolta è necessario usare il selenio al posto dello zolfo per svolgere alcuni compiti speciali 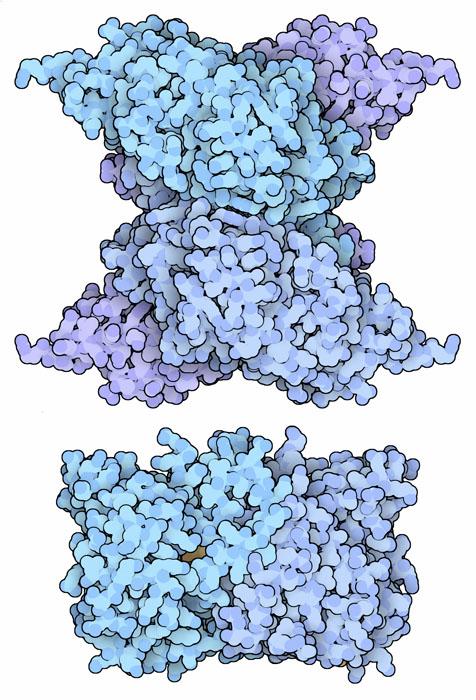 Introduzione IntroduzioneTra i vari oligoelementi contenuti in minima quantità negli integratori alimentari o nei complessi multivitaminici, spesso è presente anche il selenio. Questo, però, è un elemento che va assunto con grande cautela. In generale, infatti, i composti del selenio sono tossici e hanno uno sgradevole odore di aglio, d'altra parte, quantità minime di selenio sono essenziali per la nostra salute. Gli atomi di selenio sono simili a quelli di zolfo, si trovano uno sotto l'altro nella Tavola Periodica degli Elementi e quindi hanno proprietà simili. I composti del selenio, però, sono in generale più reattivi. Alcune proteine specializzate, che contengono selenio, hanno bisogno proprio di questa reattività extra per svolgere il loro compito. Per esempio, se è presente il selenio al posto dello zolfo, l'enzima tioredoxina reduttasi aumenta di 100 volte la sua velocità di catalisi e l'enzima formiato deidrogenasi diventa 300 volte più veloce. Selenometionina e selenocisteina Due dei venti amminoacidi che compongono le proteine contengono atomi di zolfo, metionina e cisteina, entrambi possono essere sintetizzati anche nella versione con il selenio al posto dello zolfo. La selenometionina non viene sintetizzata intenzionalmente nelle cellule, ma viene creata di tanto in tanto accidentalmente dai normali enzimi che sintetizzano la metionina. Dato che la sua reattività è abbastanza simile a quella della normale metionina, la sua eventuale presenza non costituisce un problema per la proteina. La selenometionina, d'altra parte, è molto utile ai chimici cristallografi che devono determinare la struttura tridimensionale delle proteine, infatti il selenio è più grande e ricco di elettroni dello zolfo ed è più facile da localizzare negli esperimenti di diffrazione ai raggi X. La selenocisteina, invece, è più reattiva della normale cisteina e viene introdotta in speciali selenoproteine quando è necessario. Il ventunesimo codone E' stata una sorpresa scoprire che le cellule hanno modificato il loro codice genetico per introdurre la selenocisteina nelle proteine. Il codice genetico comune a tutti gli organismi della Terra specifica venti amminoacidi oltre ad alcuni segnali di inizio e stop. Per poter aggiungere il ventunesimo amminoacido, selenocisteina, a questo codice, le cellule leggono in modo alternativo il codone UGA che normalmente significa stop. Questo però pone un problema: come fa la cellula a sapere quando leggere UGA come stop e quando deve leggerlo come selenocisteina? Questo viene realizzato con una speciale sequenza di basi azotate che è posta dopo il codone UGA. Nei batteri questo segnale segue immediatamente il codone UGA, ed è quindi all'interno della porzione codificante dell'RNA messaggero. Nelle nostre cellule, invece, il segnale si trova molto più avanti, alla fine della porzione codificante. Come si può vedere qui sotto, questa sequenza viene riconosciuta da uno speciale fattore di allungamento (mdm 09-2006) che trasporta il tRNA che lega la selenocisteina verso il ribosoma dove avviene la sintesi proteica. Il selenio può essere tossico Dato che selenio e zolfo sono molto simili, le cellule devono assicurarsi che la selenocisteina non si leghi al normale tRNA della cisteina altrimenti verrebbe introdotta accidentalmente nel punto sbagliato durante la sintesi proteica. Per risolvere questo problema, il complesso selenocisteina tRNA viene costruito in tre stadi. Nel primo stadio, un amminoacido serina (che ha un gruppo OH al posto del gruppo SH della cisteina) viene legato al tRNA della selenocisteina. Nel secondo stadio il gruppo OH della serina viene fosforilato da un enzima e viene così reso più reattivo. Nel terzo stadio, l'ossigeno fosforilato della serina viene sostituito col selenio dall'enzima selenocisteina sintasi mostrato qui sopra in alto dal file PDB 3bc8. Il piccolo enzima selenofosfato sintasi, mostrato qui sopra in basso dal file PDB 2yye, crea una forma attivata di selenio che è necessaria per questa reazione. Con questo processo a stadi, non è necessario che un enzima sappia svolgere il difficile compito di distinguere la selenocisteina dalla cisteina per legarla al corrispondente tRNA. 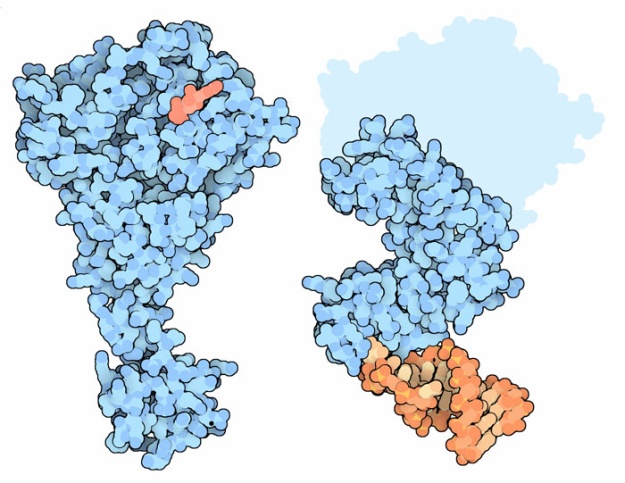 Codificare
per il selenio Codificare
per il selenioQui a fianco sulla sinistra è mostrato il fattore di allungamento SelB (file PDB 1wb1), che decide quando bisogna introdurre una selenocisteina in corrispondenza di un codone UGA invece di arrestare la sintesi proteica. SelB possiede, in basso, una coda flessibile in grado di riconosce una speciale "sequenza di inserimento della selenocisteina" nell'RNA messaggero che si trova oltre il codone UGA e forma un caratteristico tratto a forcina. La grande porzione in alto di SilB lega GTP (arancione) e trasporta il tRNA con la selenocisteina al ribosoma dove avviene la sintesi proteica. SelB è simile al fattore di allungamento Tu (mdm 9-2006) che trasporta al ribosoma i normali tRNA legati al loro amminoacido. La struttura qui a fianco sulla destra (file PDB 2uwm) mostra il legame tra la coda flessibile di SelB e la sequenza a forcina dell'RNA messaggero (in arancione e beige). La proteina SelB riconosce una particolare base azotata, guanina, che sporge dalla catena e riconosce inoltre la forma complessiva del tratto a forcina. Esplorando la struttura Si sono trovate finora 15 diverse selenoproteine nelle cellule umane. Queste includono alcuni enzimi deiodinasi che sono essenziali per la produzione degli ormoni tiroidei, alcuni enzimi glutatione perossidasi (glutatione mdm 8-2017) importanti per la conversione e la eliminazione di composti tossici che possiedono atomi di ossigeno molto reattivi e infine alcune altre proteine dalla funzione non ancora nota. Qui sotto è rappresentato il sito attivo di una delle quattro catene tutte uguali che compongono l'enzima glutatione perossidasi (file PDB 1gp1). Ognuno dei quattro siti attivi dell'enzima contiene una selenocisteina (con il selenio arancione) che è attivata da due amminoacidi, un triptofano e una glutammina (Trp e Gln) che la sfiorano con i loro atomi di azoto (blu). . . . . . . . . . . . 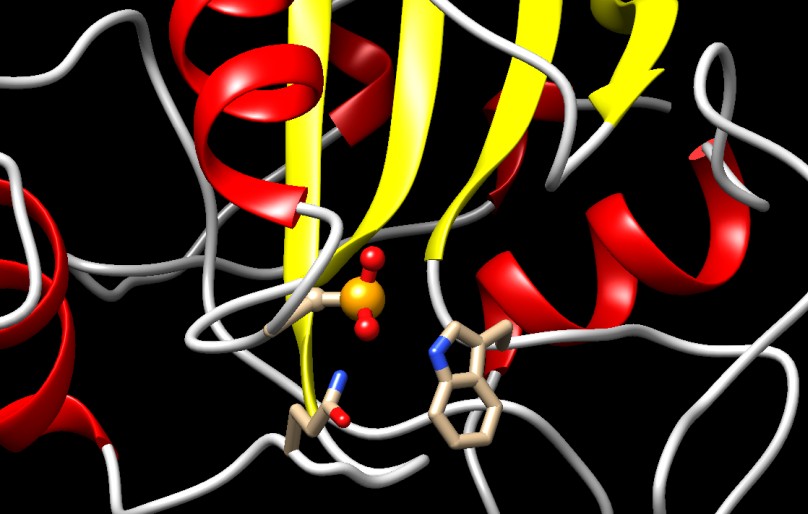 In questa struttura, la selenocisteina si trova in uno stato molto ossidato infatti il selenio è legato a due atomi di ossigeno (rossi). Nel normale ciclo catalitico dell'enzima, si pensa che il selenio estragga l'atomo di ossigeno reattivo dai perossidi, molecole tossiche e pericolose, e poi usi l'acqua e il glutatione per renderli innocui. Spunti per ulteriori esplorazioni Negli archivi PDB ci sono molte selenoproteine naturali e anche molte proteine che contengono selenometionina che è servita per determinare meglio la struttura cristallografica. Cercane qualche esempio con una ricerca alla pagina http://www.rcsb.org/ SelB riconosce la speciale sequenza di inserimento
della selenocisteina formando una particolare interazione con una base
azotata di guanina del tratto a forcina del mRNA. Identifica gli amminoacidi
coinvolti in questo riconoscimento osservando la struttura PDB 2uwm
con Chimera (vedi Chimica
al Computer)
|
||||||
|
|
||||||
