|
|
Autosplicing mRNA |
|||||
|
Molecola del mese di maggio 2005 Speciali sequenze nell'RNA messaggero sono in grado di fare splicing, cioè di eliminare un segmento e ricongiungersi, senza l'aiuto di enzimi 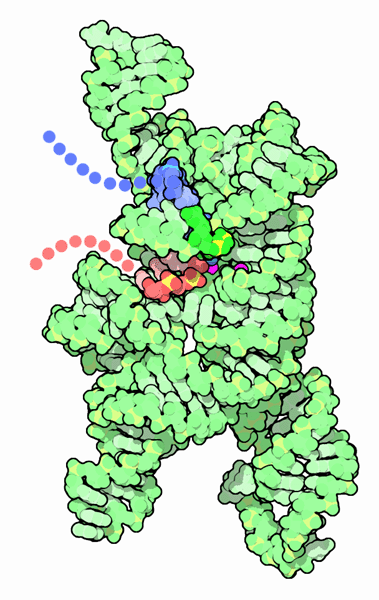 Introduzione IntroduzioneLa Natura è piena di sorprese e quando si crede di aver capito qualcosa, si trova spesso un'eccezione. Vent'anni fa questo è successo con gli enzimi. I biochimici pensavano, dopo decadi di lavoro, che le proteine fossero le uniche molecole capaci di catalizzare le reazioni chimiche nella cellula. Quindi è stata una sorpresa quando Thomas Cech ed i suoi collaboratori hanno scoperto una reazione spontanea di splicing dell'mRNA che avveniva anche quando tutte le proteine erano state rimosse. Da allora i ricercatori hanno scoperto molti altri esempi di ribozimi, molecole di RNA in grado di catalizzare le reazioni chimiche. Eliminare gli introni Nelle piante e negli animali, la maggior parte delle molecole di mRNA viene sintetizzata sotto forma di lunghi precursori, pre-mRNA, che devono subire il processo di splicing per creare la molecola finale di mRNA maturo. Nello splicing, i lunghi pre-mRNA vengono tagliati in frammenti, alcuni di questi frammenti vengono eliminati, mentre gli altri sono ricuciti insieme. I precursori dell'mRNA sono composti di esoni separati da introni. Gli esoni sono le parti importanti che vengono riassemblate, mentre gli introni sono i segmenti che devono essere rimossi. Nella maggior parte dei casi, il pre-mRNA viene tagliato e poi ricucito da uno spliceosoma (mdm 5-2020), una macchina molecolare composta da proteine e da RNA. In alcuni casi, però, il pre-mRNA può compiere da solo le reazioni di splicing. Il primo esempio, scoperto da Thomas Cech era un pre-mRNA ribosomiale di un protozoo. Da allora sono stati identificati centinaia di esempi simili nel genoma di molti organismi. L'esempio mostrato qui a fianco (file PDB 1u6b) è parte di un RNA transfer batterico che deve subire il processo di splicing prima di poter assumere la sua forma funzionale. Nella figura, la grande struttura verde è l'introne che usa un GTP e due ioni magnesio per rimuovere sè stesso dalla catena originale. I due esoni che saranno ricuciti insieme sono colorati in rosso e blu. Solo un piccolo pezzo di ogni esone è incluso nella struttura. Ribozimi ovunque Quando gli scienziati hanno cominciato a cercarli, hanno scoperto ribozimi ovunque. In alcuni casi i ribozimi si trovano all'interno delle stesse molecole di RNA che devono reagire e quindi agiscono su sè stessi, come gli introni che fanno la reazione di autosplicing. In altri casi, i ribozimi agiscono su altre molecole di RNA, rompendo e ricostruendo i legami fosfato che tengono insieme le catene. In alcuni ribozimi, come nello spliceosoma e nella ribonucleasi P (che agiscono sull'RNA tranfer e su altre molecole di RNA), l'RNA lavora insieme con proteine per compiere la reazione. Anche se la maggior parte dei ribozimi compie reazioni di taglio e di ricucitura dell'RNA, il ribosoma (che è un ribozima) mostra che l'RNA può compiere anche altre reazioni, come la formazione del legame peptidico nelle proteine. 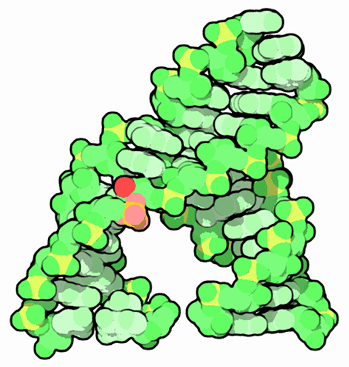 Il
più piccolo ribozima Il
più piccolo ribozima Il ribozima a testa di martello mostrato qui a fianco (così chiamato per la sua forma) è il più piccolo ribozima naturale scoperto finora (file PDB 1mme). Realizza una reazione molto semplice: il gruppo ossidrile (rosso) attacca il fosfato lì vicino (rosa) rompendo la catena di RNA. La strana struttura assunta dalla catena di RNA intorno a questo punto mantiene il gruppo ossidrile nella posizione ideale per realizzare il taglio. Questi tipi di ribozimi "nucleolitici" sono usati per risolvere un problema particolare incontrato dagli organismi che hanno un DNA circolare. Quando la RNA polimerasi sintetizza RNA a partire da DNA circolare, scorre sopra questo compiendo più giri e quindi sintetizzando molte copie del DNA sotto forma di un filamento lungo e continuo di RNA. I siti di auto-scissione dei ribozimi, che si trovano inseriti nell'RNA, rompono poi la lunga catena in frammenti funzionali più piccoli. 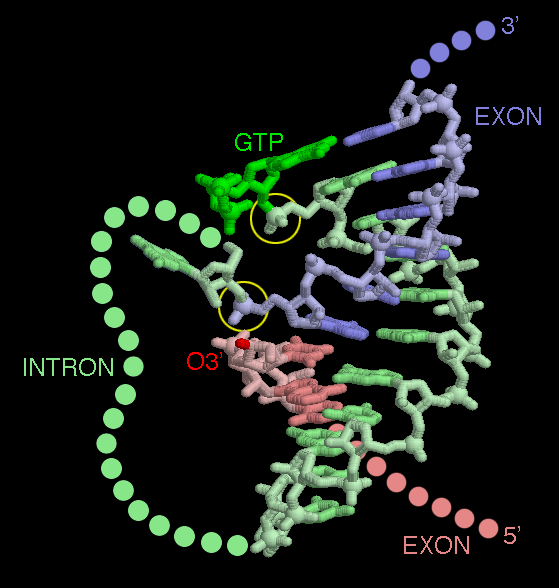 Esplorando
la struttura Esplorando
la struttura L'autosplicing RNA illustrato qui a fianco (file PDB 1u6b) è stato colto nel mezzo della reazione di splicing. Nella figura, i due esoni sono mostrati in rosso e in blu, e parte dell'introne è mostrata in verde. Il resto dell'introne è stato omesso, mentre invece era visibile nella prima pagina di questa Molecola del mese. All'inizio della reazione il filamento di RNA era continuo. L'ossigeno 3' terminale dell'esone (rosso) era legato al gruppo fosfato evidenziato con un cerchio, quello più in alto dei due. La catena dell'RNA si snodava dal terminale 5' dell'esone rosso, attraverso l'introne verde (cominciando dal lato più in alto) e fino all'esone blu dove si trova il terminale 3'. La struttura mostra la molecola dopo che il primo taglio è stato fatto. Il GTP ha interrotto la catena tra l'introne e l'esone rosso, lasciando il GTP legato ad una estremità dell'introne. A questo punto, l'atomo di ossigeno 3' terminale dell'esone rosso si trova nella posizione ideale per attaccare il fosfato alla fine dell'esone blu (quello cerchiato più in basso). Quando quel legame sarà fatto, l'introne verde sarà rilasciato, e gli esoni rosso e blu saranno ricuciti insieme. La porzione più corta dell'introne mostrata qui è chiamata "sequenza guida" perchè fa allineare perfettamente i due esoni uno vicino all'altro. T. R. Cech (2002) Ribozymes, the first 20 years. Biochemical Society Transactions 30, 1162-1166. J. A. Doudna and T. R. Cech (2002) The chemical repertoire of natural ribozymes. Nature 418, 222-228. D. M. J. Lilley (2003) The origins of RNA catalysis in ribozymes. Trends in Biochemical Sciences 28, 495-501.
|
||||||
|
|
||||||
